聚类算法与实现
“物以类聚,人以群分”,所谓的聚类,就是将样本划分为由类似的对象组成的多个类的过程。聚类后,我们可以更加准确的在每个类中单独使用统计模型进行估计、分析或预测;
也可以探究不同类之间的相关性和主要差异。
聚类和上一讲分类的区别:分类是已知类别的,聚类未知。
对于K-means算法与层次聚类算法,课本《统计分析与SPSS的应用》有更详细地介绍,本文不做过多的详细叙述
一、K-means++算法
1.算法的提出与介绍
K-means算法我们已经非常熟悉,通过迭代聚类中心,以各个点离聚类中心的距离为依据进行聚类。但是其也有缺点(1)对初值敏感。(2)对于孤立点数据敏感。
对初值敏感体现在选择不同的初值会影响聚类结果
对于孤立点数据敏感体现在会因异常值而影响聚类结果
但是我们的K-means++算法改进了这两个缺点

2.SPSS的实现
SPSS默认使用的就是K-means++算法

3.关于K-means算法的一些讨论

二、系统聚类
系统聚类相比K-means来说,可以让我们更好地来确定到底聚几类。
1.算法思想简述
一开始我们有n个个案,也就是n个类。现在我们要计算这n个类两两之间的欧氏距离(也可以是其他的距离,此处就用欧氏距离),找到欧式距离最小的两个类,把这两个类分为一类,此刻我们就得到了n-1个类。然后再次进行这个步骤,得到n-2个类,n-3,n-4,.......,1个类。最后由我们来判断应该到底聚几类
2.SPSS实现
按照图片上的设置好就行,其中冰柱图可以不勾选
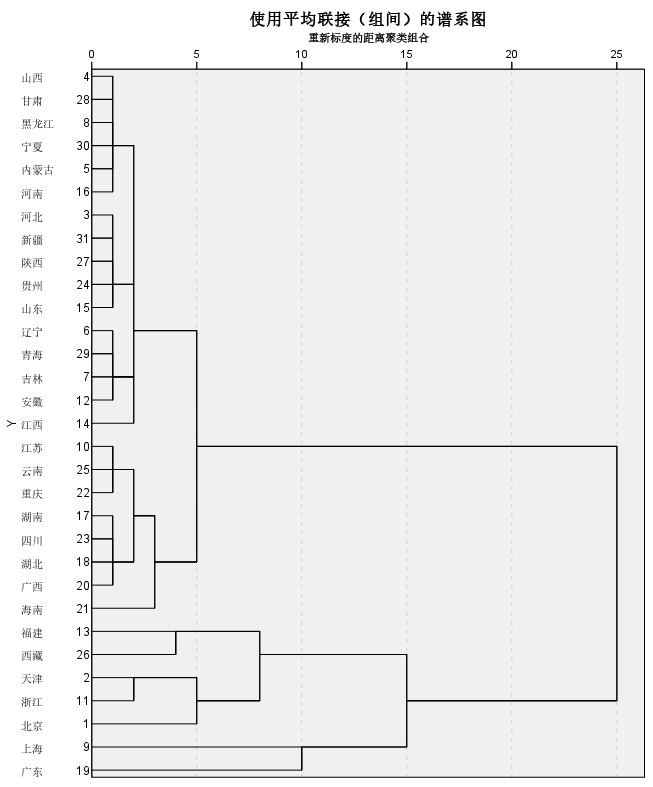
3.聚类谱系图
通过谱系图我们可以清楚地看到聚类过程
4.聚类碎石图
(1)制作碎石图

在SPSS的集中计划中,系数这一列代表着分类过程的欧氏距离。阶段一就代表着聚了n-1类,阶段二就代表着聚了n-2类。这样我们可以在EXCEL中作出以类别为纵坐标,距离为横坐标的碎石图
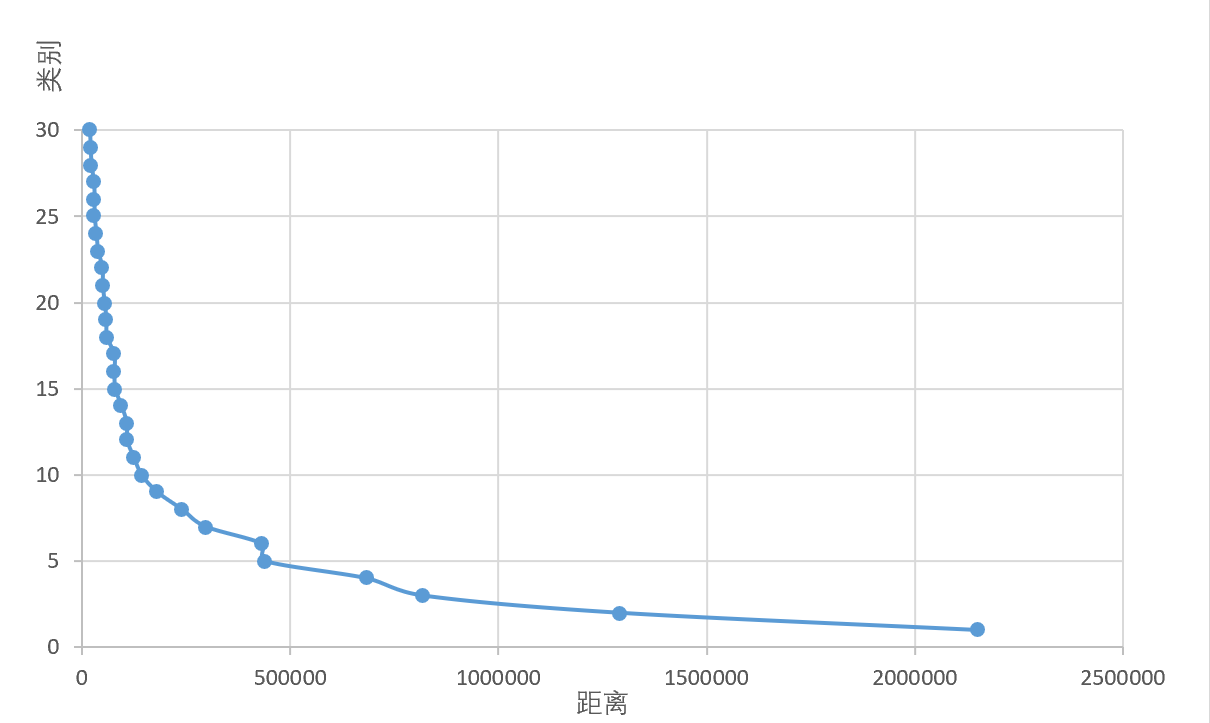
(2)确定聚类数的依据
到5类后,类间距离迅速增大。根据类间距离小形成类的相似性大,类间距离大形成类的相似性小的原则,可以找到类间距离急剧增大的拐点,以它作为确定分类数目的参考。在本例中,可以考虑聚成5类及以下
5.确定K后保存聚类结果并画图
(1)保存结果
在确定到底聚几类后,我们可以将聚类结果保存在我们的数据集里
(2)制作散点图与优化图形
只要当指标个数为2或者3的时候才能画图。利用SPSS的图表构建器,此处我们以两个指标为例进行演示。选择分类结果作为颜色分类依据
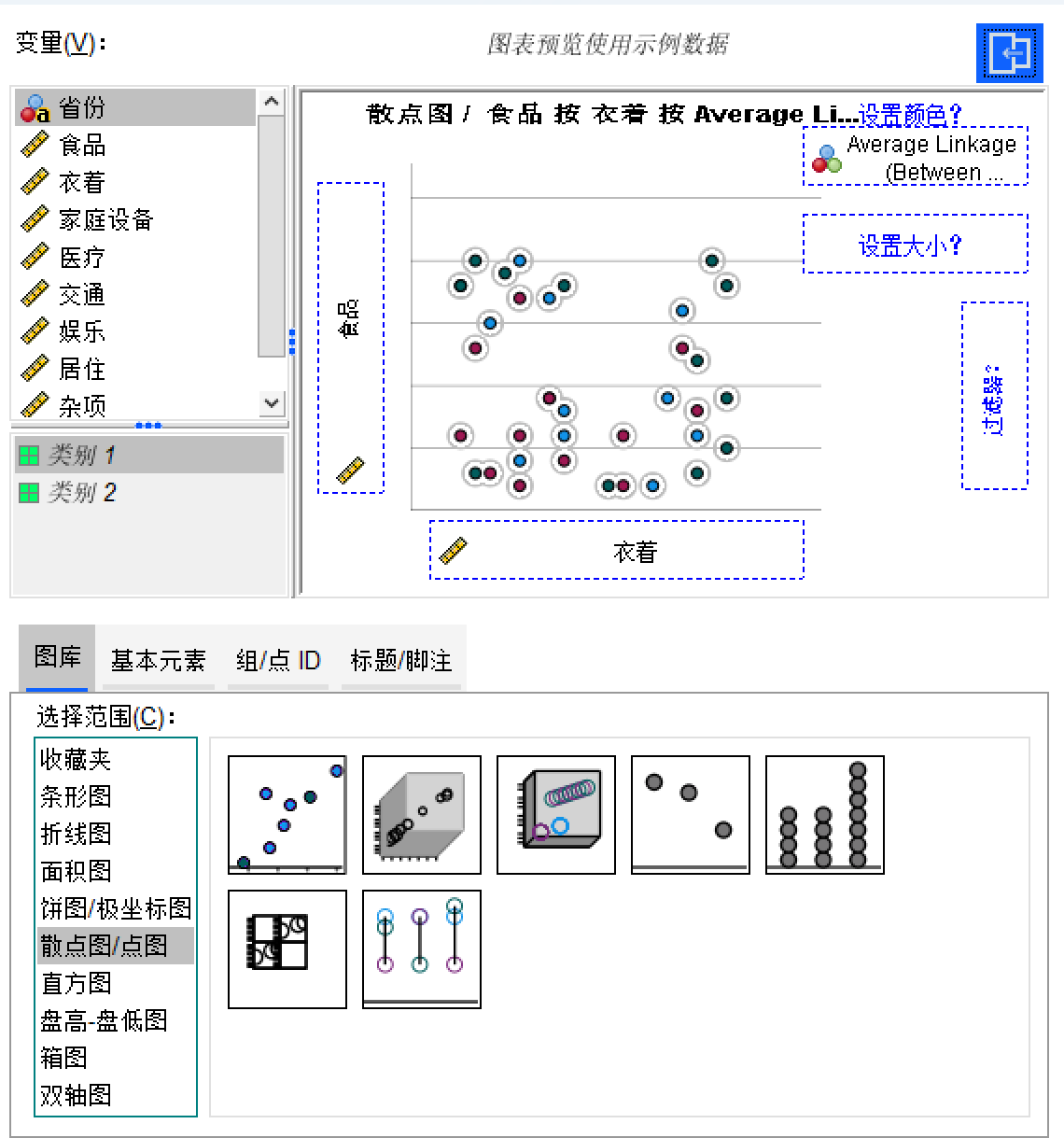
总所周知,SPSS默认的画出来的图并不美观,我们可以对其进行美化。包括但不限于把个案的颜色调整更鲜艳的颜色,修改标题等等

三、DBSCAN算法
DBSCAN算法是一种基于密度的聚类算法,其应用范围并不广泛;只有两个指标,且你做出散点图后发现数据表现得很“DBSCAN”,即能画出很明显的图形时,这时候再用DBSCAN进行聚类。
1.算法思想
该算法利用基于密度的聚类的概念,即要求聚类空间中的一定区域内所包含对象(点或其他空间对象)的数目不小于某一给定阈值。该方法能在具有噪声的空间数据库中发现任意形状的簇,可将密度足够大的相邻区域连接,能有效处理异常数据。
简单来说,把挨得近的点聚为一类
2.基本概念
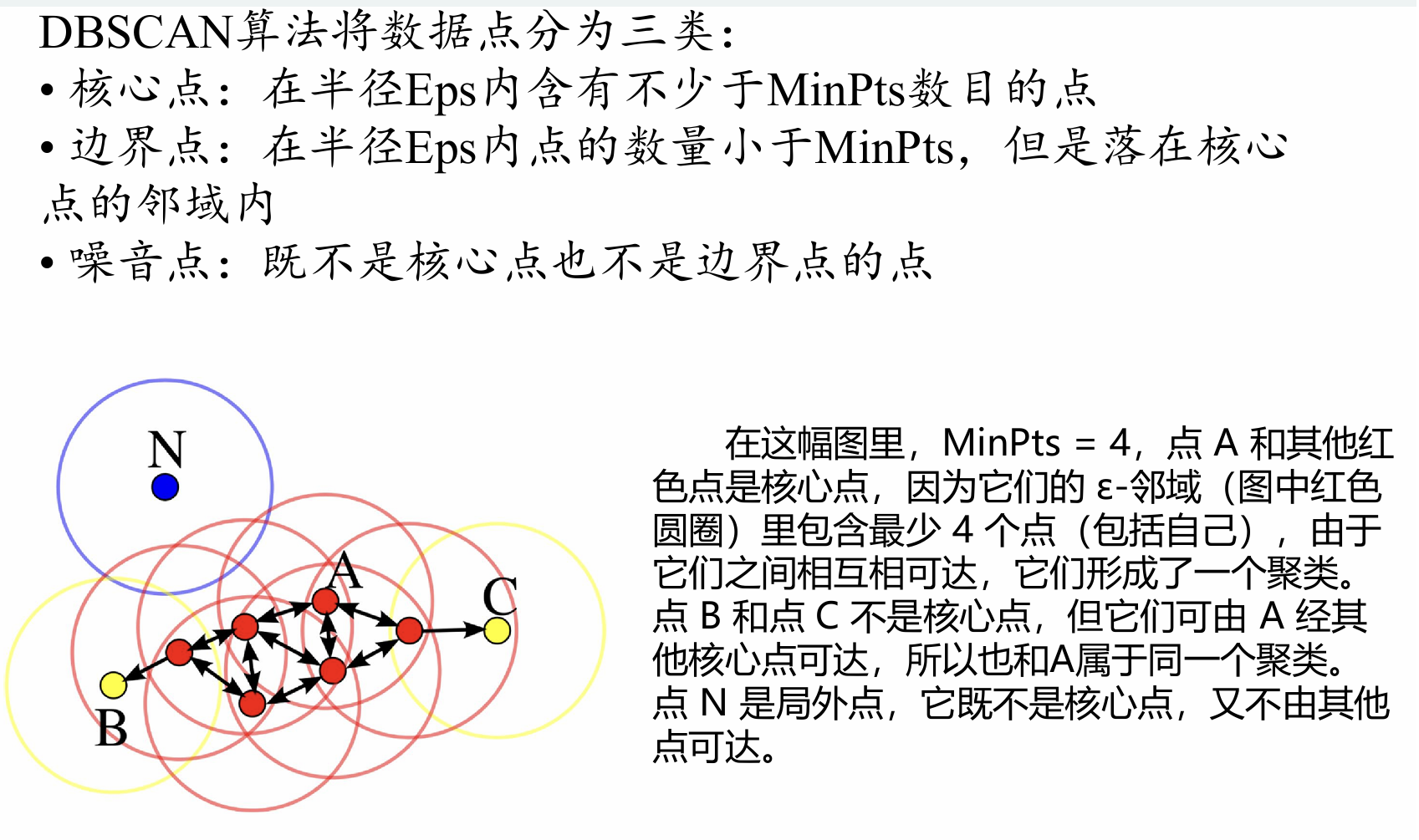
3.matlab实现过程
在matlab中没有现成的包让我们调用,于是我们自己编写了实现DBSCAN的代码(非本人编写),下面我们来介绍一下协和两个函数
函数的matlab代码在本文最后附上
(1)DBSCAN
[IDX, isnoise]=DBSCAN(X,epsilon,MinPts)
输入的X为由n行个案,m列个指标构成的矩阵。epsilon即为给定的半径,minpts为半径内至少含有的个案点的个数
IDX为我们最后的聚类结果,为一个由正整数构成的列向量。0代表噪声点,其余正数代表所属的类别。isnoise为是否是噪声点,这个参数我们也可以不输入
(2)PlotClusterinResult
PlotClusterinResult(X, IDX)
此函数是一个画图的函数,输入的X为原始数据,IDX为聚类后得到的自然数向量。输入这两个指标后可以可视化聚类结果(仅限两个指标)
(3)实例分析
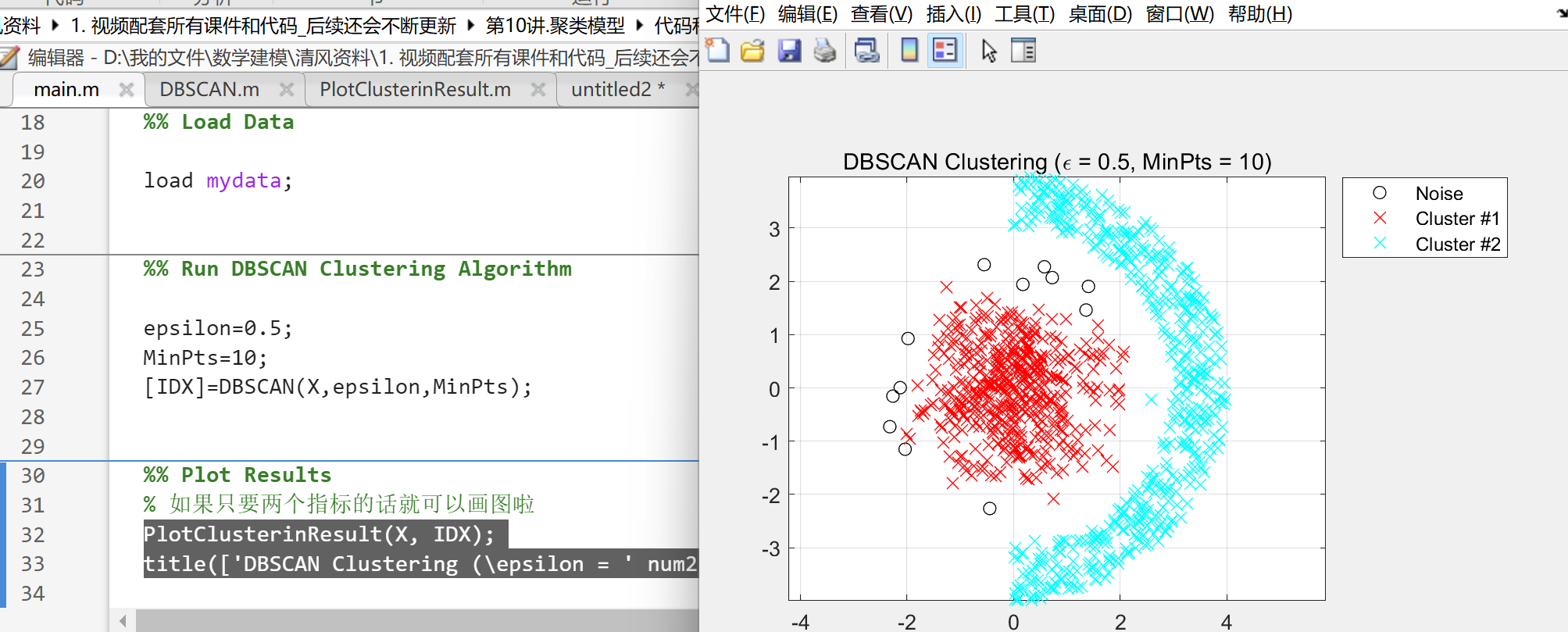
打包代码:
function [IDX, isnoise]=DBSCAN(X,epsilon,MinPts)
C=0;
n=size(X,1);
IDX=zeros(n,1); % 初始化全部为0,即全部为噪音点
D=pdist2(X,X);
visited=false(n,1);
isnoise=false(n,1);
for i=1:n
if ~visited(i)
visited(i)=true;
Neighbors=RegionQuery(i);
if numel(Neighbors)<MinPts
% X(i,:) is NOISE
isnoise(i)=true;
else
C=C+1;
ExpandCluster(i,Neighbors,C);
end
end
end
function ExpandCluster(i,Neighbors,C)
IDX(i)=C;
k = 1;
while true
j = Neighbors(k);
if ~visited(j)
visited(j)=true;
Neighbors2=RegionQuery(j);
if numel(Neighbors2)>=MinPts
Neighbors=[Neighbors Neighbors2]; %#ok
end
end
if IDX(j)==0
IDX(j)=C;
end
k = k + 1;
if k > numel(Neighbors)
break;
end
end
end
function Neighbors=RegionQuery(i)
Neighbors=find(D(i,:)<=epsilon);
end
end
function PlotClusterinResult(X, IDX)
k=max(IDX);
Colors=hsv(k);
Legends = {};
for i=0:k
Xi=X(IDX==i,:);
if i~=0
Style = 'x';
MarkerSize = 8;
Color = Colors(i,:);
Legends{end+1} = ['Cluster #' num2str(i)];
else
Style = 'o';
MarkerSize = 6;
Color = [0 0 0];
if ~isempty(Xi)
Legends{end+1} = 'Noise';
end
end
if ~isempty(Xi)
plot(Xi(:,1),Xi(:,2),Style,'MarkerSize',MarkerSize,'Color',Color);
end
hold on;
end
hold off;
axis equal;
grid on;
legend(Legends);
legend('Location', 'NorthEastOutside');
end


