1)介绍
在分析序列时,我们通常对特定的连续子序列感兴趣。 例如,a矢量可以被认为是按字母顺序排列的小写字母序列。 我们将第一个五个字母(a到e)称为连续的子序列,而仅包含元音的子序列不会是连续的。 分析任务仅关注区域的几何形状,而忽略基础序列值的情况并不少见。 索引列表是选择子序列的简单方法。 然而,连续子序列的稀疏表示将是一个范围,即开始位置和宽度的配对,如在用窗口提取序列时。Bioconductor中有两个中心类别可用于表示范围:IRanges类中定义的IRanges类用于表示单个空间上定义的范围,而GenomicRanges包中定义的GRanges类用于表示在多个空间上定义的范围。
2) IRanges objects
为了构造IRanges object, 需调用 IRanges 构造器. Ranges 有 三个参数:start, end and width。在实际应用中,一般指定2个参数就可以,第三个就会确定。
2.1)下面将用tstart, end and width 参数构建,组合构建IRanges object
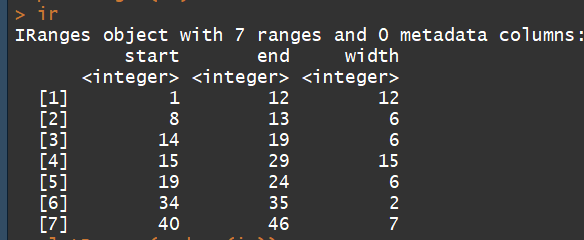
ir1 <- IRanges(start=1:10, width=10:1) ir2 <- IRanges(start=1:10, end=11) ir3 <- IRanges(end=11, width=10:1) identical(ir1, ir2) && identical(ir1, ir3) ir <- IRanges(c(1, 8, 14, 15, 19, 34, 40),width=c(12, 6, 6, 15, 6, 2, 7)) plot(range(ir))
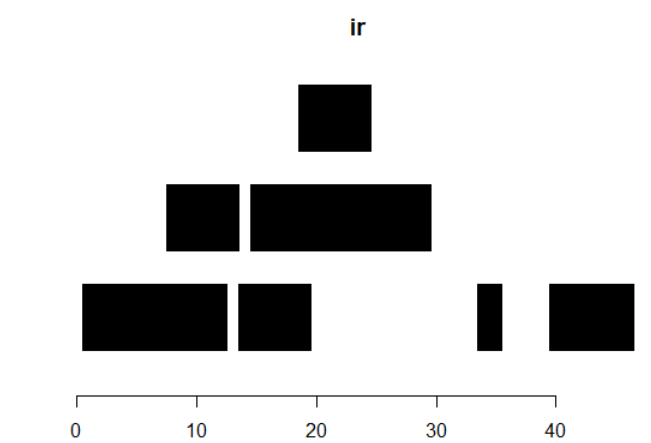
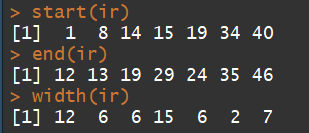
2.2)可以用starts, ends and widths函数接入 starts, ends and widths
start(ir) end(ir) width(ir)
2.3)通过数字和逻辑运算,抽取IRanges object 子集
ir[1:4] ir[start(ir) <= 15]
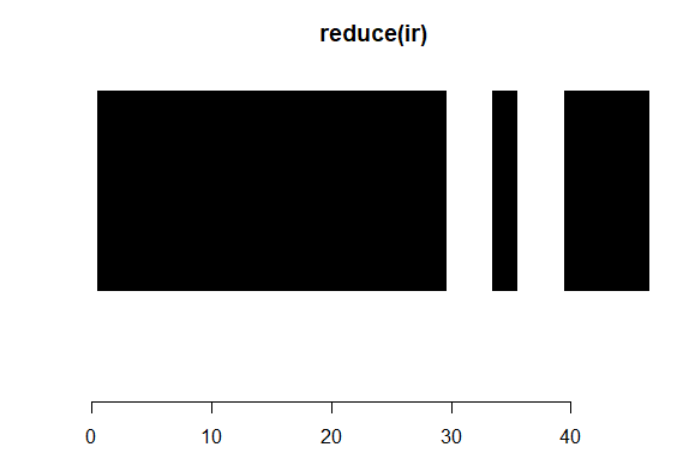
2.4)Normality :reduce函数通过合并冗余范围将任何IRanges简化为Normal IRanges。
reduce(ir) plotRanges(reduce(ir))