一 :准备工作
1)中文的 LaTeX - CTEX
2)LaTeX 的*.tex 的编辑工具-Texmarker
3)TEXshade宏包
二:安装
1) 直接双击运行下载的CTEX,安装过程中,可以不安装Ghostscript、GSview和WinEdt三个组件
2)双击安装Texmaker,直接下一步安装,点击“Close”完成
3)将解压texshade.zip
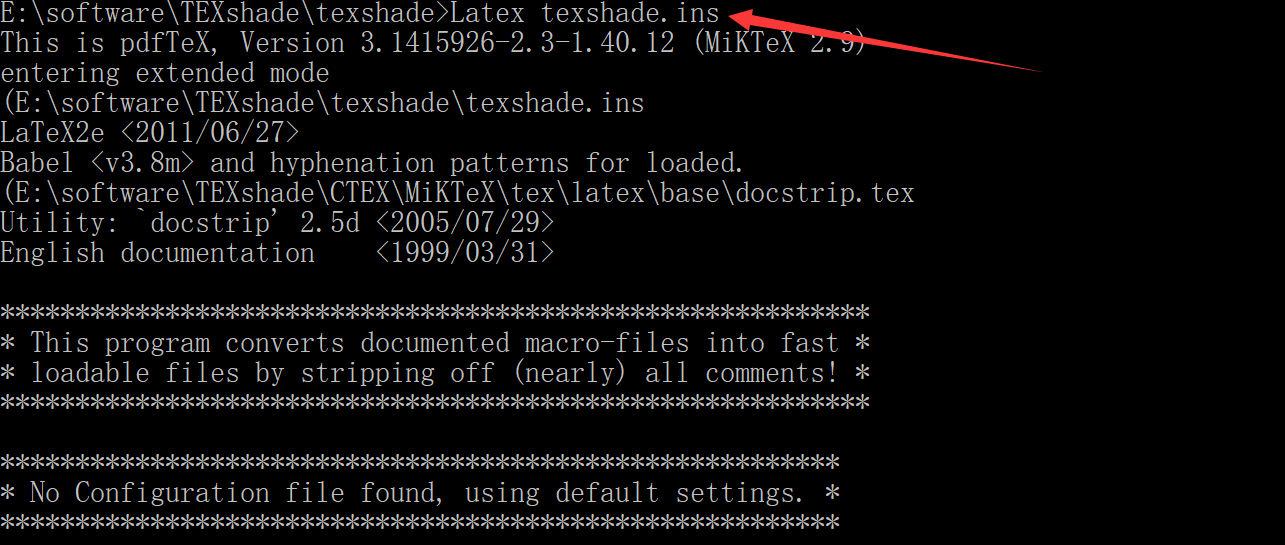
4) 在 Windows的 “开始”-运行中输入“cmd”,路径转到 TEXshade所在的目录下,此时输入“Latex texshade.ins”,回车,如果出现下图的信息,说明编译通过。
5)重复输入“Latex texshade.ins”再次编译,texshade.ins 编译2-3次后退出Windows命令行窗口,返回到TEXshade目录,此时比先前多了一些文件,这些是都编译生成的,如:AQPDNA.MSF 是比对的示例文件。
三:应用
很多人都会觉得这么漂亮的图肯定做出来很复杂,其实TEXShade需要的很简单,输出上面的结果其实也就是几分钟的事情,前提是需要你准备好需要展示的序列,记录好要标注的位置就OK!
例如:对于下面这个看似复杂的可视化结果,其实我们需要的代码很少,只需要5行代码就足够了!
上图对应的TEX代码:
第一行指定了我们的输入文件,通常是比对的结果文件
第二行是指定了TEXshade的显示模式,这里用根据 不同 functional groups的chemical properties标注颜色
第三行是表示只显示第一条序列(AQP1.PRO)138-170个碱基范围
第四号表示显示 legend
第五行表示代码结束
其实TEXshade能做的很多,要靠大家的想象,我一向认为,只要人能想得出,数据可视化就能做得到。
图像实例:





