详细可参考https://www.jianshu.com/p/2f125cdf8262;https://blog.csdn.net/qq_34296043/article/details/54427786两篇文章
1)下载网址:ftp://ftp.ncbi.nlm.nih.gov/blast/executables/blast+
2)解压到文件夹。此处为E:\software\blast_2.8.0_alpha\。会自动出现一个bin文件夹(放置程序,如下图),一个doc文件夹(放置文件)。然后自己创建一个Blastdb(放置数据库)
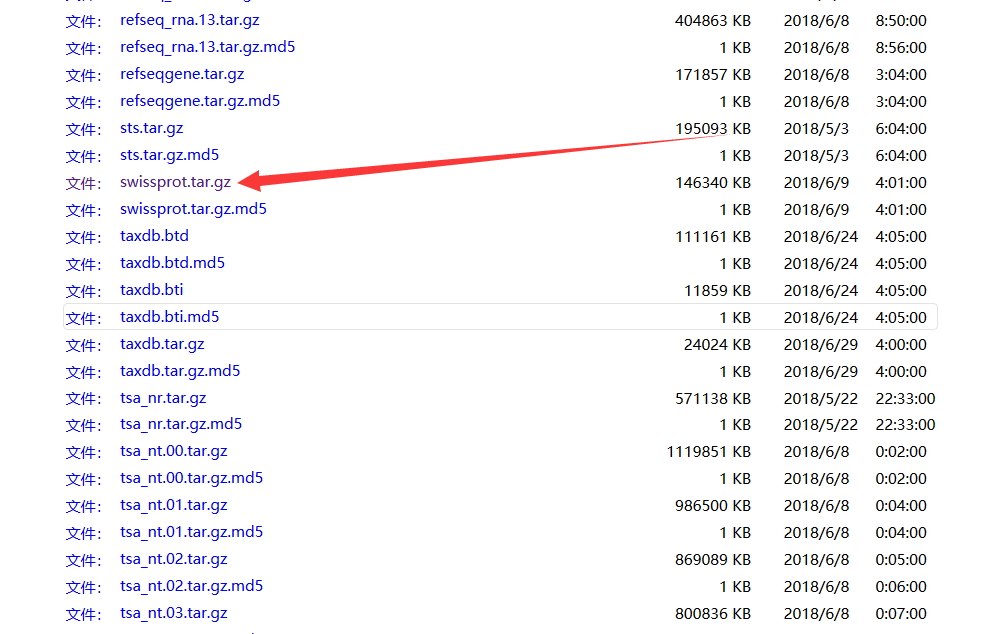
3)下载各种数据库,网址:ftp://ftp.ncbi.nlm.nih.gov/blast/db。本次下载的为swissprot及16S。解压缩到Blastdb下(这些下载的数据库已建好索引)。如果是自己的序列集合做数据库则需要建立索引。

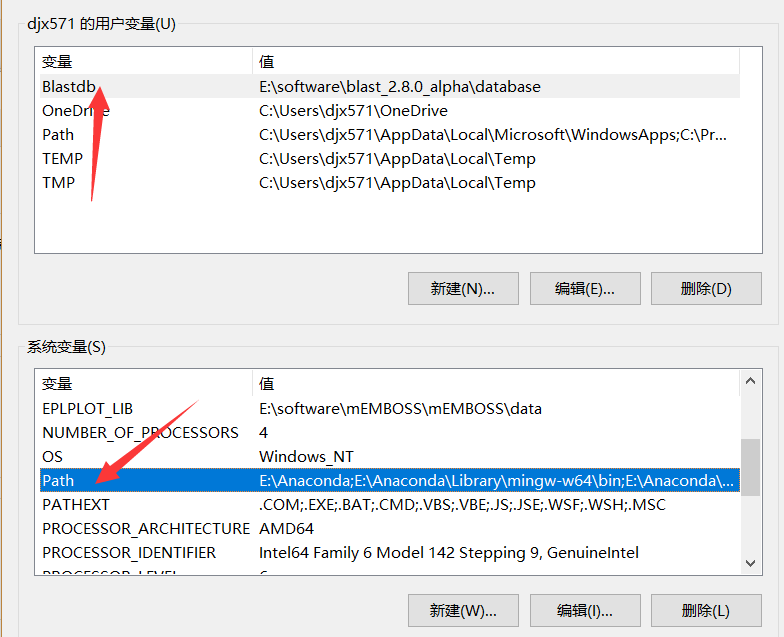
4)环境变量设置。我的电脑>鼠标右键>属性>高级系统设置>环境变量。然后1:在系统变量中“Path”添加变量值:E:\software\blast_2.8.0_alpha\bin(即bin文件所在的路径);2)在用户变量下方点击“新建”-变量名:Blastdb,变量值:E:\software\blast_2.8.0_alpha\blastdb(即数据库路径)
5)win+R然后输入cmd,然后enter键,进入dos界面
如果是自己的数据建库(fasta格式)需要首先建索引:
makeblastdb -in E:\software\blast_2.8.0_alpha\my_query_data\test_data_Unigene.fa -out E:\software\blast_2.8.0_alpha\database\PF_transcriptdb -dbtype nucl
其中:-in 参数后面为自己数据库(此处是一个转录组)所在的绝对路径,-out是索引结果输出路径(会输出三个文件.pin;.phr;.psq),-dbtype是数据类型,核酸用nucl,蛋白用prot。
tblastx -query E:\software\blast_2.8.0_alpha\my_query_data\nac019.txt -db PF_transcriptdb -out E:\software\blast_2.8.0_alpha\my_query_data\result_n.txt -outfmt 7 -evalue 1e-5
其中-query是你自己的query序列所在的绝对路径;-db 上一步所建立的数据库;-out 是输出文件路径;-outfmt 输出格式;6表示不加注释的m8格式,7表示加注释的m8格式;-num_threads:线程数,笔记本不要设大了,2就够了;-perc_identity :比对的最低相似度。
如果是和NCBI 下载的数据库比对:
blastp -query E:\software\blast_2.8.0_alpha\my_query_data\test.fasta -db swissprot -out E:\software\blast_2.8.0_alpha\my_query_data\out.txt -outfmt 7 -evalue 1e-10
blastn -query E:\software\blast_2.8.0_alpha\my_query_data\Macromonas.fa -db 16SMicrobial -out E:\software\blast_2.8.0_alpha\my_query_data\out.txt -outfmt 7 -evalue 1e-90
其中-db swissprot,-db 16SMicrobial 就是上一步NCBI下载解压过(自己本身就带有索引,不用自己建),且因为数据库Database路径已经加入了环境变量,因此这里不用写绝对路径。


