以脚本形式运行python库
技术背景
当我们尝试运行python的帮助文档时,会看到如下这样的一个说明:
$ python3 -h
usage: python3 [option] ... [-c cmd | -m mod | file | -] [arg] ...
Options and arguments (and corresponding environment variables):
...
-m mod : run library module as a script (terminates option list)
...
这一个条目的意思是,我们可以使用python3 -m这样的指令,在终端的命令行内运行python的一些仓库。比如我们常用的pip,就可以通过python3 -m pip install numpy这样的操作指令来运行。还有一个比较常见的上传python编译安装包到pypi网站上面的工具twine,可以通过python3 -m twine的方法来使用。本文我们主要探讨一下如何在代码中,实现python3 -m 这种命令行运行的模式。
基础功能代码实现
通过python3 -m 这样的方法来运行,本质上只是一个实现方式的改变,而不影响到具体算法的实现,这个形式跟我们直接通过python的API接口去调用是一样的。所以我们需要先按照正常的API接口调用的方法,先把基础代码模块写好。这里我们使用一个开源代码仓库hadder为例,来介绍一下具体的操作方法。我们先看一下Hadder的具体代码架构与相关模块内容:
$ tree hadder/
hadder/
├── examples # 示例
│ ├── case1-complete.pdb
│ ├── case1.pdb
│ ├── case2-complete.pdb
│ ├── case2-complete.png
│ ├── case2.pdb
│ └── case2.png
├── hadder # 根目录
│ ├── constants.py # 存放一些常数
│ ├── __init__.py # 核心算法
│ └── parsers.py # 读取PDB文件
├── LICENSE
├── README.md
├── requirements.txt
└── setup.py
2 directories, 13 files
这里先简单说明一下背景,关于Hadder的具体内容和算法,可以参考这篇博客。Hadder是一个用于给PDB文件补氢原子的小工具,因为在蛋白质折叠的预测过程中,主要以骨架为主,因此氢原子大部分情况下是被忽略的。而在后期建立蛋白质力场的时候,氢原子又是必须使用到的,因此我们可以用hadder这样一个工具来实现补氢的功能。关于hadder我们就不进行更多的介绍了,主要看下其API接口的调用方法:
from hadder import AddHydrogen
AddHydrogen('input.pdb', 'output.pdb')
对外开放的API接口就这么一个,较为简单。接下来我们就可以基于这个功能模块,去创建一个可以通过命令行来运行的方法。
创建__main__.py文件
当我们使用python3 -m模式来运行的时候,python会去自动索引到__main__.py这个文件作为入口文件,因此首先我们在根目录下创建一个__main__.py文件,如下所示:
$ tree hadder/
hadder/
├── examples # 示例
│ ├── case1-complete.pdb
│ ├── case1.pdb
│ ├── case2-complete.pdb
│ ├── case2-complete.png
│ ├── case2.pdb
│ └── case2.png
├── hadder # 根目录
│ ├── constants.py # 存放一些常数
│ ├── __init__.py # 核心算法
│ ├── __main__.py # python -m 模式运行接口文件
│ └── parsers.py # 读取PDB文件
├── LICENSE
├── README.md
├── requirements.txt
└── setup.py
2 directories, 14 files
然后我们就可以在__main__.py文件中结合argparse来使用,实现一个命令行模式运行的功能,如下是__main__.py文件中的代码内容:
# __main__.py
import argparse
from hadder import AddHydrogen
parser = argparse.ArgumentParser()
parser.add_argument("-i", help="Set the input pdb file path.")
parser.add_argument("-o", help="Set the output pdb file path.")
args = parser.parse_args()
pdb_name = args.i
save_pdb_name = args.o
AddHydrogen(pdb_name, save_pdb_name)
我们还是同样的调用AddHydrogen这个API接口,但是由于使用了argparse,使得我们可以在命令行里面输入相关的输入文件路径和输出文件路径。最终运行效果如下:
$ python3 -m hadder -h
usage: __main__.py [-h] [-i I] [-o O]
optional arguments:
-h, --help show this help message and exit
-i I Set the input pdb file path.
-o O Set the output pdb file path.
$ python3 -m hadder -i input.pdb -o ouput.pdb
1 H-Adding task with 3032 atoms complete in 0.116 seconds.
感兴趣的也可以看看使用这个算法加氢前后的构象区别:
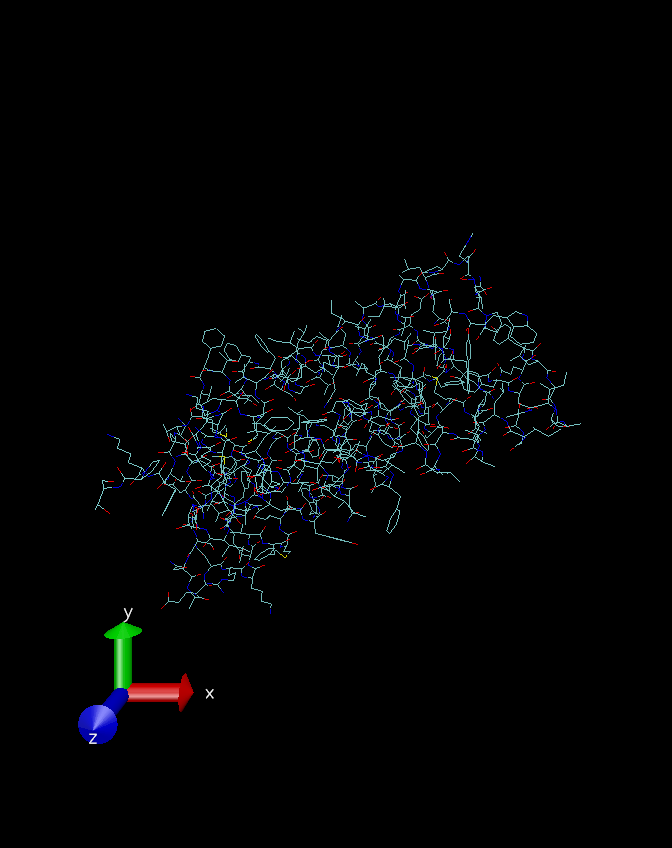
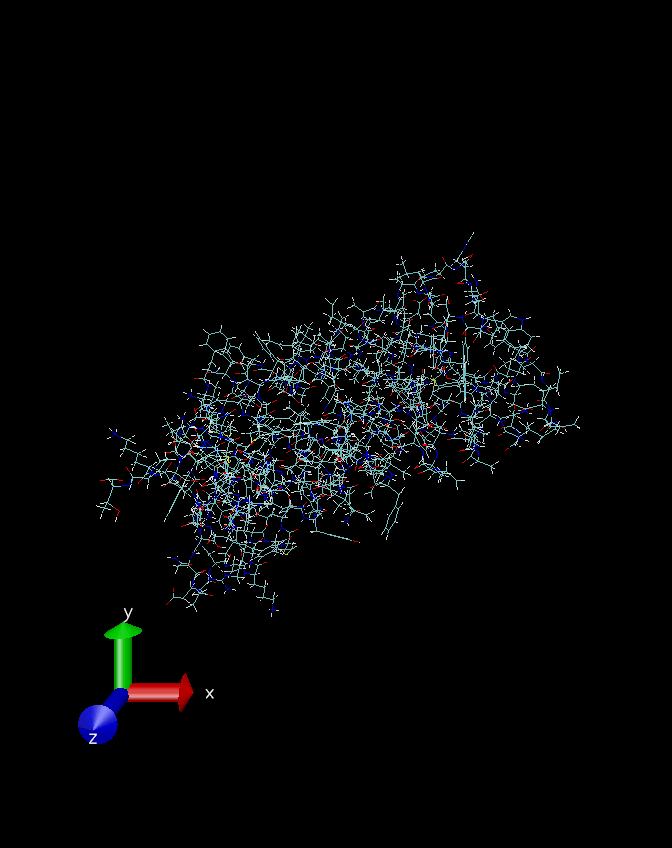
补充
一般我们完成了一个算法实现,需要开放给别人使用的时候。以python为例,最方便的做法是将python仓库编译后上传到pypi网站上面,这样大家可以使用pip来进行安装和管理。这里我们补充一个编译上传python仓库的“三步走”方法:
$ python3 setup.py check
$ python3 setup.py sdist bdist_wheel
$ twine upload --repository-url https://upload.pypi.org/legacy/ dist/*
这样一来,我们就可以通过pip来对我们的仓库进行安装和管理,比如可以使用如下的指令安装hadder:
$ python3 -m pip install hadder --upgrade
总结概要
本文主要通过一个实际的案例,介绍了如何可以在命令行中调用和运行我们的python模块。“python -m”这个方案为我们提供了一个新的选项,这个运行方法以“main.py”文件为入口文件运行,结合python中常用的命令行工具argparse,我们就可以很容易的创建一个可以通过命令行运行和获取参数的python模块。并且可以使用twine上传到pypi网站上,用pip进行安装和管理,会更加的便捷。
版权声明
本文首发链接为:https://www.cnblogs.com/dechinphy/p/pym.html
作者ID:DechinPhy
更多原著文章请参考:https://www.cnblogs.com/dechinphy/
打赏专用链接:https://www.cnblogs.com/dechinphy/gallery/image/379634.html
腾讯云专栏同步:https://cloud.tencent.com/developer/column/91958
CSDN同步链接:https://blog.csdn.net/baidu_37157624?spm=1008.2028.3001.5343
51CTO同步链接:https://blog.51cto.com/u_15561675


 本文主要通过一个实际的案例,介绍了如何可以在命令行中调用和运行我们的python模块。“python -m”这个方案为我们提供了一个新的选项,这个运行方法以“__main__.py”文件为入口文件运行,结合python中常用的命令行工具argparse,我们就可以很容易的创建一个可以通过命令行运行和获取参数的python模块。并且可以使用twine上传到pypi网站上,用pip进行安装和管理,会更加的便捷。
本文主要通过一个实际的案例,介绍了如何可以在命令行中调用和运行我们的python模块。“python -m”这个方案为我们提供了一个新的选项,这个运行方法以“__main__.py”文件为入口文件运行,结合python中常用的命令行工具argparse,我们就可以很容易的创建一个可以通过命令行运行和获取参数的python模块。并且可以使用twine上传到pypi网站上,用pip进行安装和管理,会更加的便捷。

 浙公网安备 33010602011771号
浙公网安备 33010602011771号