PCA主成分分析的Python实现
技术背景
PCA主成分分析在数据处理和降维中经常被使用到,是一个非常经典的降维算法,本文提供一个PCA降维的流程分解,和对应的Python代码实现。
二维数据生成
如果没有自己的测试数据,我们可以生成一些特殊的随机数据点。例如我们使用Numpy的均匀随机数生成一系列二维的数据点\(\mathbf{r}=\left(x,y\right)\),其中数据点分布在一个椭圆内:
生成数据点的Python代码如下所示:
import numpy as np
def plot_points(data):
import matplotlib.pyplot as plt
plt.figure()
plt.xlim(-1, 1)
plt.ylim(-1, 1)
plt.plot(data[:, 0], data[:, 1], '.', color='blue')
plt.savefig("PCA.png")
def data_generator(nodes, seed=0):
np.random.seed(seed)
data = 2 * np.random.random((nodes, 2)) - 1
mask_index = np.where(data[:,0]**2+4*data[:,1]**2<=1)[0]
masked_data = data[mask_index]
return masked_data
if __name__ == "__main__":
masked_data = data_generator(1000)
plot_points(masked_data)
运行上述代码会在当前路径下生成一个PCA.png的图片,结果如下所示:

数据标准化
因为不同类型的数据有不同的范围和特征,可以做一个标准化方便后续处理,但标准化之后的数据,记得降维之后要进行还原:
这是\(x\)方向的标准化,\(y\)方向的标准化同理,最终可以得到\(\mathbf{Z}=\left(\mathbf{x}',\mathbf{y}'\right)\)。对应的Python函数实现为:
def normalization(data):
data_avg = np.average(data, axis=0)
data_shift = data - data_avg
output = np.zeros_like(data)
for i in range(data.shape[-1]):
output[:, i] = data_shift[:, i] / np.sqrt(np.sum(data_shift[:, i] ** 2)/(data.shape[0]-1))
return output
协方差矩阵
正常我们写样本协方差矩阵的形式是这样的:
但是因为前面已经分别得到了两个方向的标准化数据,所以我们直接用下面这个公式计算就可以了:
对应的Python实现为:
def cov_matrix(Z):
return (Z.T @ Z) / (Z.shape[0] - 1)
如果对标准化之后的数据计算一个协方差矩阵可以得到:
[[1. 0.04955086]
[0.04955086 1. ]]
特征值分解
关于特征值分解的内容,可以参考上一篇文章中的介绍。总体来说就是把一个矩阵分解为如下形式:
其中\(\Sigma\)是由本征值组成的对角矩阵,\(U\)是由本征列向量组成的本征矩阵。对应的Python代码实现为:
def eig_decomp(C):
vals, vecs = np.linalg.eig(C)
sort_idx = np.argsort(vals)
return np.diag(vals), vecs, sort_idx
如果对上面的协方差矩阵做一个特征值分解,可以得到输出的特征值为:
[[1.04955086 0. ]
[0. 0.95044914]]
输出的特征列向量组成的矩阵\(U\)为:
[[ 0.70710678 -0.70710678]
[ 0.70710678 0.70710678]]
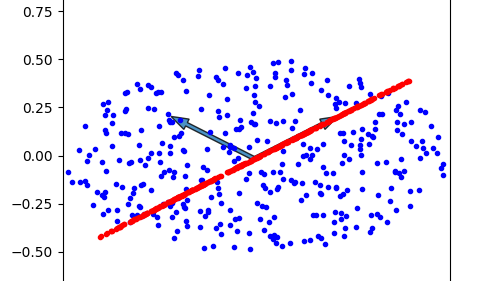
可以把这两个列向量画到数据点中:

PCA降维
根据上面生成的对角化矩阵,我们已经可以从中筛选一些比较大的值和对应的本征向量,作为数据点的“主要成分”。一般是在本征值之间的值差异较大的时候可以更好的降维,这里生成的数据其实两个本征值没有很好的降维效果,但是我们依然可以执行降维的操作。例如我们选取第一个本征向量\(\mathbf{v}\)作为投影空间,把所有的数据点都投影到这个向量上:
就可以完成对数据点的降维,效果如下所示:

所用到的完整Python代码示例如下:
PCA Python完整代码
import numpy as np
def plot_points(data):
import matplotlib.pyplot as plt
plt.figure()
plt.xlim(-1, 1)
plt.ylim(-1, 1)
plt.plot(data[:, 0], data[:, 1], '.', color='blue')
plt.savefig("PCA.png")
def plot_vec(data, center, vecs):
import matplotlib.pyplot as plt
plt.figure()
plt.xlim(-1, 1)
plt.ylim(-1, 1)
plt.plot(data[:, 0], data[:, 1], '.', color='blue')
for i in range(vecs.shape[-1]):
plt.arrow(center[0], center[1], vecs[:,i][0], vecs[:,i][1], width=0.02, alpha=0.8)
plt.savefig("PCA.png")
def plot_reduced(data, center, vec_proj, vecs):
import matplotlib.pyplot as plt
plt.figure(figsize=(5,5))
plt.xlim(-1, 1)
plt.ylim(-1, 1)
plt.plot(data[:, 0], data[:, 1], '.', color='blue')
vec_proj += center
plt.plot(vec_proj[:, 0], vec_proj[:, 1], '.', color='red')
for i in range(vecs.shape[-1]):
plt.arrow(center[0], center[1], vecs[:,i][0], vecs[:,i][1], width=0.02, alpha=0.8)
plt.savefig("PCA.png")
def data_generator(nodes, seed=0):
np.random.seed(seed)
data = 2 * np.random.random((nodes, 2)) - 1
mask_index = np.where(data[:,0]**2+4*data[:,1]**2<=1)[0]
masked_data = data[mask_index]
return masked_data
def normalization(data):
data_avg = np.average(data, axis=0)
data_shift = data - data_avg
output = np.zeros_like(data)
sigmai = np.zeros(data.shape[-1])
for i in range(data.shape[-1]):
sigmai[i] = np.sqrt(np.sum(data_shift[:, i] ** 2)/(data.shape[0]-1))
output[:, i] = data_shift[:, i] / sigmai[i]
return output, data_avg, sigmai
def cov_matrix(Z):
return (Z.T @ Z) / (Z.shape[0] - 1)
def eig_decomp(C):
vals, vecs = np.linalg.eig(C)
sort_idx = np.argsort(vals)
return np.diag(vals), vecs, sort_idx
def dimension_reduction(data, center, v):
return np.einsum('ij,j->i', data-center, v/np.linalg.norm(v))[:,None] * v[None]/np.linalg.norm(v)
if __name__ == "__main__":
masked_data = data_generator(1000)
normalized_data, center, sigmai = normalization(masked_data)
C = cov_matrix(normalized_data)
Sigma, U, idx = eig_decomp(C)
reduced_data = dimension_reduction(masked_data, center, (U*sigmai[:,None])[:, 0])
plot_reduced(masked_data, center, reduced_data, U*sigmai[:,None])
总结概要
接上一篇文章介绍的矩阵特征分解,本文介绍了矩阵特征分解在主成分分析(PCA)算法中的应用。对于PCA算法,最直观的理解就是,在高维数据中找到一个低维的空间,使得所有的数据点投影到该低维空间之后尽可能的分离。
版权声明
本文首发链接为:https://www.cnblogs.com/dechinphy/p/pca.html
作者ID:DechinPhy
更多原著文章:https://www.cnblogs.com/dechinphy/
请博主喝咖啡:https://www.cnblogs.com/dechinphy/gallery/image/379634.html


 接上一篇文章介绍的矩阵特征分解,本文介绍了矩阵特征分解在主成分分析(PCA)算法中的应用。对于PCA算法,最直观的理解就是,在高维数据中找到一个低维的空间,使得所有的数据点投影到该低维空间之后尽可能的分离。
接上一篇文章介绍的矩阵特征分解,本文介绍了矩阵特征分解在主成分分析(PCA)算法中的应用。对于PCA算法,最直观的理解就是,在高维数据中找到一个低维的空间,使得所有的数据点投影到该低维空间之后尽可能的分离。

 浙公网安备 33010602011771号
浙公网安备 33010602011771号