使用SpongeExt快捷完成CudaSPONGE结合PySAGES的增强采样
技术背景
在前面的一些文章中,我们介绍过关于CudaSPONGE的安装和基础使用方法,CudaSPONGE提供的Python接口,PySAGES增强采样软件的基本使用方法,还有一篇关于CudaSPONGE和PySAGES相结合的文章,并且做了一些简单的CudaSPONGE结合PySAGES的性能测试。其中因为CudaSPONGE与PySAGES的接口实际上是可以多场景共用的,因此考虑把这部分的接口单独封装一个包出来,使得用户可以尽可能无感的在CudaSPONGE中使用PySAGES。
Pip安装
SpongeExt最新的源码存储地址为:https://gitee.com/dechin/sponge-ext/tree/master,都是非常简单的Python代码,只是对环境有一些先置条件,例如Jax、Jaxlib、Cupy等,当然,PySAGES也是必须先安装好的。用户可以下载源码进行编译安装,也可以直接用pip安装:
$ python3 -m pip install SpongeExt --upgrade
没有报错即为安装成功。
使用方法
在开源仓库的example路径中,我们已经存放了一个完整的演示案例。其目录结构大致是这样的:
.
|-- README.md
|-- protein
| |-- alad_LJ.txt
| |-- alad_angle.txt
| |-- alad_atom_name.txt
| |-- alad_atom_type_name.txt
| |-- alad_bond.txt
| |-- alad_charge.txt
| |-- alad_coordinate.txt
| |-- alad_dihedral.txt
| |-- alad_exclude.txt
| |-- alad_mass.txt
| |-- alad_nb14.txt
| |-- alad_residue.txt
| `-- alad_resname.txt
|-- pysages_metad.py
`-- workspace
|-- mdbox.txt
|-- mdcrd.dat
|-- mdinfo.txt
|-- mdout.txt
|-- nvt.in
|-- nvt_restart_coordinate.txt
`-- nvt_restart_velocity.txt
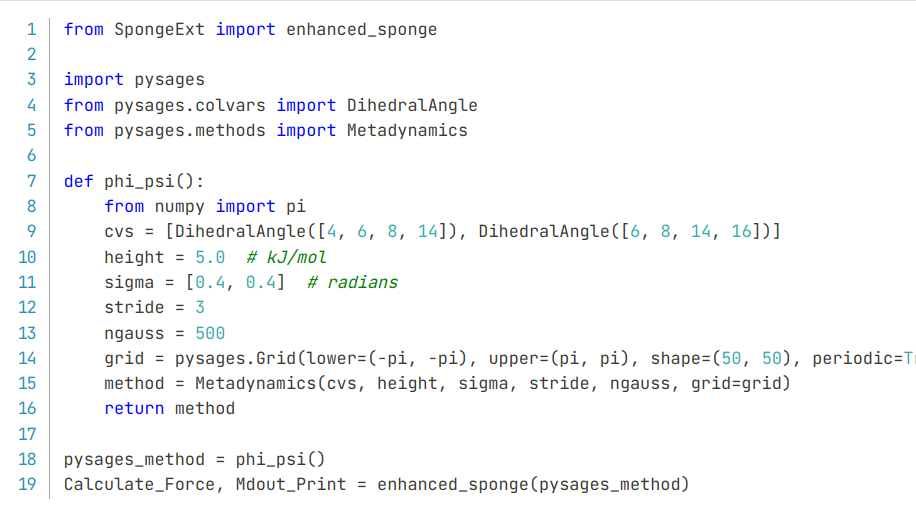
其中pysages_metad.py就是我们所定义的PySAGES增强采样方法,文件内容是这样的:
from SpongeExt import enhanced_sponge
import pysages
from pysages.colvars import DihedralAngle
from pysages.methods import Metadynamics
def phi_psi():
from numpy import pi
cvs = [DihedralAngle([4, 6, 8, 14]), DihedralAngle([6, 8, 14, 16])]
height = 5.0 # kJ/mol
sigma = [0.4, 0.4] # radians
stride = 3
ngauss = 500
grid = pysages.Grid(lower=(-pi, -pi), upper=(pi, pi), shape=(50, 50), periodic=True)
method = Metadynamics(cvs, height, sigma, stride, ngauss, grid=grid)
return method
pysages_method = phi_psi()
Calculate_Force, Mdout_Print = enhanced_sponge(pysages_method)
可以看到的是,我们只是调用了SpongeExt中的一个生成函数,没有在该脚本下直接调用CudaSPONGE底层的任何参数。而在PySAGES这边,也仅仅是定义了一个MetaDynamics的方法,没有定义任何其他PySAGES底层的SnapShot之类的参数,使用SpongeExt这个插件可以做到一个几乎无感的衔接。在protein/路径下存放的是我们使用Xponge生成的CudaSPONGE运行所需的输入参数文件。在workspace/路径下是我们运行的一个普通NVT的CudaSPONGE参数配置文件,其中只有一个nvt.in文件是我们需要配置的参数输入文件,其他文件都是通过CudaSPONGE运行产生的输出文件。nvt.in的文件内容为:
case1 MD simulation
mode = NVT
default_in_file_prefix = ../protein/alad
pbc=0
cutoff=999
dt = 1e-3
step_limit = 2000
write_information_interval = 10
thermostat = middle_langevin
middle_langevin_gamma = 10
rst = nvt_restart
coordinate_in_file = ../protein/alad_coordinate.txt
plugin = /usr/local/python-3.7.5/lib/python3.7/site-packages/prips/_prips.so
py = ../pysages_metad.py
相比于普通的NVT过程,这里只是多配置了plugin和py这两个参数,意指通过prips这个插件来调用python文件中的相关接口函数,从而达到一个外接软件实现增强采样方法的效果。
总结概要
基于CudaSPONGE高性能分子动力学模拟采样工具,和PySAGES高性能增强采样软件,我简单的封装了一个SpongeExt插件,可以用于无感结合CudaSPONGE和PySAGES进行增强采样分子动力学模拟。
版权声明
本文首发链接为:https://www.cnblogs.com/dechinphy/p/sponge-ext.html
作者ID:DechinPhy
更多原著文章:https://www.cnblogs.com/dechinphy/
请博主喝咖啡:https://www.cnblogs.com/dechinphy/gallery/image/379634.html
参考链接
本文作者:Dechin的博客
本文链接:https://www.cnblogs.com/dechinphy/p/18687146/sponge-ext
版权声明:本作品采用CC BY-NC-SA 4.0许可协议进行许可。


 基于CudaSPONGE高性能分子动力学模拟采样工具,和PySAGES高性能增强采样软件,我简单的封装了一个SpongeExt插件,可以用于无感结合CudaSPONGE和PySAGES进行增强采样分子动力学模拟。
基于CudaSPONGE高性能分子动力学模拟采样工具,和PySAGES高性能增强采样软件,我简单的封装了一个SpongeExt插件,可以用于无感结合CudaSPONGE和PySAGES进行增强采样分子动力学模拟。






【推荐】编程新体验,更懂你的AI,立即体验豆包MarsCode编程助手
【推荐】凌霞软件回馈社区,博客园 & 1Panel & Halo 联合会员上线
【推荐】抖音旗下AI助手豆包,你的智能百科全书,全免费不限次数
【推荐】轻量又高性能的 SSH 工具 IShell:AI 加持,快人一步