随笔分类 - 分子动力学
使用SpongeExt快捷完成CudaSPONGE结合PySAGES的增强采样
摘要: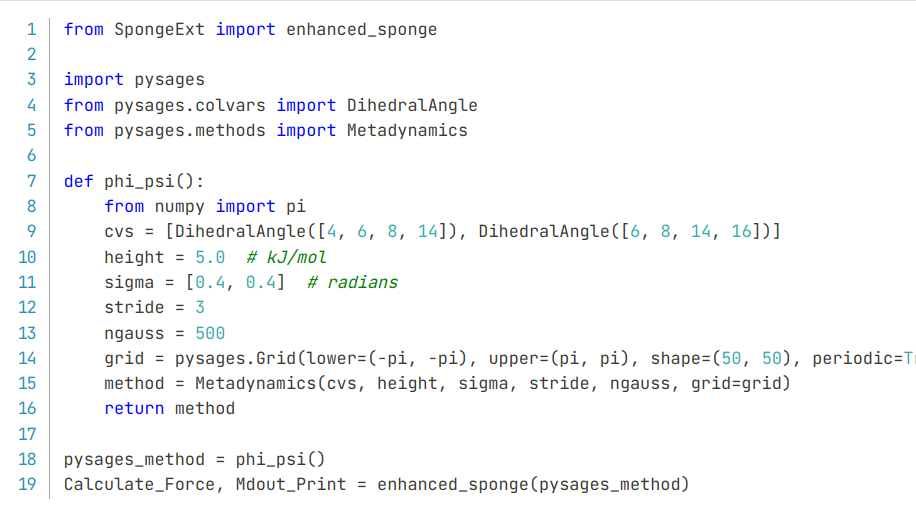 基于CudaSPONGE高性能分子动力学模拟采样工具,和PySAGES高性能增强采样软件,我简单的封装了一个SpongeExt插件,可以用于无感结合CudaSPONGE和PySAGES进行增强采样分子动力学模拟。
基于CudaSPONGE高性能分子动力学模拟采样工具,和PySAGES高性能增强采样软件,我简单的封装了一个SpongeExt插件,可以用于无感结合CudaSPONGE和PySAGES进行增强采样分子动力学模拟。
 基于CudaSPONGE高性能分子动力学模拟采样工具,和PySAGES高性能增强采样软件,我简单的封装了一个SpongeExt插件,可以用于无感结合CudaSPONGE和PySAGES进行增强采样分子动力学模拟。
基于CudaSPONGE高性能分子动力学模拟采样工具,和PySAGES高性能增强采样软件,我简单的封装了一个SpongeExt插件,可以用于无感结合CudaSPONGE和PySAGES进行增强采样分子动力学模拟。
CudaSPONGE与PySAGES初步性能测试
摘要: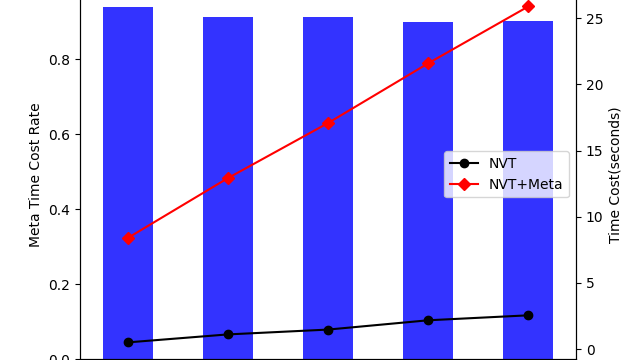 接前一篇关于PySAGES结合CudaSPONGE使用方法的文章,本文主要还是使用了一样的测试案例。仅通过不同的测试步长,来定性的分析PySAGES的MetaDynamics实现方案结合MD软件之后的性能数据。
接前一篇关于PySAGES结合CudaSPONGE使用方法的文章,本文主要还是使用了一样的测试案例。仅通过不同的测试步长,来定性的分析PySAGES的MetaDynamics实现方案结合MD软件之后的性能数据。
 接前一篇关于PySAGES结合CudaSPONGE使用方法的文章,本文主要还是使用了一样的测试案例。仅通过不同的测试步长,来定性的分析PySAGES的MetaDynamics实现方案结合MD软件之后的性能数据。
接前一篇关于PySAGES结合CudaSPONGE使用方法的文章,本文主要还是使用了一样的测试案例。仅通过不同的测试步长,来定性的分析PySAGES的MetaDynamics实现方案结合MD软件之后的性能数据。
PySAGES结合CUDA SPONGE增强采样
摘要: 本文探索并梳理了一下CUDA SPONGE高性能分子模拟采样软件,和PySAGES高性能增强采样软件,这两者强强联合的MD模拟新范式。
本文探索并梳理了一下CUDA SPONGE高性能分子模拟采样软件,和PySAGES高性能增强采样软件,这两者强强联合的MD模拟新范式。
 本文探索并梳理了一下CUDA SPONGE高性能分子模拟采样软件,和PySAGES高性能增强采样软件,这两者强强联合的MD模拟新范式。
本文探索并梳理了一下CUDA SPONGE高性能分子模拟采样软件,和PySAGES高性能增强采样软件,这两者强强联合的MD模拟新范式。
CudaSPONGE之Python接口
摘要: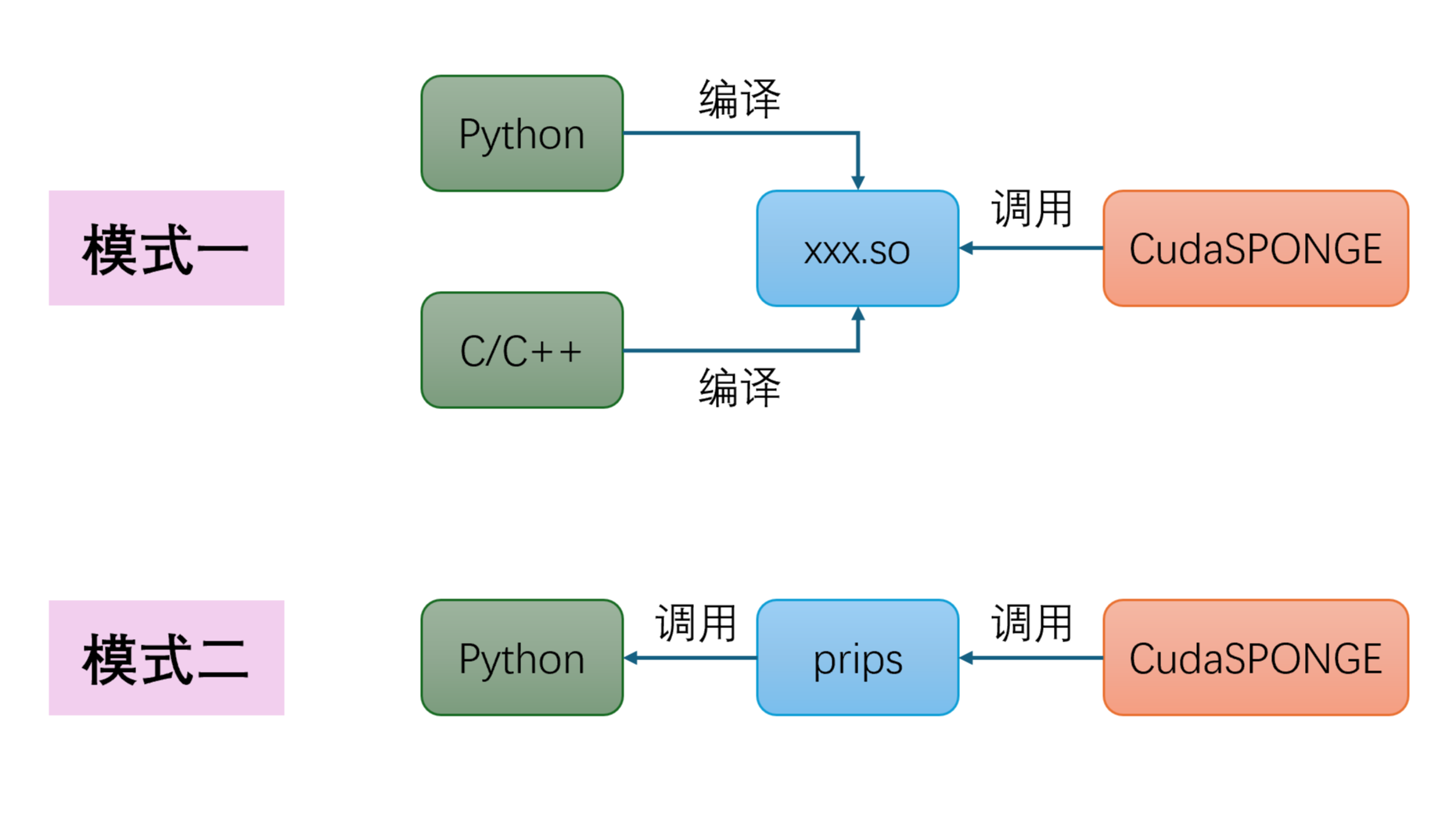 本文介绍了高性能GPU分子动力学模拟软件CudaSPONGE的Python API接口,通过官方开发的prips插件,使得我们可以在Python框架下很方便的开发一些分子动力学模拟的Force Wrapper,例如Meta Dynamics中就有很多可以外界的工具,非常方便开发者的二次开发,同时又能够兼顾到性能。
本文介绍了高性能GPU分子动力学模拟软件CudaSPONGE的Python API接口,通过官方开发的prips插件,使得我们可以在Python框架下很方便的开发一些分子动力学模拟的Force Wrapper,例如Meta Dynamics中就有很多可以外界的工具,非常方便开发者的二次开发,同时又能够兼顾到性能。
 本文介绍了高性能GPU分子动力学模拟软件CudaSPONGE的Python API接口,通过官方开发的prips插件,使得我们可以在Python框架下很方便的开发一些分子动力学模拟的Force Wrapper,例如Meta Dynamics中就有很多可以外界的工具,非常方便开发者的二次开发,同时又能够兼顾到性能。
本文介绍了高性能GPU分子动力学模拟软件CudaSPONGE的Python API接口,通过官方开发的prips插件,使得我们可以在Python框架下很方便的开发一些分子动力学模拟的Force Wrapper,例如Meta Dynamics中就有很多可以外界的工具,非常方便开发者的二次开发,同时又能够兼顾到性能。
CudaSPONGE高性能GPU分子模拟
摘要: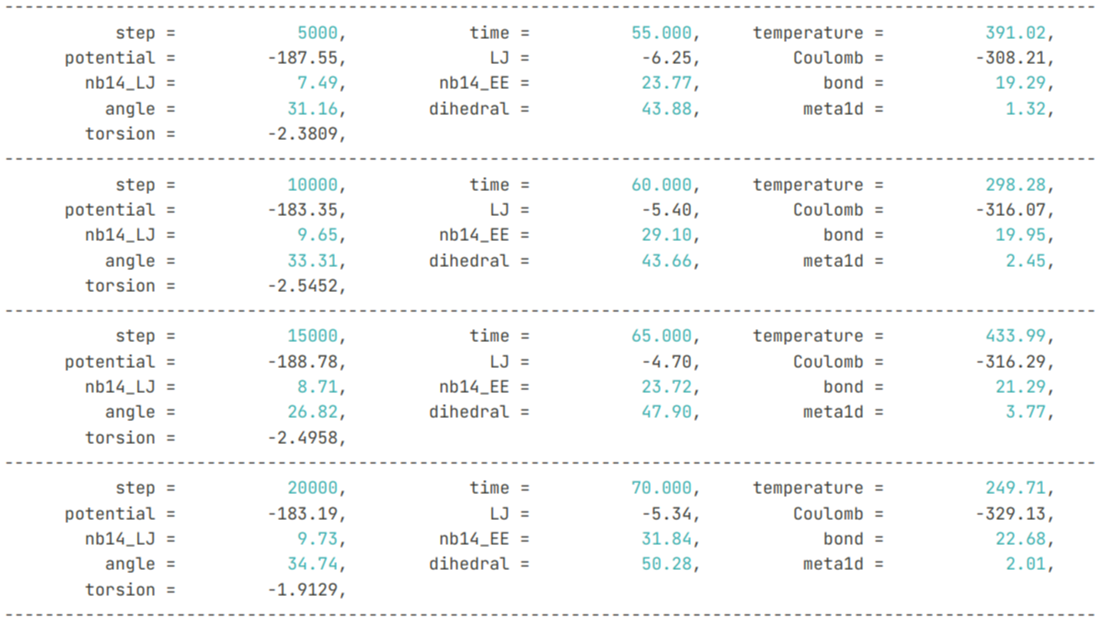 本文简单介绍了一下CudaSPONGE高性能分子动力学模拟软件,其基于原生的CUDA C开发,具有极高的模拟效率。结合前处理工具Xponge用于生成和处理输入文件,可以很好的兼容目前常用的很多力场形式。
本文简单介绍了一下CudaSPONGE高性能分子动力学模拟软件,其基于原生的CUDA C开发,具有极高的模拟效率。结合前处理工具Xponge用于生成和处理输入文件,可以很好的兼容目前常用的很多力场形式。
 本文简单介绍了一下CudaSPONGE高性能分子动力学模拟软件,其基于原生的CUDA C开发,具有极高的模拟效率。结合前处理工具Xponge用于生成和处理输入文件,可以很好的兼容目前常用的很多力场形式。
本文简单介绍了一下CudaSPONGE高性能分子动力学模拟软件,其基于原生的CUDA C开发,具有极高的模拟效率。结合前处理工具Xponge用于生成和处理输入文件,可以很好的兼容目前常用的很多力场形式。
PySAGES实记
摘要: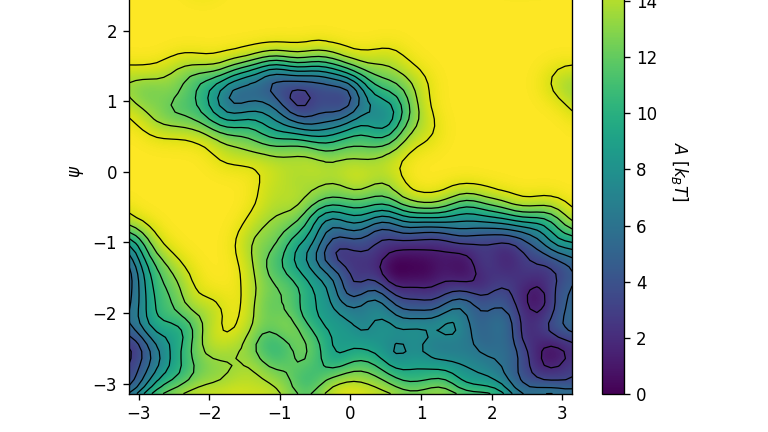 本文主要介绍了增强采样外接软件PySAGES的基本安装和使用方法,重点是安装过程中没有写清楚的一些环境依赖和可能出现的问题介绍,以及相应的解决方案。并简单的梳理了一下PySAGES软件的工作流机制,其能够做到Zero Copy,并使得Enhanced Sampling不再成为很多模拟的Bottleneck,这是一个相当出色的结果。
本文主要介绍了增强采样外接软件PySAGES的基本安装和使用方法,重点是安装过程中没有写清楚的一些环境依赖和可能出现的问题介绍,以及相应的解决方案。并简单的梳理了一下PySAGES软件的工作流机制,其能够做到Zero Copy,并使得Enhanced Sampling不再成为很多模拟的Bottleneck,这是一个相当出色的结果。
 本文主要介绍了增强采样外接软件PySAGES的基本安装和使用方法,重点是安装过程中没有写清楚的一些环境依赖和可能出现的问题介绍,以及相应的解决方案。并简单的梳理了一下PySAGES软件的工作流机制,其能够做到Zero Copy,并使得Enhanced Sampling不再成为很多模拟的Bottleneck,这是一个相当出色的结果。
本文主要介绍了增强采样外接软件PySAGES的基本安装和使用方法,重点是安装过程中没有写清楚的一些环境依赖和可能出现的问题介绍,以及相应的解决方案。并简单的梳理了一下PySAGES软件的工作流机制,其能够做到Zero Copy,并使得Enhanced Sampling不再成为很多模拟的Bottleneck,这是一个相当出色的结果。
OpenMM的安装与使用
摘要: 本文介绍了AlphaFold2中所使用到的开源分子动力学模拟软件OpenMM的安装和基础使用方法,其中包含了pdbfixer蛋白质构象文件修复工具的介绍和一个真空蛋白体系的能量极小化示例,并且提供了一些有可能在OpenMM的安装和使用过程中遇到的问题和解决方法。
本文介绍了AlphaFold2中所使用到的开源分子动力学模拟软件OpenMM的安装和基础使用方法,其中包含了pdbfixer蛋白质构象文件修复工具的介绍和一个真空蛋白体系的能量极小化示例,并且提供了一些有可能在OpenMM的安装和使用过程中遇到的问题和解决方法。
 本文介绍了AlphaFold2中所使用到的开源分子动力学模拟软件OpenMM的安装和基础使用方法,其中包含了pdbfixer蛋白质构象文件修复工具的介绍和一个真空蛋白体系的能量极小化示例,并且提供了一些有可能在OpenMM的安装和使用过程中遇到的问题和解决方法。
本文介绍了AlphaFold2中所使用到的开源分子动力学模拟软件OpenMM的安装和基础使用方法,其中包含了pdbfixer蛋白质构象文件修复工具的介绍和一个真空蛋白体系的能量极小化示例,并且提供了一些有可能在OpenMM的安装和使用过程中遇到的问题和解决方法。
MindSponge分子动力学模拟——增强采样(2024.11)
摘要: 本文介绍了在MindSponge中进行分子动力学模拟以及增强采样的实现方法。通过使用MetaDynamics增强采样算法,我们可以将分子模拟的采样子空间,从某个能量极小值区域,扩大到尽可能大的采样子空间。
本文介绍了在MindSponge中进行分子动力学模拟以及增强采样的实现方法。通过使用MetaDynamics增强采样算法,我们可以将分子模拟的采样子空间,从某个能量极小值区域,扩大到尽可能大的采样子空间。
 本文介绍了在MindSponge中进行分子动力学模拟以及增强采样的实现方法。通过使用MetaDynamics增强采样算法,我们可以将分子模拟的采样子空间,从某个能量极小值区域,扩大到尽可能大的采样子空间。
本文介绍了在MindSponge中进行分子动力学模拟以及增强采样的实现方法。通过使用MetaDynamics增强采样算法,我们可以将分子模拟的采样子空间,从某个能量极小值区域,扩大到尽可能大的采样子空间。
PME算法简单Python实现
摘要: 基于前面几篇博客关于PME算法的理论推导,本文给出了一个简单版本的Python代码实现,并且对比了PME算法相比于实空间迭代算法的优越性。从结果上来看,一维的静电势能计算中,PME单步得到的计算结果非常接近于实空间迭代1万个Box的近似结果。
基于前面几篇博客关于PME算法的理论推导,本文给出了一个简单版本的Python代码实现,并且对比了PME算法相比于实空间迭代算法的优越性。从结果上来看,一维的静电势能计算中,PME单步得到的计算结果非常接近于实空间迭代1万个Box的近似结果。
 基于前面几篇博客关于PME算法的理论推导,本文给出了一个简单版本的Python代码实现,并且对比了PME算法相比于实空间迭代算法的优越性。从结果上来看,一维的静电势能计算中,PME单步得到的计算结果非常接近于实空间迭代1万个Box的近似结果。
基于前面几篇博客关于PME算法的理论推导,本文给出了一个简单版本的Python代码实现,并且对比了PME算法相比于实空间迭代算法的优越性。从结果上来看,一维的静电势能计算中,PME单步得到的计算结果非常接近于实空间迭代1万个Box的近似结果。
格点拉格朗日插值与PME算法
摘要: 本文介绍了使用基于格点拉格朗日插值法的Particle Mesh Ewald算法,降低分子力场中的静电势能项计算复杂度的基本原理。静电势能的计算在Ewald求和框架下被拆分成了远程相互作用项和短程相互作用项,其中短程相互作用项关于实空间的点电荷间距快速收敛,而远程相互作用项在倒易空间慢速收敛。因此在远程相互作用的计算中,可以使用插值法降低单个倒易格点的计算复杂度,从而使得整体的远程相互作用项计算也能够快速收敛。
本文介绍了使用基于格点拉格朗日插值法的Particle Mesh Ewald算法,降低分子力场中的静电势能项计算复杂度的基本原理。静电势能的计算在Ewald求和框架下被拆分成了远程相互作用项和短程相互作用项,其中短程相互作用项关于实空间的点电荷间距快速收敛,而远程相互作用项在倒易空间慢速收敛。因此在远程相互作用的计算中,可以使用插值法降低单个倒易格点的计算复杂度,从而使得整体的远程相互作用项计算也能够快速收敛。
 本文介绍了使用基于格点拉格朗日插值法的Particle Mesh Ewald算法,降低分子力场中的静电势能项计算复杂度的基本原理。静电势能的计算在Ewald求和框架下被拆分成了远程相互作用项和短程相互作用项,其中短程相互作用项关于实空间的点电荷间距快速收敛,而远程相互作用项在倒易空间慢速收敛。因此在远程相互作用的计算中,可以使用插值法降低单个倒易格点的计算复杂度,从而使得整体的远程相互作用项计算也能够快速收敛。
本文介绍了使用基于格点拉格朗日插值法的Particle Mesh Ewald算法,降低分子力场中的静电势能项计算复杂度的基本原理。静电势能的计算在Ewald求和框架下被拆分成了远程相互作用项和短程相互作用项,其中短程相互作用项关于实空间的点电荷间距快速收敛,而远程相互作用项在倒易空间慢速收敛。因此在远程相互作用的计算中,可以使用插值法降低单个倒易格点的计算复杂度,从而使得整体的远程相互作用项计算也能够快速收敛。
Ewald求和在分子静电势能计算中的应用
摘要: 本文介绍了Ewald求和计算方法在周期性边界条件下计算静电势能的方法。周期性的静电势函数并不是一个空间收敛的函数,通过Ewald求和可以将静电势切分为短程相互作用和长程相互作用,两项分别在实空间和倒易空间(或称傅里叶空间、k空间等)收敛。然后就可以进一步进行截断,用更少的代价获得更高精度的电势能计算结果。
本文介绍了Ewald求和计算方法在周期性边界条件下计算静电势能的方法。周期性的静电势函数并不是一个空间收敛的函数,通过Ewald求和可以将静电势切分为短程相互作用和长程相互作用,两项分别在实空间和倒易空间(或称傅里叶空间、k空间等)收敛。然后就可以进一步进行截断,用更少的代价获得更高精度的电势能计算结果。
 本文介绍了Ewald求和计算方法在周期性边界条件下计算静电势能的方法。周期性的静电势函数并不是一个空间收敛的函数,通过Ewald求和可以将静电势切分为短程相互作用和长程相互作用,两项分别在实空间和倒易空间(或称傅里叶空间、k空间等)收敛。然后就可以进一步进行截断,用更少的代价获得更高精度的电势能计算结果。
本文介绍了Ewald求和计算方法在周期性边界条件下计算静电势能的方法。周期性的静电势函数并不是一个空间收敛的函数,通过Ewald求和可以将静电势切分为短程相互作用和长程相互作用,两项分别在实空间和倒易空间(或称傅里叶空间、k空间等)收敛。然后就可以进一步进行截断,用更少的代价获得更高精度的电势能计算结果。
使用CyFES对配体运动轨迹进行数据透视
摘要: 分子动力学模拟是一个以时间换空间的方法,那么在时间尺度上留下轨迹之后,如何把轨迹做一个静态的展现,正是数据透视所解决的问题。CyFES是一个开源的、基于GPU硬件加速的数据透视高性能计算工具,我们通过一个蛋白-配体相互作用的运动轨迹的示例,演示一下CyFES的基本使用方法。
分子动力学模拟是一个以时间换空间的方法,那么在时间尺度上留下轨迹之后,如何把轨迹做一个静态的展现,正是数据透视所解决的问题。CyFES是一个开源的、基于GPU硬件加速的数据透视高性能计算工具,我们通过一个蛋白-配体相互作用的运动轨迹的示例,演示一下CyFES的基本使用方法。
 分子动力学模拟是一个以时间换空间的方法,那么在时间尺度上留下轨迹之后,如何把轨迹做一个静态的展现,正是数据透视所解决的问题。CyFES是一个开源的、基于GPU硬件加速的数据透视高性能计算工具,我们通过一个蛋白-配体相互作用的运动轨迹的示例,演示一下CyFES的基本使用方法。
分子动力学模拟是一个以时间换空间的方法,那么在时间尺度上留下轨迹之后,如何把轨迹做一个静态的展现,正是数据透视所解决的问题。CyFES是一个开源的、基于GPU硬件加速的数据透视高性能计算工具,我们通过一个蛋白-配体相互作用的运动轨迹的示例,演示一下CyFES的基本使用方法。
Windows版本免费PyMol的安装
摘要: 接上一篇介绍Linux下安装和使用免费版本的PyMol之后,这里再介绍一下Windows系统下的安装方法。同时在本文中列举了一些在PyMol中有可能使用到的脚本指令,例如设置球体模型的大小、设置表面模型的透明度、平移分子和批量执行脚本等操作。
接上一篇介绍Linux下安装和使用免费版本的PyMol之后,这里再介绍一下Windows系统下的安装方法。同时在本文中列举了一些在PyMol中有可能使用到的脚本指令,例如设置球体模型的大小、设置表面模型的透明度、平移分子和批量执行脚本等操作。
 接上一篇介绍Linux下安装和使用免费版本的PyMol之后,这里再介绍一下Windows系统下的安装方法。同时在本文中列举了一些在PyMol中有可能使用到的脚本指令,例如设置球体模型的大小、设置表面模型的透明度、平移分子和批量执行脚本等操作。
接上一篇介绍Linux下安装和使用免费版本的PyMol之后,这里再介绍一下Windows系统下的安装方法。同时在本文中列举了一些在PyMol中有可能使用到的脚本指令,例如设置球体模型的大小、设置表面模型的透明度、平移分子和批量执行脚本等操作。
MindSponge分子动力学模拟——多路径分子模拟(2024.05)
摘要: 随着硬件算力的发展,以及AI技术的日益增进,我们不仅可以借助深度学习框架来加速分子动力学模拟,以及降低分子模拟开发的门槛。还可以实现高通量模拟,使得用最小的开销并行的运行多个分子模拟成为可能。
随着硬件算力的发展,以及AI技术的日益增进,我们不仅可以借助深度学习框架来加速分子动力学模拟,以及降低分子模拟开发的门槛。还可以实现高通量模拟,使得用最小的开销并行的运行多个分子模拟成为可能。
 随着硬件算力的发展,以及AI技术的日益增进,我们不仅可以借助深度学习框架来加速分子动力学模拟,以及降低分子模拟开发的门槛。还可以实现高通量模拟,使得用最小的开销并行的运行多个分子模拟成为可能。
随着硬件算力的发展,以及AI技术的日益增进,我们不仅可以借助深度学习框架来加速分子动力学模拟,以及降低分子模拟开发的门槛。还可以实现高通量模拟,使得用最小的开销并行的运行多个分子模拟成为可能。
MindSponge分子动力学模拟——体系控制(2024.05)
摘要: 本文是一个比较泛的分子体系控制器实现方案,因为MindSponge分子动力学模拟框架基于Python编程语言和MindSpore框架开发,因此在高度定制化的控制器实现上有先天的优势。我们可以在MindSponge中基于力对体系进行控制、基于坐标对体系进行控制,还能基于反应坐标对体系进行控制。
本文是一个比较泛的分子体系控制器实现方案,因为MindSponge分子动力学模拟框架基于Python编程语言和MindSpore框架开发,因此在高度定制化的控制器实现上有先天的优势。我们可以在MindSponge中基于力对体系进行控制、基于坐标对体系进行控制,还能基于反应坐标对体系进行控制。
 本文是一个比较泛的分子体系控制器实现方案,因为MindSponge分子动力学模拟框架基于Python编程语言和MindSpore框架开发,因此在高度定制化的控制器实现上有先天的优势。我们可以在MindSponge中基于力对体系进行控制、基于坐标对体系进行控制,还能基于反应坐标对体系进行控制。
本文是一个比较泛的分子体系控制器实现方案,因为MindSponge分子动力学模拟框架基于Python编程语言和MindSpore框架开发,因此在高度定制化的控制器实现上有先天的优势。我们可以在MindSponge中基于力对体系进行控制、基于坐标对体系进行控制,还能基于反应坐标对体系进行控制。
MindSponge分子动力学模拟——自定义控制器(2024.05)
摘要: 本文介绍了在MindSponge分子动力学模拟框架先实现自定义Controller控制器的方法,通过调控体系中的原子坐标和原子速度等,来控制系综的参量。MindSponge分子模拟框架基于MindSpore深度学习框架开发而成,对于开发者尤其是深度学习开发者来说,非常的友好。
本文介绍了在MindSponge分子动力学模拟框架先实现自定义Controller控制器的方法,通过调控体系中的原子坐标和原子速度等,来控制系综的参量。MindSponge分子模拟框架基于MindSpore深度学习框架开发而成,对于开发者尤其是深度学习开发者来说,非常的友好。
 本文介绍了在MindSponge分子动力学模拟框架先实现自定义Controller控制器的方法,通过调控体系中的原子坐标和原子速度等,来控制系综的参量。MindSponge分子模拟框架基于MindSpore深度学习框架开发而成,对于开发者尤其是深度学习开发者来说,非常的友好。
本文介绍了在MindSponge分子动力学模拟框架先实现自定义Controller控制器的方法,通过调控体系中的原子坐标和原子速度等,来控制系综的参量。MindSponge分子模拟框架基于MindSpore深度学习框架开发而成,对于开发者尤其是深度学习开发者来说,非常的友好。
统计力学中的概率论基础(二)
摘要: 可以理解的是,概率密度函数,一般情况下都是连续的。但是对于采样或者随机试验来说,其实都是离散采样。大数定理通过取一个极限,将概率密度函数跟试验联系了起来。这篇文章主要介绍的是常用的几个概率密度函数的期望值和方差的计算,以及大数定理的基本概念。
可以理解的是,概率密度函数,一般情况下都是连续的。但是对于采样或者随机试验来说,其实都是离散采样。大数定理通过取一个极限,将概率密度函数跟试验联系了起来。这篇文章主要介绍的是常用的几个概率密度函数的期望值和方差的计算,以及大数定理的基本概念。
 可以理解的是,概率密度函数,一般情况下都是连续的。但是对于采样或者随机试验来说,其实都是离散采样。大数定理通过取一个极限,将概率密度函数跟试验联系了起来。这篇文章主要介绍的是常用的几个概率密度函数的期望值和方差的计算,以及大数定理的基本概念。
可以理解的是,概率密度函数,一般情况下都是连续的。但是对于采样或者随机试验来说,其实都是离散采样。大数定理通过取一个极限,将概率密度函数跟试验联系了起来。这篇文章主要介绍的是常用的几个概率密度函数的期望值和方差的计算,以及大数定理的基本概念。
统计力学中的概率论基础(一)
摘要: 本文的主要内容是一些统计力学中的基础的概率论知识,如密度函数、分布函数和贝叶斯定理的一些基本概念,主要作为一个简单的知识内容记录和分享,后续还有更多的同系列文章。
本文的主要内容是一些统计力学中的基础的概率论知识,如密度函数、分布函数和贝叶斯定理的一些基本概念,主要作为一个简单的知识内容记录和分享,后续还有更多的同系列文章。
 本文的主要内容是一些统计力学中的基础的概率论知识,如密度函数、分布函数和贝叶斯定理的一些基本概念,主要作为一个简单的知识内容记录和分享,后续还有更多的同系列文章。
本文的主要内容是一些统计力学中的基础的概率论知识,如密度函数、分布函数和贝叶斯定理的一些基本概念,主要作为一个简单的知识内容记录和分享,后续还有更多的同系列文章。
Plumed分子模拟后分析
摘要: Plumed是一个强大的分子模拟数据处理工具,可以在模拟的过程中逐步分析,也可以保存模拟的轨迹做后分析。本文紧接前面的“增强采样软件PLUMED的安装与使用”文章,还有“直方图与核密度估计”文章。介绍了如何使用Plumed后分析工具,对输出的反应坐标的轨迹进行核密度估计。
Plumed是一个强大的分子模拟数据处理工具,可以在模拟的过程中逐步分析,也可以保存模拟的轨迹做后分析。本文紧接前面的“增强采样软件PLUMED的安装与使用”文章,还有“直方图与核密度估计”文章。介绍了如何使用Plumed后分析工具,对输出的反应坐标的轨迹进行核密度估计。
 Plumed是一个强大的分子模拟数据处理工具,可以在模拟的过程中逐步分析,也可以保存模拟的轨迹做后分析。本文紧接前面的“增强采样软件PLUMED的安装与使用”文章,还有“直方图与核密度估计”文章。介绍了如何使用Plumed后分析工具,对输出的反应坐标的轨迹进行核密度估计。
Plumed是一个强大的分子模拟数据处理工具,可以在模拟的过程中逐步分析,也可以保存模拟的轨迹做后分析。本文紧接前面的“增强采样软件PLUMED的安装与使用”文章,还有“直方图与核密度估计”文章。介绍了如何使用Plumed后分析工具,对输出的反应坐标的轨迹进行核密度估计。
直方图与核密度估计
摘要: 核密度估计(KDE)方法,相当于用多个波包的组合形式来近似一个真实的概率密度,以获得一个连续可微分的概率密度函数。本文通过一些简单的概率分布的示例,演示了一下KDE的使用方法。其实KDE的思想在很多领域都会以不同的形式出现,是一个比较基础的概率分布近似手段。
核密度估计(KDE)方法,相当于用多个波包的组合形式来近似一个真实的概率密度,以获得一个连续可微分的概率密度函数。本文通过一些简单的概率分布的示例,演示了一下KDE的使用方法。其实KDE的思想在很多领域都会以不同的形式出现,是一个比较基础的概率分布近似手段。
 核密度估计(KDE)方法,相当于用多个波包的组合形式来近似一个真实的概率密度,以获得一个连续可微分的概率密度函数。本文通过一些简单的概率分布的示例,演示了一下KDE的使用方法。其实KDE的思想在很多领域都会以不同的形式出现,是一个比较基础的概率分布近似手段。
核密度估计(KDE)方法,相当于用多个波包的组合形式来近似一个真实的概率密度,以获得一个连续可微分的概率密度函数。本文通过一些简单的概率分布的示例,演示了一下KDE的使用方法。其实KDE的思想在很多领域都会以不同的形式出现,是一个比较基础的概率分布近似手段。






