【python爬虫课程设计】大数据分析——土壤、菌类、环境对乔木植物的生存影响数据的预测模型
选题方向:2.大数据分析
一、选题背景介绍
新华社北京7月18日电 全国生态环境保护大会17日至18日在北京召开。今后5年是美丽中国建设的重要时期,要深入贯彻新时代中国特色社会主义生态文明思想,坚持以人民为中心,牢固树立和践行绿水青山就是金山银山的理念,把建设美丽中国摆在强国建设、民族复兴的突出位置,推动城乡人居环境明显改善、美丽中国建设取得显著成效,以高品质生态环境支撑高质量发展,加快推进人与自然和谐共生的现代化。
***在党的二十大报告中强调,“立足我国能源资源禀赋,坚持先立后破,有计划分步骤实施碳达峰行动。”“完善能源消耗总量和强度调控,重点控制化石能源消费,逐步转向碳排放总量和强度‘双控’制度。”
在大数据时代,我们可以利用海量的数据信息进行深度分析和挖掘,从而更好地了解自然环境的状况和变化趋势。通过对环境数据的收集、整合和分析,我们可以预测环境问题的发生和发展趋势,为政府和企业提供更加精准的环境管理决策支持。大数据分析还可以促进公众参与和监督。通过公开环境数据和信息,可以增强公众对环境问题的关注和参与度,形成政府、企业和公众共同参与的环境治理格局。
我们应该深入学习领会重要讲话精神,认真贯彻落实生态文明建设的各项决策部署,积极推动大数据技术在生态环境保护领域的应用和发展,为实现人与自然和谐共生的现代化目标贡献力量。
二、选题意义
选择乔木植物作为大数据分析对象与国家碳中和的战略意义密切相关:
1. 碳吸存和减排:乔木植物通过光合作用吸收大量的二氧化碳,并将其固定在植物组织中。将乔木植物作为大数据分析对象,可以帮助我们更好地了解它们在吸收和储存碳方面的能力。这有助于国家实现碳中和目标,减少温室气体排放,应对气候变化。
2. 林业管理和可持续发展:通过对乔木植物的大数据分析,可以了解森林资源的分布、生长情况、病虫害等信息。这有助于制定科学合理的林业管理策略,推动森林的可持续利用和保护。同时,可持续林业发展也可以为国家提供木材、纤维和其他森林产品,促进经济发展。
3. 生态系统恢复和生物多样性保护:乔木植物是生态系统中的重要组成部分,对维护生态平衡和保护生物多样性至关重要。通过对乔木植物进行大数据分析,可以深入了解不同物种的分布、生态特征和相互关系。这有助于科学规划和实施生态系统的恢复计划,保护和恢复受损的生态系统,并维护关键物种的栖息地。
4. 水资源管理:乔木植物在水循环中发挥着重要作用,通过蒸腾作用调节水分蒸发和降水分布。通过对乔木植物的大数据分析,可以了解它们对水资源的利用和影响,帮助科学合理地管理和保护水资源,以应对干旱、水源污染等问题。
5. 环境教育和公众意识:乔木植物是生态系统和环境保护的重要教育资源。通过对乔木植物的大数据分析,可以为公众提供有关森林生态系统、气候变化和生物多样性保护等方面的科学知识。这有助于提高公众对环境保护的认识和意识,促进可持续发展理念的传播和落实。
选择乔木植物作为大数据分析对象可以为国家碳中和的战略提供重要支持。通过深入了解乔木植物的生态特征、碳吸存能力和生态系统服务功能,可以制定科学有效的政策和措施,推动碳减排、可持续林业发展、生态系统保护和环境教育等方面的工作。
三、数据集简介
本数据源包含:Plot、Subplot、Species、Light_ISF、Light_Cat、Core、Soil、Adult、Sterile、Conspecific、Myco、SoilMyco、PlantDate、AMF、EMF、Phenolics、Lignin、NSC、Census、Time、Event、Harvest等24个指标,以及红栎,白栎,枫树,樱桃木四个树种。其中观察树种在不同情况下,哪个树种在哪个环境下生长的好,并解释为什么出现这种情况的原因。
该研究在数据集测量数据采自北半球地中海沿岸地区、黑海沿岸地区、国家(纬度30至40度)进行。夏季炎热干燥,冬季温和多雨,是13种气候类型中唯一一种雨热不同期的气候类型。
夏季在副热带高压控制下,气流下沉,气候炎热干燥少雨,云量稀少,阳光充足。冬季受西风带控制,锋面气旋频繁活动,气候温和,最冷月气温在4-10℃之间,降水量丰沛。
全年降水量300-1000毫米,冬季半年约占60%-70%,夏季半年只有30%-40%,冬季降水量多于夏季。该数据集与4种乔木植物有关,此数据集已预先清理,没有任何问题。我们只需要一个模型来预测谁在相同情况下生存得最多。
数据采集时间在2018年5月15日~2018年5月15日,对于每个数据的每个物种,均采集于北半球地区域1m高度的相同高度。在2018年5月(干旱季节)中,从选定的树种中采集了叶片和根际样品。为了采集叶片样品,我们使用了一个长距离修剪机从树冠上收集了暴露于阳光下的树枝所选物种的顶部。通过对叶片营养成分和根际理化性质、根际微生物量以及根际P组分和根际酸性磷酸酶活性进行了测定。并分析树木枝叶各成分含量(丛枝菌根真菌(AMF)和外生菌根真菌(EMF)、Phenolics酚、Lignin木质素、NSC(非结构碳水化合物))Census(测量数)
数据验证使用中国生物志库(验证该植物是否真实存在,且在中国地区出现频繁,设立同科对照组,对比数据,得出准确的项目结果):
https://species.sciencereading.cn/biology/v/biologicalIndex/122.html
使用数据集:Cleaned_Tree_Dataset.csv
四、大数据前期准备
4.1数据集清洗
对数据进行数据清洗,数据清洗在数据分析和建模过程中起着重要的作用。用来提高数据的质量和准确性,确保数据的可靠性和可用性:
1 import pandas as pd 2 3 # 导入数据集 4 data = pd.read_csv('Cleaned_Tree_Dataset.csv') 5 # 数据清洗:去除缺失值或异常值 6 data = data.dropna() # 去除包含缺失值的行 7 # 其他数据清洗操作... 8 # 获取所有树种的唯一值 9 species = data['Species'].unique() 10 # 计算每个树种的数据指标平均值 11 averages = data.groupby(['Species'])[['AMF', 'EMF', 'Phenolics', 'Lignin', 'NSC']].mean() 12 # 输出数据对比表格 13 comparison_table = pd.DataFrame(averages.loc[species], columns=['AMF', 'EMF', 'Phenolics', 'Lignin', 'NSC']) 14 print(comparison_table)
实验结果:
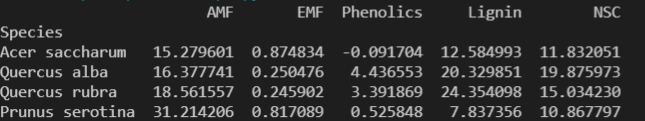
输出各个树种的元素值以达到基本数据集使用演奏求,提高了数据分析和建模的准确性和可靠性,从而支持更可靠的决策和洞察力的发现,并得出总结。
4.2数据集划分
对数据集划分训练集和测试集,做前期数据分析的准备:
1 import pandas as pd 2 from sklearn.model_selection import train_test_split 3 4 # 导入数据集 5 data = pd.read_csv('Cleaned_Tree_Dataset.csv') 6 # 划分特征和目标变量 7 X = data.drop('Species', axis=1) # 特征 8 y = data['Species'] # 目标变量 9 # 数据集划分 10 X_train, X_test, y_train, y_test = train_test_split(X, y, test_size=0.2, random_state=42) 11 # 获取所有树种的唯一值 12 species = data['Species'].unique() 13 # 计算训练集每个树种的数据指标平均值 14 train_averages = X_train.groupby(y_train)[['AMF', 'EMF', 'Phenolics', 'Lignin', 'NSC']].mean() 15 # 输出训练集数据对比表格 16 train_comparison_table = pd.DataFrame(train_averages.loc[species], columns=['AMF', 'EMF', 'Phenolics', 'Lignin', 'NSC']) 17 print("训练集数据对比表格:") 18 print(train_comparison_table) 19 # 计算测试集每个树种的数据指标平均值 20 test_averages = X_test.groupby(y_test)[['AMF', 'EMF', 'Phenolics', 'Lignin', 'NSC']].mean() 21 # 输出测试集数据对比表格 22 test_comparison_table = pd.DataFrame(test_averages.loc[species], columns=['AMF', 'EMF', 'Phenolics', 'Lignin', 'NSC']) 23 print("测试集数据对比表格:") 24 print(test_comparison_table)
实验结果:
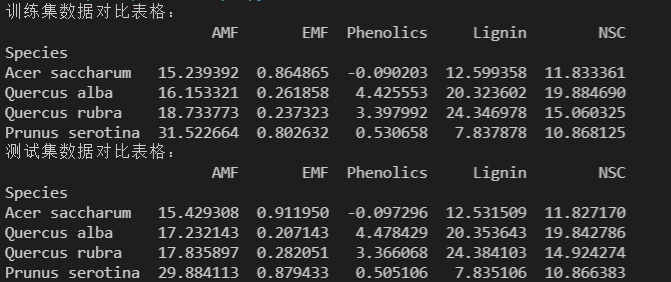
对于数据集有了明确的拆分,输出两个数据集的对比,实现了对后期结果的可验证性和对比性,为接下来的实验分析打下坚实基础。
五、大数据分析实验
5.1数据集各树种分布合理性
观察数据集乔木植物的分布占比,导入数据集,可视化数据绘制饼图,分析数据集中的种类总数占比:
1 from textwrap import wrap 2 import pandas as pd 3 import matplotlib.pyplot as plt 4 import numpy as np 5 6 plt.rcParams['font.sans-serif'] = ['SimHei'] 7 # 导入数据集 8 df = pd.read_csv('Cleaned_Tree_Dataset.csv') 9 # 创建图形 10 fig = plt.figure() 11 # 计算每种类型的数量 12 type_counts = df['Species'].value_counts() 13 # 计算总数量 14 total = type_counts.sum() 15 # 计算占比 16 percentages = (type_counts / total) * 100 17 # 绘制饼图 18 plt.pie(percentages, labels=percentages.index, autopct='%1.1f%%') 19 plt.title('种类总数占比图') 20 plt.show()
实验结果:
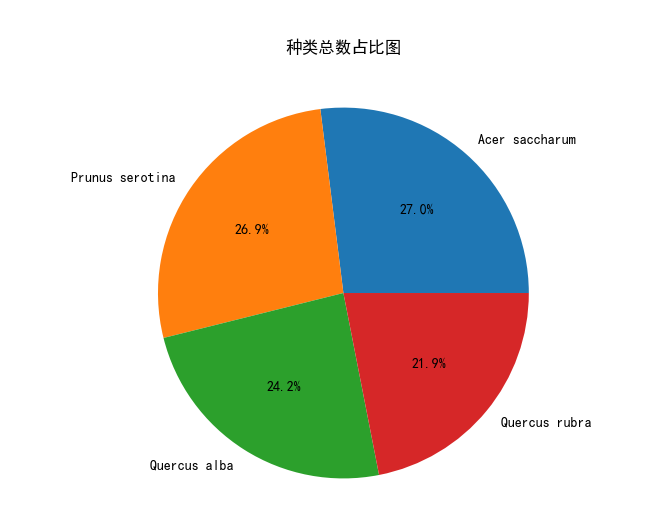
数据图表分析:
- 确定占比的合理性标准:在评估数据集的占比合理性之前,需要明确什么样的占比被认为是合理的。
- 分析占比分布:对数据集中的各个关键指标进行占比分析,以了解它们之间的分布情况。
- 确定占比的合理范围:基于业务或研究需求,确定每个关键指标的占比合理范围。
- 比较和分析:将数据集中的实际占比与确定的合理范围进行比较和分析。
- 数据集的占比关系合理且满足研究需求,我认为该数据集在占比方面是可行的。Quercus alba(白橡)、Quercus rubra(红橡)、Acer saccharum(枫树)和Prunus serotina(樱桃树)确实是四种不同的树种,它们在自然界中广泛分布并具有生态功能。数据集中,展示的四个树种在其占比合理,可以使用该数据集进行继续的讨论研究。
5.2数据集树种多样性证明
观察数据集同种、异种、无菌的分布占比,绘制可视化数据绘制饼图,分析数据集中的种类总数占比:
1 from textwrap import wrap 2 import pandas as pd 3 import matplotlib.pyplot as plt 4 import numpy as np 5 6 plt.rcParams['font.sans-serif'] = ['SimHei'] 7 # 导入数据集 8 df = pd.read_csv('Cleaned_Tree_Dataset.csv') 9 # 创建图形 10 fig = plt.figure() 11 # 计算每种类型的数量 12 type_counts = df['Conspecific'].value_counts() 13 # 计算总数量 14 total = type_counts.sum() 15 # 计算占比 16 percentages = (type_counts / total) * 100 17 # 绘制饼图 18 plt.pie(percentages, labels=percentages.index, autopct='%1.1f%%') 19 plt.title('数据同种的总数占比图') 20 plt.show()
实验结果:
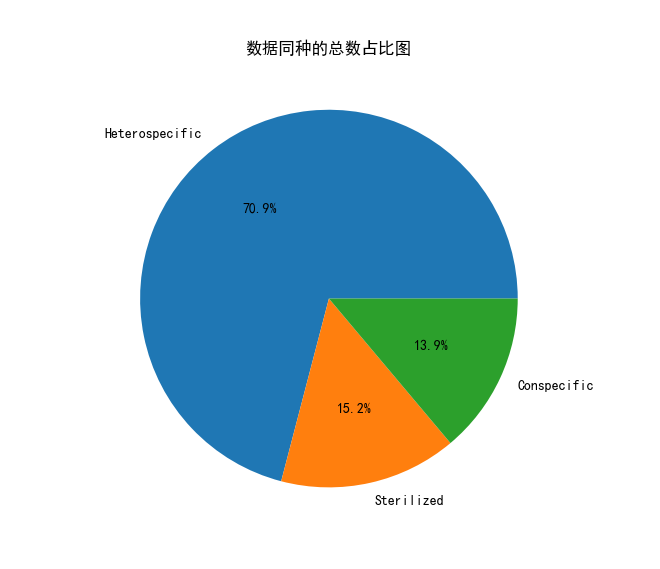
数据分布计算:计算每种数据类型的数量,直观看到计算它们在数据集中的占比。
数据可视化:使用可视化工具来绘制饼图,以显示每种数据类型的分布占比。
数据分析:通过观察饼图,清楚地看到每种数据类型的占比。如果发现我呢提,可以提前根据要求,清洗数据集,脏数据,并根据需要调整数据收集策略或分析方法。
树种的Sterilized、Heterospecific和Conspecific是描述不同植物间杂交方式的术语。在图中,这些数据被表示为饼状图中的百分比。根据饼状图,我们可以得知:
- Sterilized(纯合):占总数的70.9%。
- Heterospecific(杂合):占总数的13.9%。
- Conspecific(同种):占总数的15.2%。
树种的种类繁多。在植物分类学中,我们通常根据一些特定的标准将树种进行分类。按照热量因子,树种可以分为热带树种、亚热带树种、温带树种和寒带亚寒带树种;按照水分因子,树种可以划分为耐旱树种和耐湿树种;光照因子则可以将树种分为阳性树、中性数、阴性树,每类中又可分数级。另外,我们还可以根据树种的枝干特点进行分类,比如乔木,它的树体高大,具有明显主干,高度在10米以上。
在中国,由于地理环境的差异,形成了生物种类繁多、植被类型丰富的情况。湖南省的树木资源就非常丰富,有114科431属1868种及变种。其中松树就是种类非常繁多的一种,不仅分布广泛,而且形态各异。
总的来说,不同的分类方式可以让我们对树种有更深入的了解,同时也反映出生物多样性的特点。
5.3各个树种生长影响因子展示
导入数据集,分类提取四种树种的(Acer saccharum、、Quercus rubra、Quercus alba、Prunus serotina)的,取得各个AMF、EMF、Phenolics、Lignin、NSC数据,分析每一个树种在实验结束后取得各个数据指标的平均值,绘制表格:
1 import pandas as pd 2 import matplotlib.pyplot as plt 3 4 # 导入数据集 5 data = pd.read_csv('Cleaned_Tree_Dataset.csv') 6 # 指定要提取的树种 7 species_list = ['Acer saccharum', 'Quercus rubra', 'Quercus alba', 'Prunus serotina'] 8 subset = data[data['Species'].isin(species_list)] 9 # 提取所需的数据列 10 columns = ['Species', 'AMF', 'EMF', 'Phenolics', 'Lignin', 'NSC'] 11 subset = subset[columns] 12 # 计算每个数据指标的平均值 13 averages = subset.groupby('Species').mean() 14 # 创建表格 15 fig, ax = plt.subplots() 16 ax.axis('off') 17 table = ax.table(cellText=averages.values, colLabels=averages.columns, rowLabels=averages.index, loc='center') 18 # 设置表格样式 19 table.auto_set_font_size(False) 20 table.set_fontsize(12) 21 table.scale(1.2, 1.2) 22 # 显示表格 23 plt.show()
实验结果:
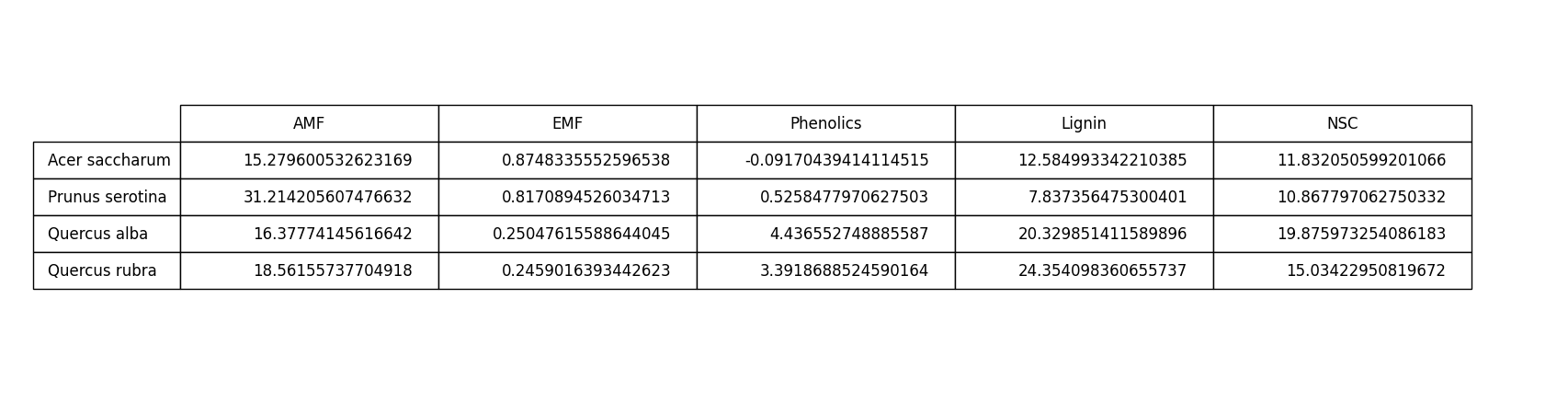
表格是数据展示的一种常用方式,具有以下优点:
- 清晰明了:表格以行和列的形式组织数据,可以直观地展示数据的分布和关系。
- 结构化:表格可以将复杂的数据以结构化的形式呈现,方便阅读和理解。
- 可比性:在表格中,同一类数据可以使用相同的格式和单位展示,使得数据之间的比较变得容易。
- 易于编辑和更新:表格可以轻松地编辑和更新,可以很方便地添加、删除或修改数据。
- 可结合其他工具:表格可以结合各种数据分析工具,进行数据分析和处理。
- 可定制化:可以根据需要定制表格的样式、颜色、字体等,使得数据展示更加美观和易读。
- 可共享:可以将表格分享给其他人,使得数据更加易于共享和使用。
5.4各个树种生长影响因子柱状图展示
分类提取Acer saccharum、、Quercus rubra、Quercus alba、Prunus serotina的,取得各个AMF、EMF、Phenolics、Lignin、NSC数据,分析每一个树种在实验结束后取得各个数据指标的平均值,绘制柱状图,并对比
说明各个树种所需要的微量元素和生长要求是不同的,需要对不同的树种分情况,处理:
1 import pandas as pd 2 import matplotlib.pyplot as plt 3 4 plt.rcParams['font.sans-serif'] = ['SimHei'] 5 # 导入数据集 6 data = pd.read_csv('Cleaned_Tree_Dataset.csv') 7 # 指定要提取的树种 8 species_list = ['Acer saccharum', 'Quercus rubra', 'Quercus alba', 'Prunus serotina'] 9 subset = data[data['Species'].isin(species_list)] 10 # 提取所需的数据列 11 columns = ['Species', 'AMF', 'EMF', 'Phenolics', 'Lignin', 'NSC'] 12 subset = subset[columns] 13 # 计算每个数据指标的平均值 14 averages = subset.groupby('Species').mean().reset_index() 15 # 绘制柱状图 16 x = range(len(averages)) 17 bar_width = 0.15 18 plt.bar(x, averages['AMF'], width=bar_width, label='AMF') 19 plt.bar([i + bar_width for i in x], averages['EMF'], width=bar_width, label='EMF') 20 plt.bar([i + 2 * bar_width for i in x], averages['Phenolics'], width=bar_width, label='Phenolics') 21 plt.bar([i + 3 * bar_width for i in x], averages['Lignin'], width=bar_width, label='Lignin') 22 plt.bar([i + 4 * bar_width for i in x], averages['NSC'], width=bar_width, label='NSC') 23 # 设置图表属性 24 plt.title('2018年5月-6月各树种数据指标的平均值对比图') 25 plt.xlabel('Species') 26 plt.ylabel('Average Value') 27 plt.xticks([i + 2 * bar_width for i in x], averages['Species']) 28 plt.legend() 29 # 显示图表 30 plt.show()
实验结果:
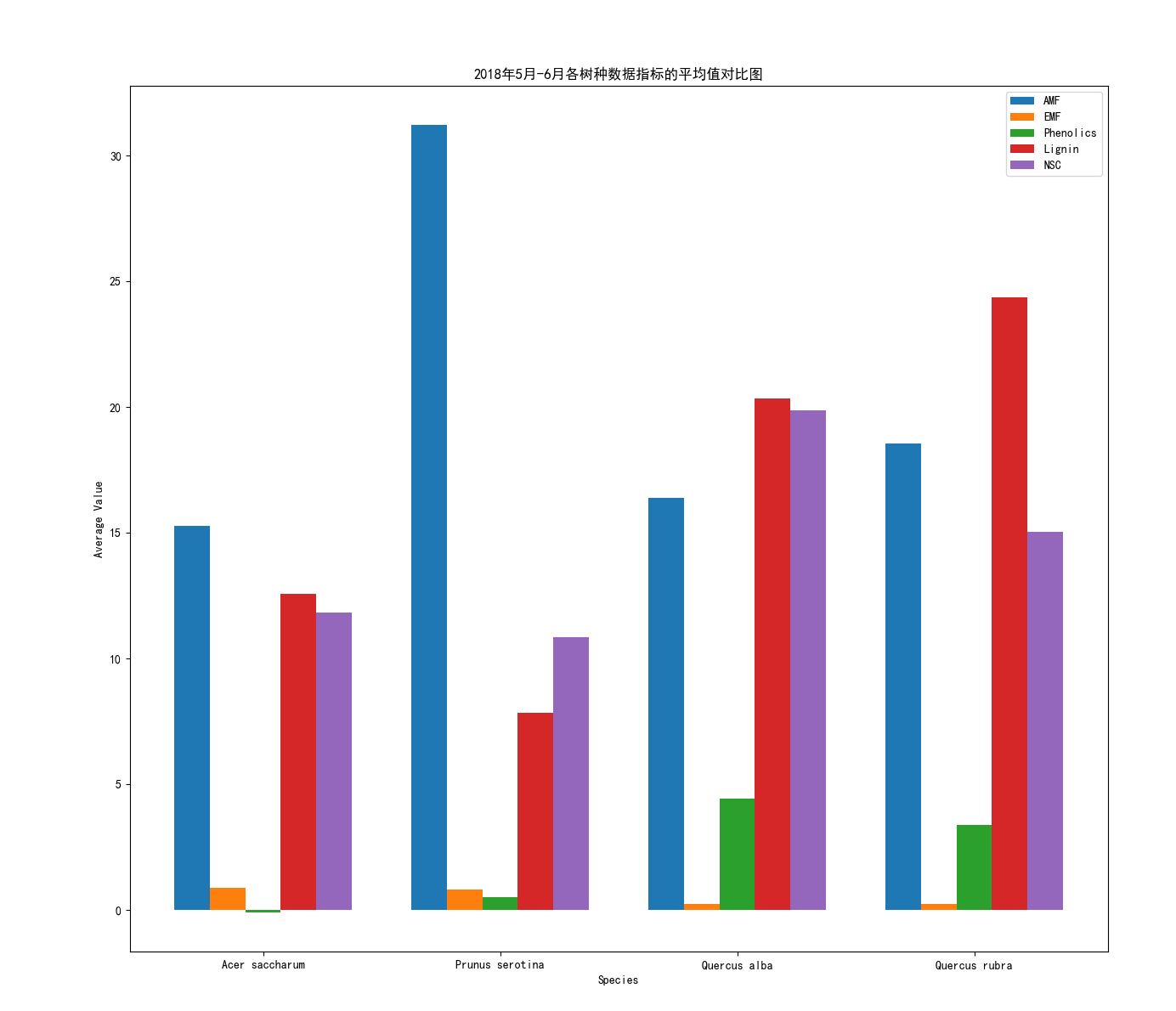
每个树种对于生长的所需元素是不一样的,按照四种不同同属的乔木植物讨论。
- Prunus serotina对于AMF(丛枝菌根真菌)的需求是最高的。
- Acer saccharum对于AMF(丛枝菌根真菌)的需求是最高的。
- Quercus rubra对于Lignin(木质素)的需求是最高的。
- Quercus alba对于Lignin(木质素)的需求是最高的。
5.5各个树种土壤影响因子散点图展示
分类提取Soil的Prunus serotina、Quercus rubra、Acer rubrum、Populus grandidentata、Sterile、Acer saccharum、Quercus alba的,取得各个AMF、EMF、Phenolics、Lignin、NSC数据,分析每一个树种在实验结束后取得各个数据指标的平均值,绘制散点图,并对比:
1 import pandas as pd 2 import matplotlib.pyplot as plt 3 4 # 导入数据集 5 data = pd.read_csv('Cleaned_Tree_Dataset.csv') 6 # 指定要提取的树种和数据指标 7 species_list = ['Prunus serotina', 'Quercus rubra', 'Acer rubrum', 'Populus grandidentata', 'Sterile', 'Acer saccharum', 'Quercus alba'] 8 indicators = ['AMF', 'EMF', 'Phenolics', 'Lignin', 'NSC'] 9 # 提取所需的数据列和指定的树种 10 columns = ['Species'] + indicators 11 subset = data[data['Species'].isin(species_list)][columns] 12 # 绘制散点图 13 for indicator in indicators: 14 plt.figure() 15 for species in species_list: 16 species_data = subset[subset['Species'] == species] 17 x = range(len(species_data)) 18 y = species_data[indicator] 19 plt.scatter(x, y, label=species) 20 plt.xlabel('Data Point') 21 plt.ylabel(indicator) 22 plt.title(f'{indicator} Comparison') 23 plt.legend() 24 # 显示图表 25 plt.show()
实验结果:
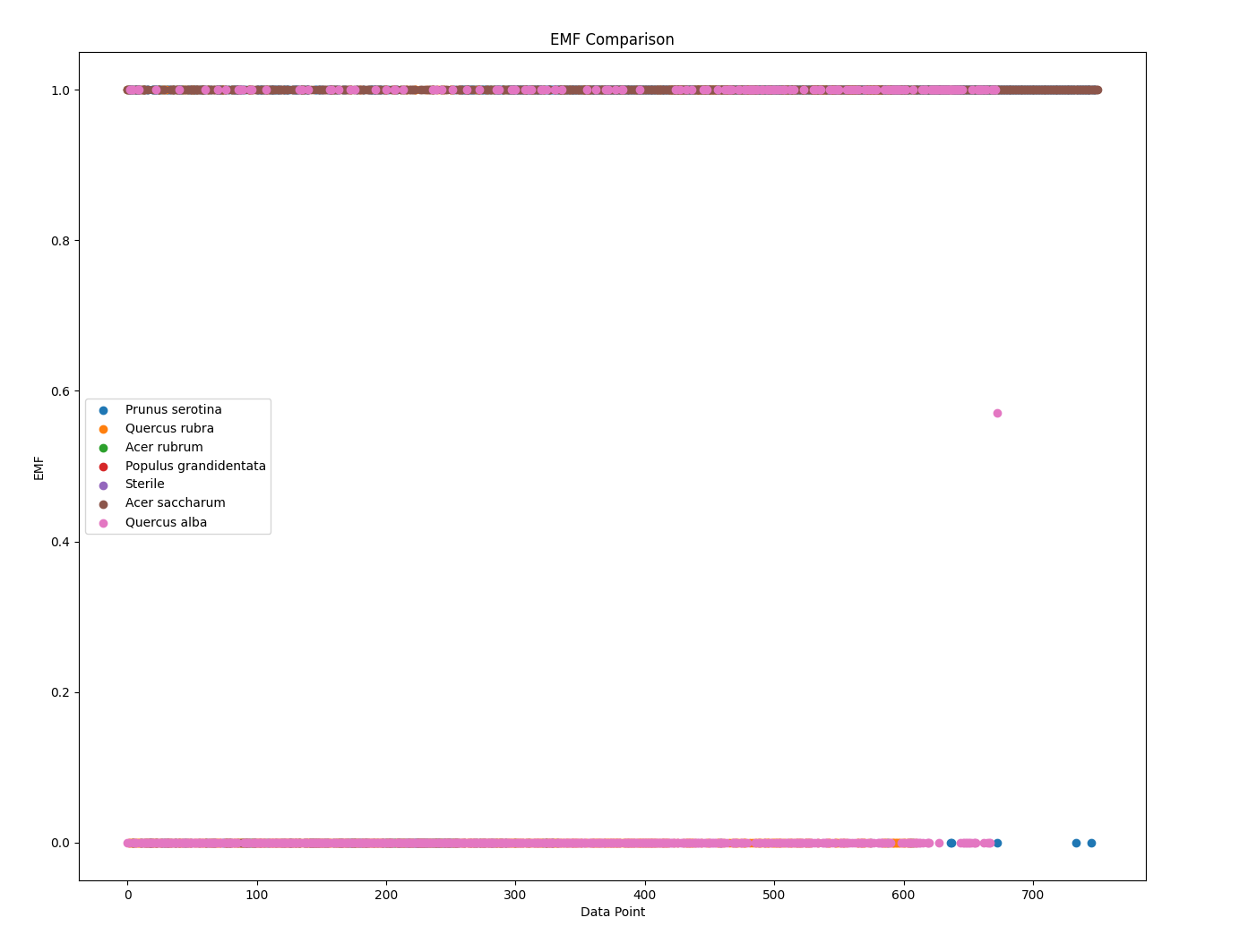
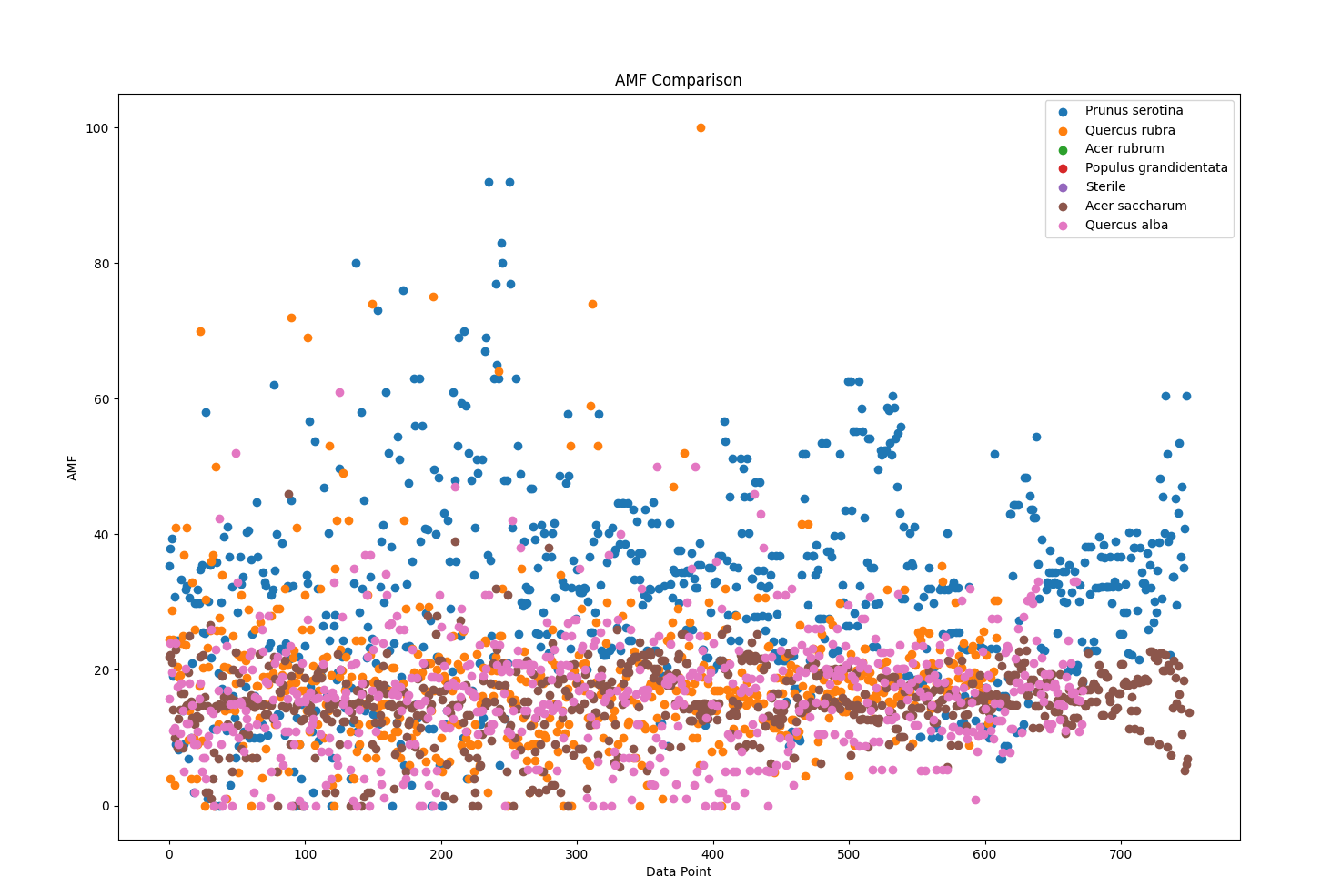
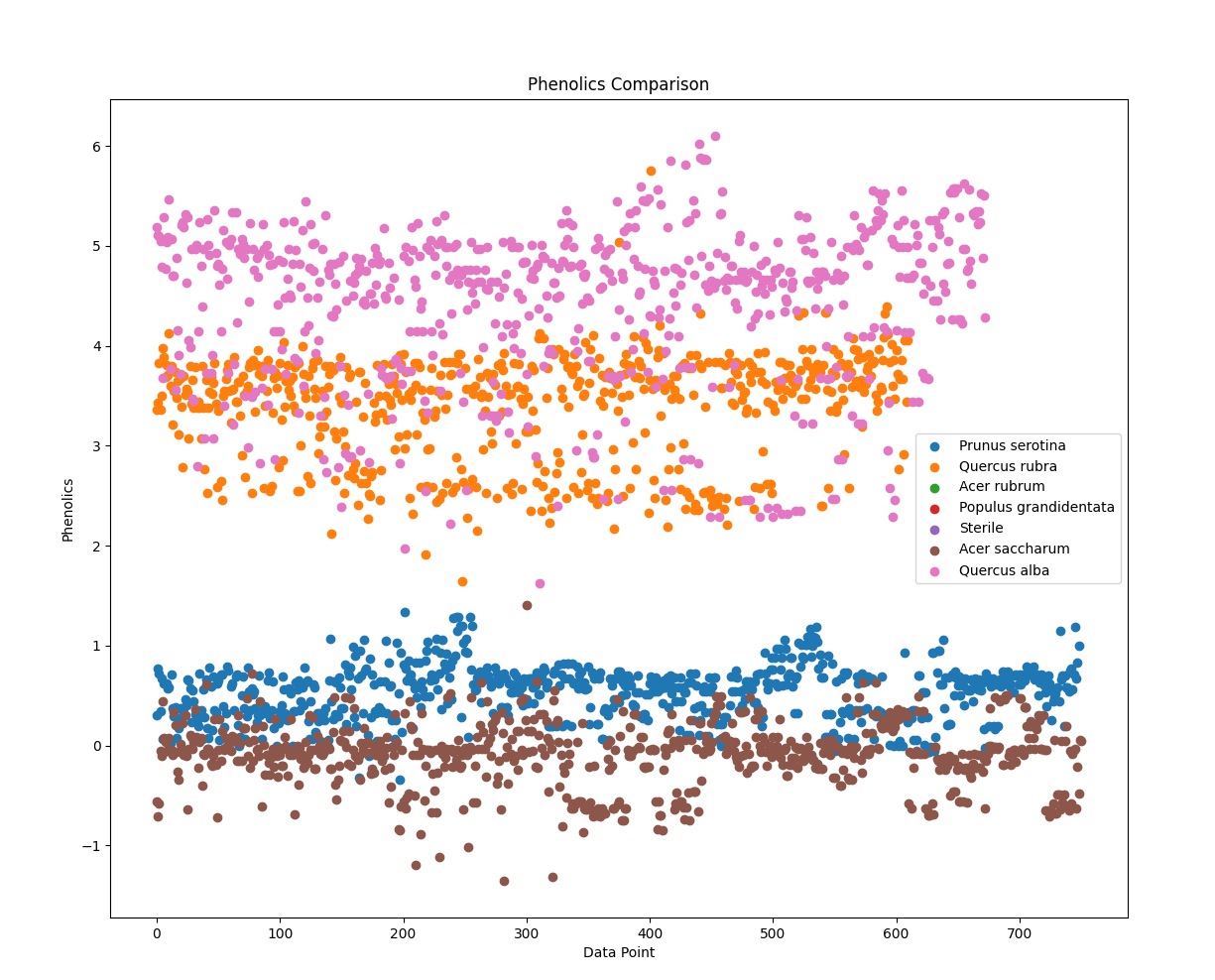
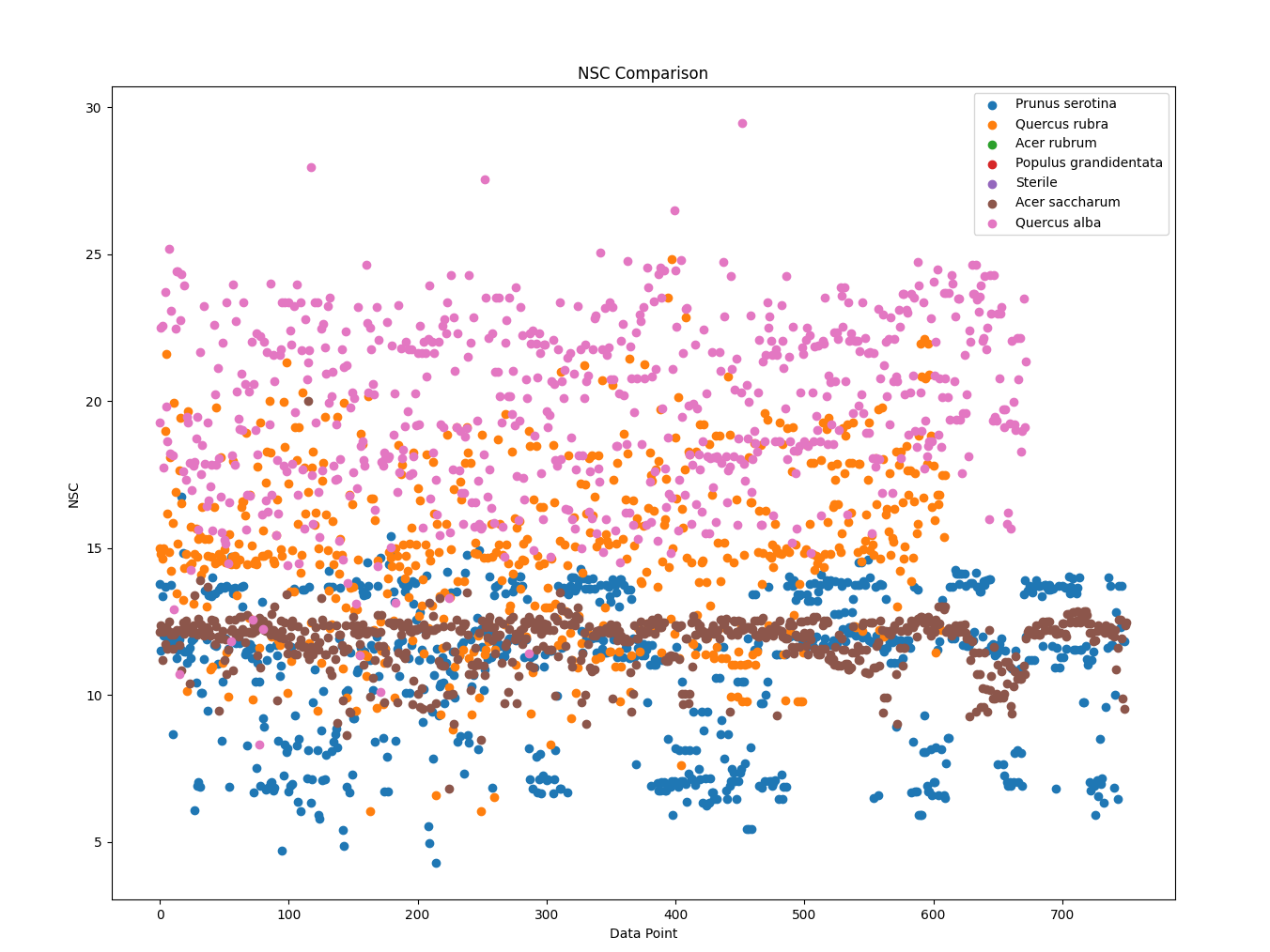
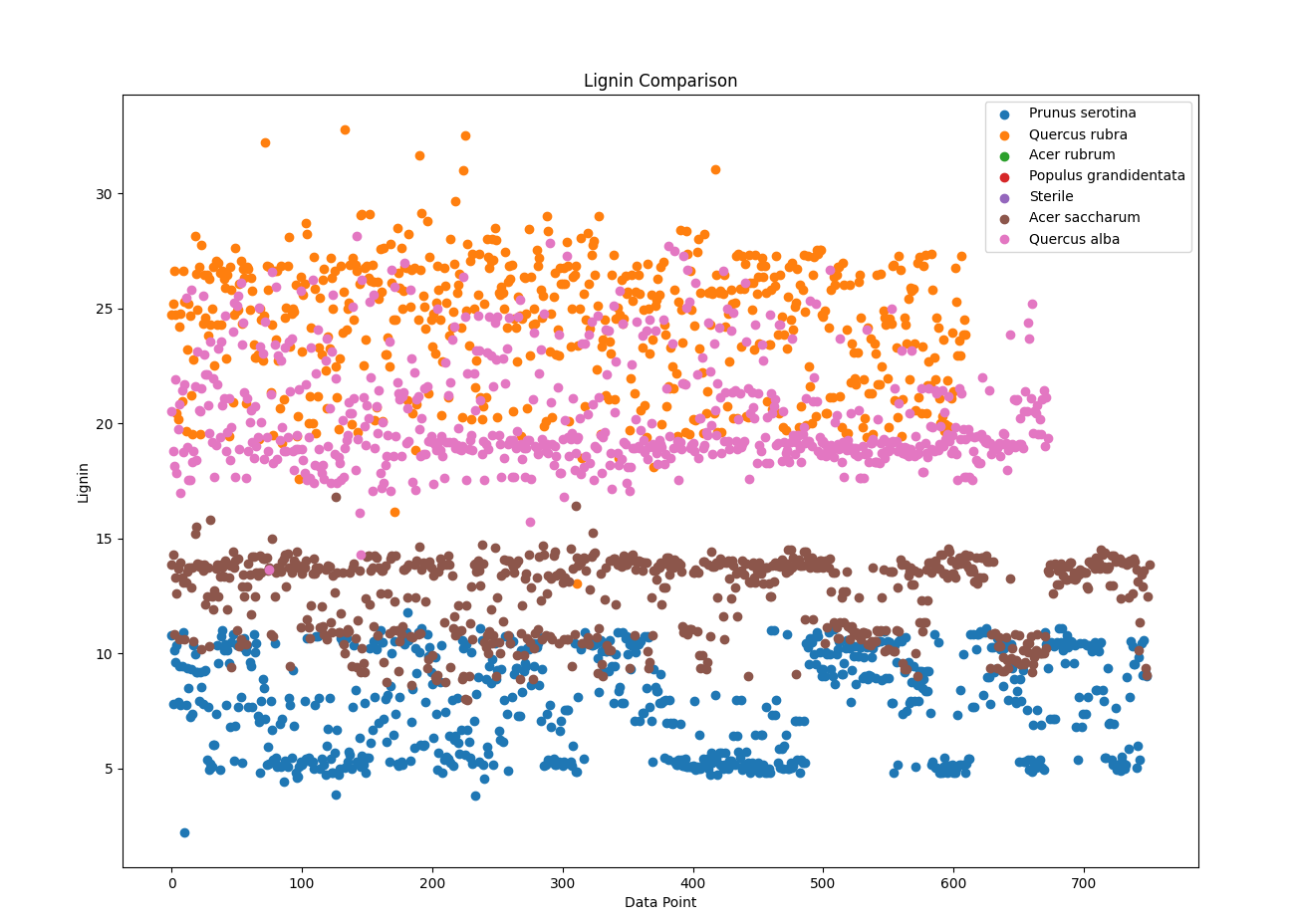
不同树种在散点图上面产生了不同的趋势,能够充分的展现出所属种的特征,基本能够证明,不同的种类生存需要不同的影响元素因子。而在同一科的乔木类,体现了差别迥异的特征,不能够一概而论,每一种的树木都有自己的所需环境,土壤和菌类都有严格要求。
5.6乔木类有无菌,影响因子雷达图展示
土壤有菌和无菌对于树木生长所需的各个元素之间的变化关系平均值:
1 import pandas as pd 2 import matplotlib.pyplot as plt 3 import numpy as np 4 5 plt.rcParams['font.sans-serif'] = ['SimHei'] 6 # 导入数据集 7 data = pd.read_csv('Cleaned_Tree_Dataset.csv') 8 # 指定要提取的数据指标和Sterile类型 9 indicators = ['AMF', 'EMF', 'Phenolics', 'Lignin', 'NSC'] 10 sterile_types = ['Non-Sterile', 'Sterile'] 11 # 提取所需的数据列和指定的Sterile类型 12 columns = ['Species', 'Sterile'] + indicators 13 subset = data[data['Sterile'].isin(sterile_types)][columns] 14 # 计算每个数据指标的平均值 15 averages = subset.groupby(['Species', 'Sterile']).mean().reset_index() 16 # 创建雷达图 17 num_indicators = len(indicators) 18 # 计算角度 19 angles = np.linspace(0, 2 * np.pi, num_indicators, endpoint=False).tolist() 20 angles += angles[:1] 21 # 设置图表属性 22 plt.figure(figsize=(8, 8)) 23 ax = plt.subplot(111, polar=True) 24 ax.set_xticks(angles[:-1]) 25 ax.set_xticklabels(indicators) 26 ax.set_yticklabels([]) 27 # 绘制雷达图 28 for sterile_type in sterile_types: 29 species_data = averages[averages['Sterile'] == sterile_type] 30 values = species_data[indicators].values.tolist()[0] 31 values += values[:1] 32 ax.plot(angles, values, label=sterile_type) 33 ax.fill(angles, values, alpha=0.25) 34 # 添加图例 35 plt.legend() 36 # 显示图表 37 plt.title('2018年5月-6月各树种在有菌和无菌的数据指标值对比雷达图') 38 plt.show()
实验结果:
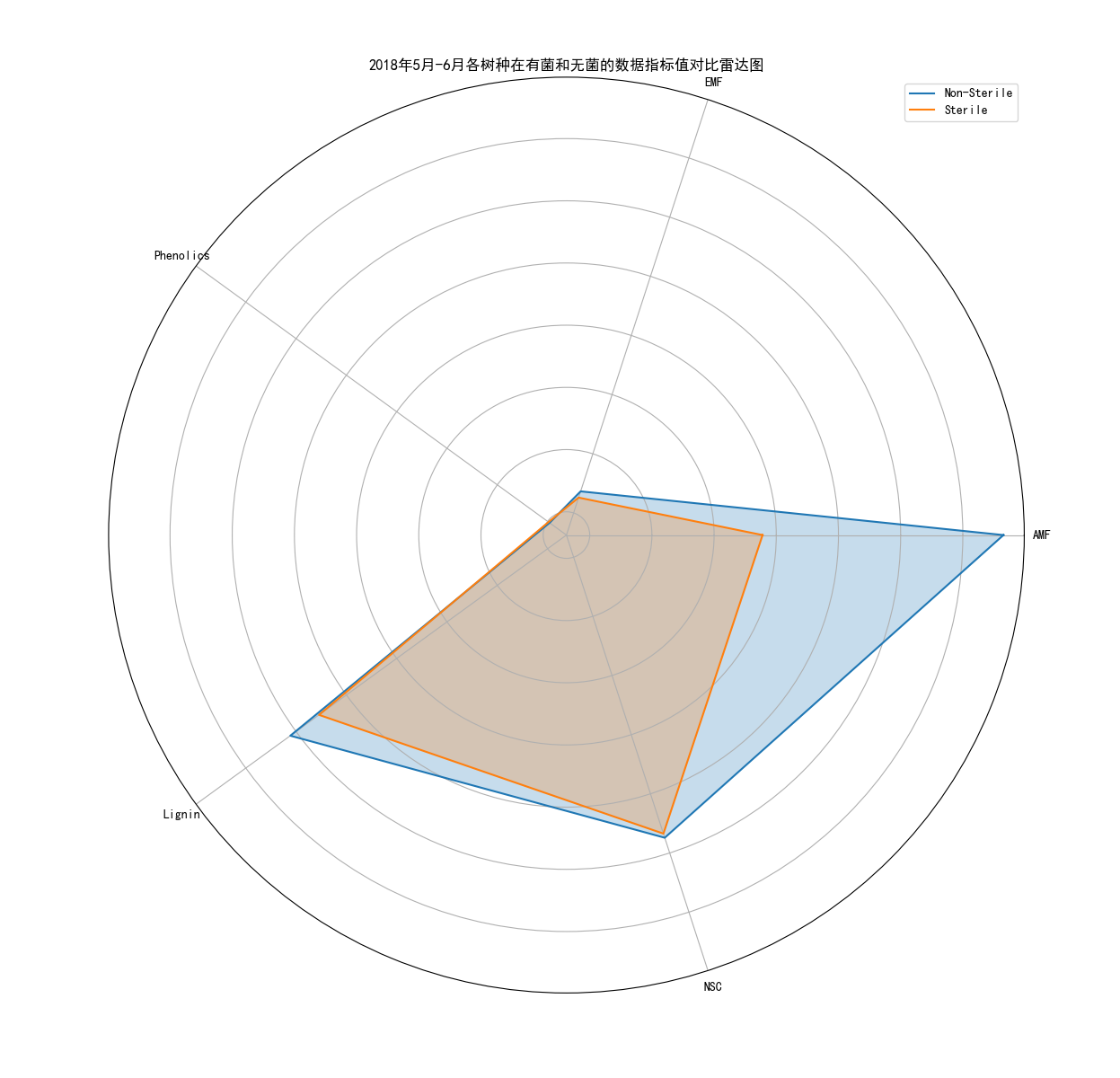
在无菌环境下,通过雷达图表现出树木成长更好,并且生长影响因子,能够更加丰富,吸收更好,有助于树木的生长。
5.7单个树种和土壤环境影响因子数据输出、折线图预测模型
导入数据集,输入树种,输入土壤环境构建预测模型,提取该条件的各个AMF、EMF、Phenolics、Lignin、NSC数据计算平均值,最后打印输出表格,绘制折线图
1 import pandas as pd 2 import matplotlib.pyplot as plt 3 4 plt.rcParams['font.sans-serif'] = ['SimHei'] 5 # 导入数据集 6 data = pd.read_csv('Cleaned_Tree_Dataset.csv') 7 # 输入树种和土壤环境 8 species = input("请输入树种: ") 9 soil = input("请输入土壤环境: ") 10 # 指定要提取的数据指标 11 indicators = ['AMF', 'EMF', 'Phenolics', 'Lignin', 'NSC'] 12 # 提取满足条件的数据 13 subset = data[(data['Species'] == species) & (data['Soil'] == soil)][indicators] 14 # 计算每个数据指标的平均值 15 averages = subset.mean() 16 # 打印输出表格 17 print(f"树种: {species}, 土壤环境: {soil}") 18 print("各个数据指标的平均值:") 19 print(averages) 20 # 绘制折线图 21 plt.figure(figsize=(10, 6)) 22 for indicator in indicators: 23 plt.plot(subset[indicator], label=indicator) 24 plt.xlabel('样本') 25 plt.ylabel('数值') 26 plt.title(f"{species}在{soil}条件下各个数据指标的折线图") 27 plt.legend() 28 plt.show()
数据输出:

数据折线图:
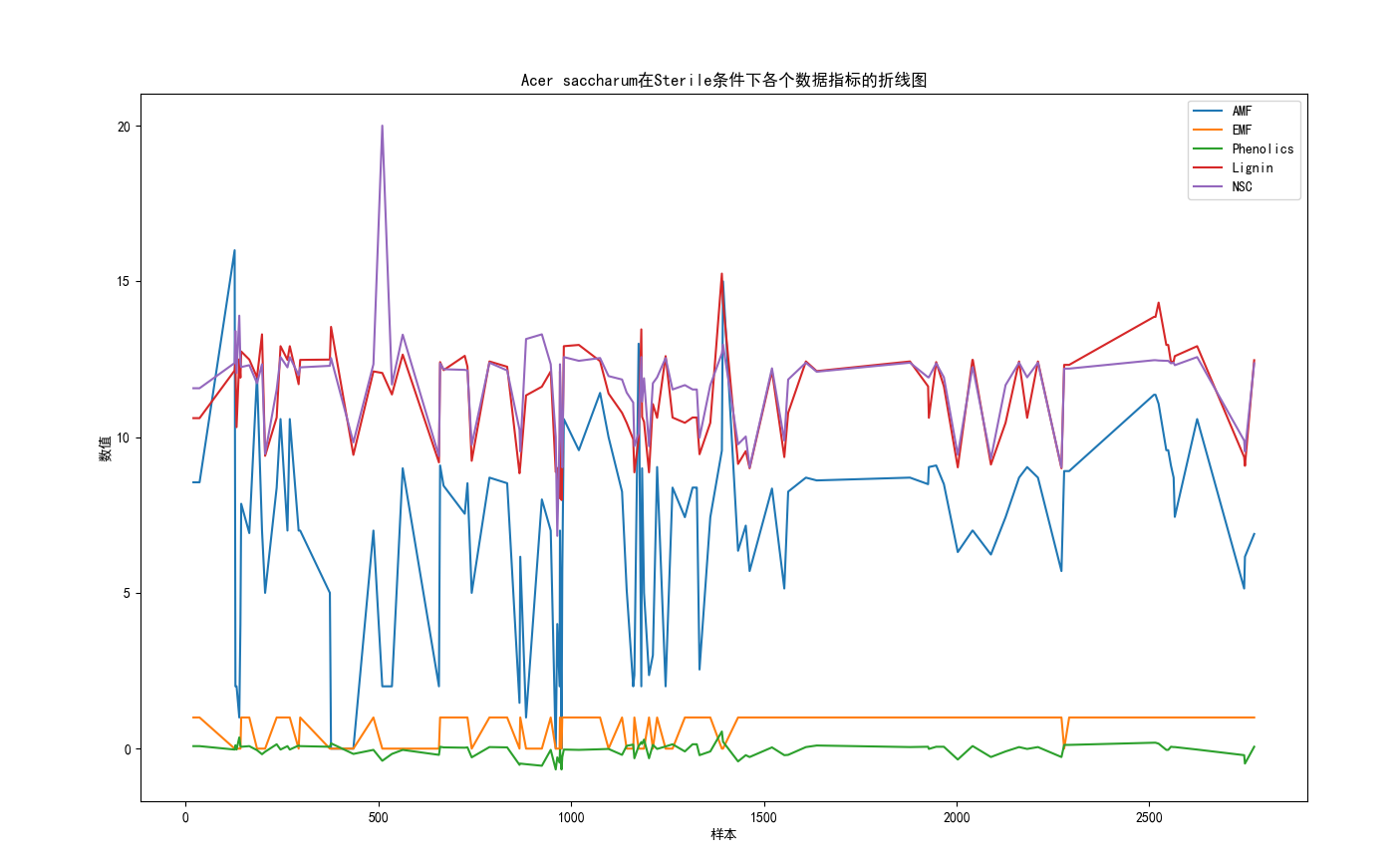
折线图的波动是由于无菌环境下,对于植物生长的影响,大部分数据均处于正常水平,可以得到完整Acer saccharum的树木分析,在Sterile土壤环境下,正常水平。
|
影响因子 |
各个数据指标的平均值: |
|
AMF |
6.949429 |
|
EMF |
0.647619 |
|
Phenolics |
-0.045048 |
|
Lignin |
11.360095 |
|
NSC |
11.683333 |
5.8各个树种土壤影响因子柱状图对比展示
导入数据集,分类提取Sterile的Non-Sterile和Sterile,各个树种的AMF、EMF、Phenolics、Lignin、NSC数据对比,分析每一个树种在实验结束后取得各个数据指标的平均值,输出对比柱状图
1 import pandas as pd 2 import matplotlib.pyplot as plt 3 4 plt.rcParams['font.sans-serif'] = ['SimHei'] 5 # 导入数据集 6 data = pd.read_csv('Cleaned_Tree_Dataset.csv') 7 # 分类提取Sterile的Non-Sterile和Sterile数据 8 sterile_data = data[data['Sterile'] == 'Sterile'] 9 non_sterile_data = data[data['Sterile'] == 'Non-Sterile'] 10 # 获取所有树种的唯一值 11 species = data['Species'].unique() 12 # 计算每个树种的数据指标平均值 13 sterile_averages = sterile_data.groupby('Species')[['AMF', 'EMF', 'Phenolics', 'Lignin', 'NSC']].mean() 14 non_sterile_averages = non_sterile_data.groupby('Species')[['AMF', 'EMF', 'Phenolics', 'Lignin', 'NSC']].mean() 15 # 设置柱状图的宽度和位置 16 bar_width = 0.35 17 index = range(len(species)) 18 opacity = 0.8 19 # 创建图表对象 20 fig, ax = plt.subplots() 21 # 绘制Sterile数据的柱状图 22 sterile_bars = ax.bar(index, sterile_averages.loc[species]['AMF'], bar_width, alpha=opacity, color='b', label='Sterile') 23 # 绘制Non-Sterile数据的柱状图 24 non_sterile_bars = ax.bar([i + bar_width for i in index], non_sterile_averages.loc[species]['AMF'], bar_width, alpha=opacity, color='r', label='Non-Sterile') 25 # 设置图表标题和x轴标签 26 ax.set_title('不同树种的AMF数据对比') 27 ax.set_xlabel('树种') 28 ax.set_xticks([i + bar_width/2 for i in index]) 29 ax.set_xticklabels(species, rotation=45) 30 # 设置y轴标签 31 ax.set_ylabel('平均值') 32 # 添加图例 33 ax.legend() 34 # 显示图表 35 plt.tight_layout() 36 plt.show() 37 # 生成不同树种的EMF、Phenolics、Lignin、NSC数据对比柱状图 38 fig, ax = plt.subplots(2, 2, figsize=(10, 8)) 39 # 绘制EMF数据对比柱状图 40 emf_bars = ax[0, 0].bar(index, sterile_averages.loc[species]['EMF'], bar_width, alpha=opacity, color='b', label='Sterile') 41 ax[0, 0].bar([i + bar_width for i in index], non_sterile_averages.loc[species]['EMF'], bar_width, alpha=opacity, color='r', label='Non-Sterile') 42 ax[0, 0].set_title('EMF数据对比') 43 ax[0, 0].set_xlabel('树种') 44 ax[0, 0].set_ylabel('平均值') 45 ax[0, 0].set_xticks([i + bar_width/2 for i in index]) 46 ax[0, 0].set_xticklabels(species, rotation=45) 47 ax[0, 0].legend() 48 # 绘制Phenolics数据对比柱状图 49 phenolics_bars = ax[0, 1].bar(index, sterile_averages.loc[species]['Phenolics'], bar_width, alpha=opacity, color='b', label='Sterile') 50 ax[0, 1].bar([i + bar_width for i in index], non_sterile_averages.loc[species]['Phenolics'], bar_width, alpha=opacity, color='r', label='Non-Sterile') 51 ax[0, 1].set_title('Phenolics数据对比') 52 ax[0, 1].set_xlabel('树种') 53 ax[0, 1].set_ylabel('平均值') 54 ax[0, 1].set_xticks([i + bar_width/2 for i in index]) 55 ax[0, 1].set_xticklabels(species, rotation=45) 56 ax[0, 1].legend() 57 # 绘制Lignin数据对比柱状图 58 lignin_bars = ax[1, 0].bar(index, sterile_averages.loc[species]['Lignin'], bar_width, alpha=opacity, color='b', label='Sterile') 59 ax[1, 0].bar([i + bar_width for i in index], non_sterile_averages.loc[species]['Lignin'], bar_width, alpha=opacity, color='r', label='Non-Sterile') 60 ax[1, 0].set_title('Lignin数据对比') 61 ax[1, 0].set_xlabel('树种') 62 ax[1, 0].set_ylabel('平均值') 63 ax[1, 0].set_xticks([i + bar_width/2 for i in index]) 64 ax[1, 0].set_xticklabels(species, rotation=45) 65 ax[1, 0].legend() 66 # 绘制NSC数据对比柱状图 67 nsc_bars = ax[1, 1].bar(index, sterile_averages.loc[species]['NSC'], bar_width, alpha=opacity, color='b', label='Sterile') 68 ax[1, 1].bar([i + bar_width for i in index], non_sterile_averages.loc[species]['NSC'], bar_width, alpha=opacity, color='r', label='Non-Sterile') 69 ax[1, 1].set_title('NSC数据对比') 70 ax[1, 1].set_xlabel('树种') 71 ax[1, 1].set_ylabel('平均值') 72 ax[1, 1].set_xticks([i + bar_width/2 for i in index]) 73 ax[1, 1].set_xticklabels(species, rotation=45) 74 ax[1, 1].legend() 75 # 调整子图之间的间距 76 plt.tight_layout() 77 78 # 显示图表 79 plt.show()
实验结果:
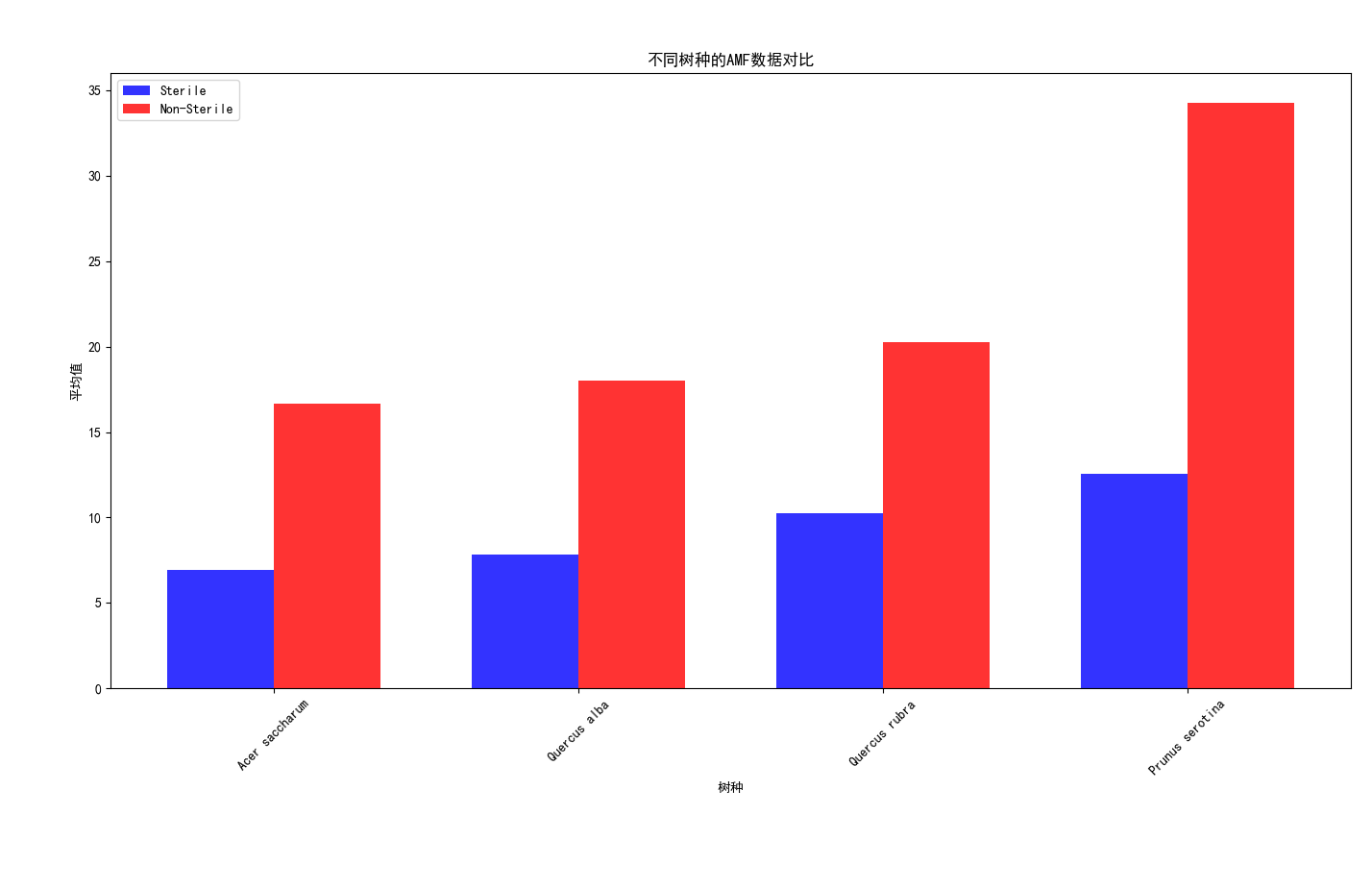
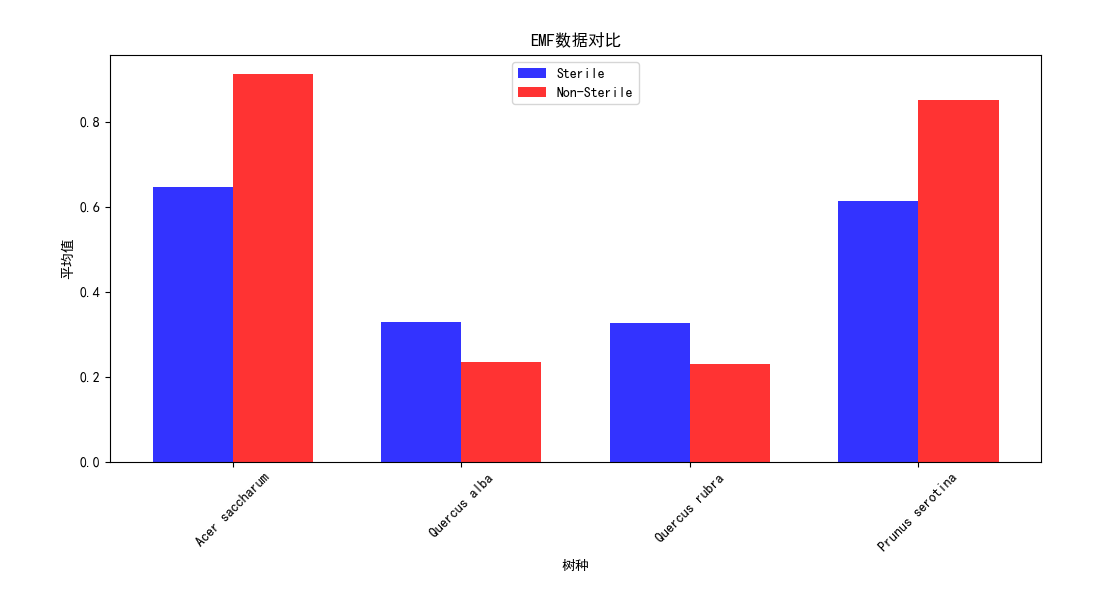
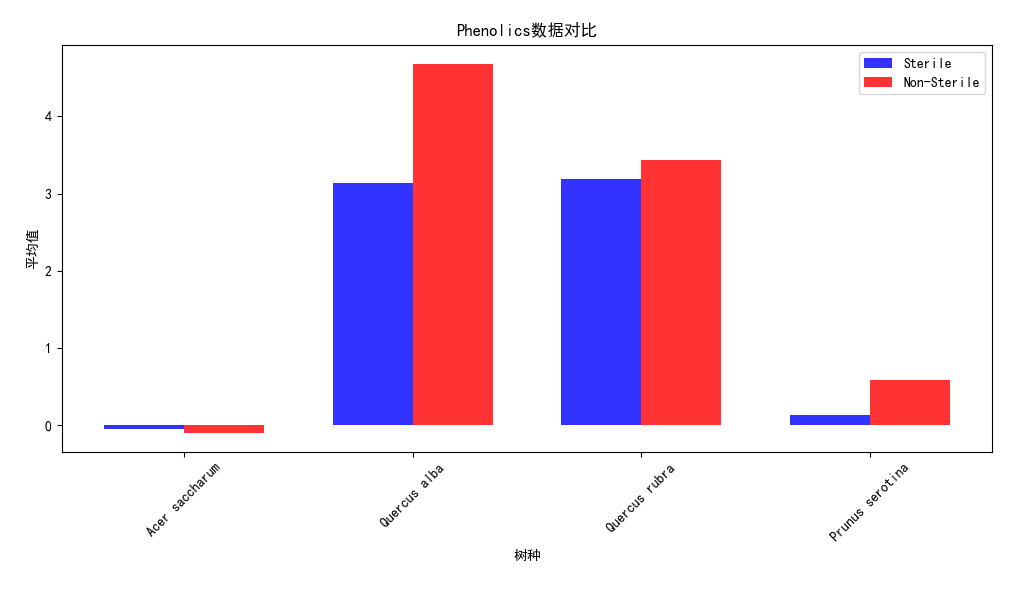
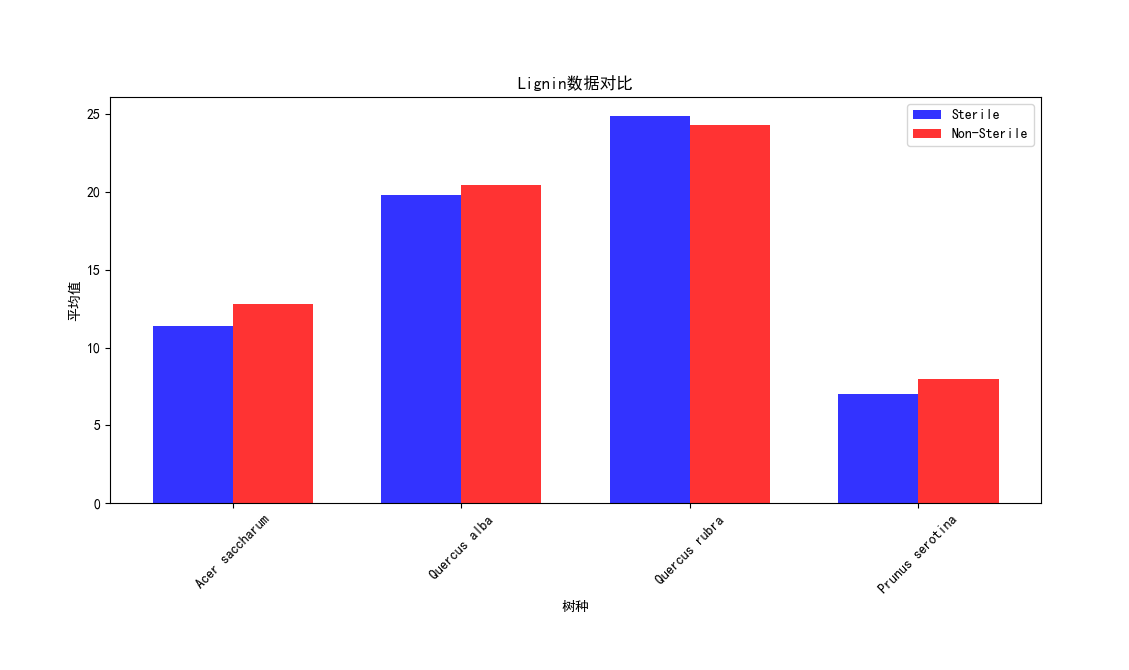
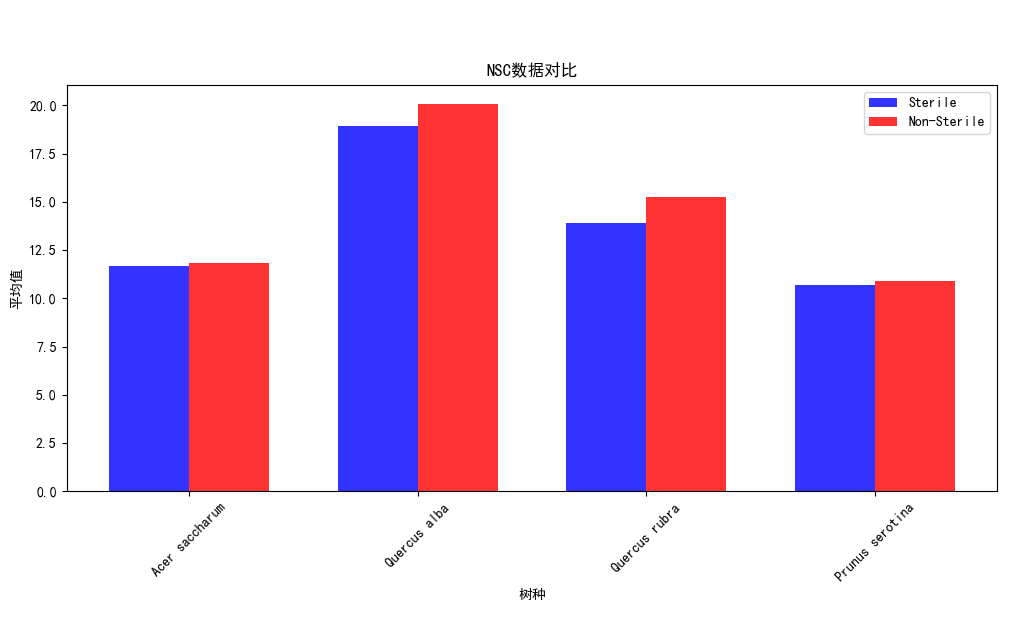
Acer saccharum、、Quercus rubra、Quercus alba、Prunus serotina树种,在有菌和无菌环境下,的数据对比。可以发现先无菌情况下,生存影响化合物数值均有提高,足以证明无菌环境对于植物生存的重要性,对于Lignin木质素、NSC(非结构碳水化合物)这两项数值的增长并不明显,益处不大。
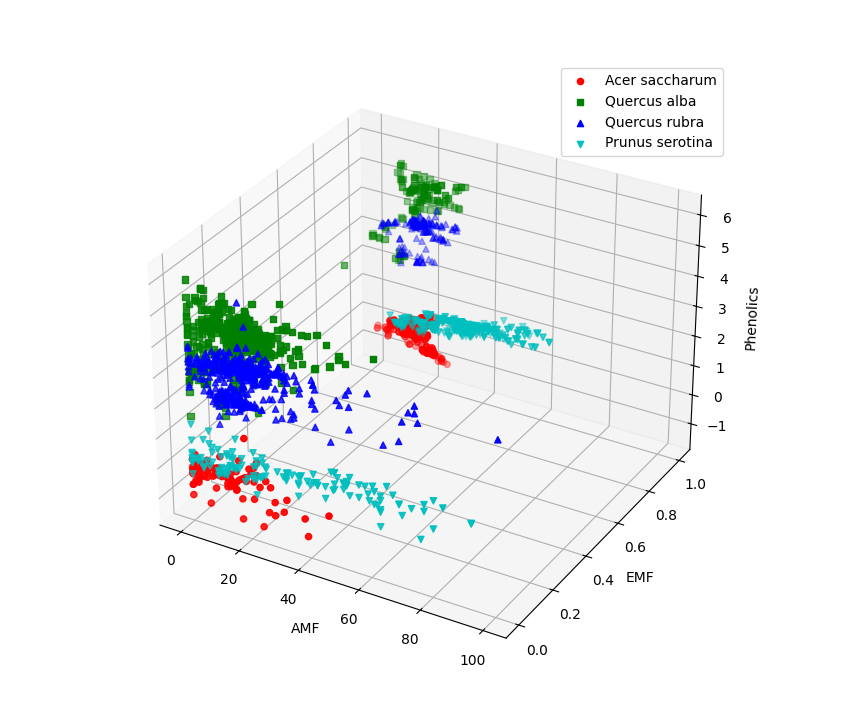
5.9单个树种的AMF、EMF、Phenolics的生长元素,输出为三维视图模型
1 import pandas as pd 2 import matplotlib.pyplot as plt 3 from mpl_toolkits.mplot3d import Axes3D 4 5 # 导入数据集 6 data = pd.read_csv("Cleaned_Tree_Dataset.csv") 7 8 # 提取特征和目标变量 9 features = data[['AMF', 'EMF', 'Phenolics']] 10 target = data['Species'] 11 12 # 创建字典以存储每个树种的数据 13 species_data = {} 14 15 # 根据树种分组,将数据存储到字典中 16 for species in target.unique(): 17 species_data[species] = features[target == species] 18 19 # 创建三维视图 20 fig = plt.figure(figsize=(10, 8)) 21 ax = fig.add_subplot(111, projection='3d') 22 23 # 定义不同树种的颜色和标记样式 24 colors = ['r', 'g', 'b', 'c', 'm', 'y', 'k'] 25 markers = ['o', 's', '^', 'v', 'D', 'p', '*'] 26 27 # 绘制每个树种的三维视图 28 for i, species in enumerate(species_data): 29 ax.scatter(species_data[species]['AMF'], species_data[species]['EMF'], species_data[species]['Phenolics'], 30 c=colors[i], marker=markers[i], label=species) 31 32 # 设置坐标轴标签 33 ax.set_xlabel('AMF') 34 ax.set_ylabel('EMF') 35 ax.set_zlabel('Phenolics') 36 37 # 添加图例 38 ax.legend() 39 40 # 显示图形 41 plt.show()
程序输出结果:
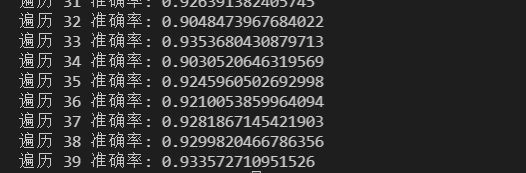
5.10借用机器学习,提高树木生存预测模型的真确率
导入数据集,提取数据集中No Plot Subplot Species Light_ISF Light_Cat Core Soil Adult Sterile Conspecific Myco SoilMyco PlantDate AMF EMF Phenolics Lignin NSC Census Time Event Harvest元素,使用机器学习,建立基卷,遍历39次增加预测准确性;
实现代码:
1 import pandas as pd 2 from sklearn.model_selection import train_test_split 3 from sklearn.linear_model import LogisticRegression 4 from sklearn.metrics import accuracy_score 5 data = pd.read_csv("Cleaned_Tree_Dataset.csv") 6 features = data[['AMF', 'EMF', 'Phenolics', 'Lignin', 'NSC']] 7 target = data['Species'] 8 9 X_train, X_test, y_train, y_test = train_test_split(features, target, test_size=0.2, random_state=42) 10 model = LogisticRegression() 11 model.fit(X_train, y_train) 12 predictions = model.predict(X_test) 13 accuracy = accuracy_score(y_test, predictions) 14 print("基准模型准确率:", accuracy) 15 num_iterations = 39 16 accuracies = [] 17 18 for i in range(num_iterations): 19 X_train, X_test, y_train, y_test = train_test_split(features, target, test_size=0.2, random_state=i) 20 model = LogisticRegression() 21 model.fit(X_train, y_train) 22 predictions = model.predict(X_test) 23 accuracy = accuracy_score(y_test, predictions) 24 accuracies.append(accuracy) 25 26 # 输出每次遍历的准确率 27 for i, accuracy in enumerate(accuracies): 28 print("遍历", i+1, "准确率:", accuracy)
程序实验结果:
机器学习已经成为了现代科技发展的关键驱动力,尤其在数据科学和预测模型的构建中。对于树木生存预测模型来说,借助机器学习技术可以显著提高预测真确率。传统的生存分析方法,如比例风险回归模型,虽然在某些情境下仍然适用,但它们受到一些假设条件的限制,如比例风险假定和对数线性假定。
随机生存森林模型是机器学习中处理生存数据的一种扩展方法,它结合了随机森林的强大功能并针对生存数据进行了优化。与传统方法相比,随机生存森林模型的优势在于它不受上述假设条件的约束,从而在更广泛的情境下都能提供准确的预测结果。此外,随着技术的进步和研究的深入,越来越多的机器学习工具和库,都被开发出来,以帮助研究者更方便地构建和评估这些高级的预测模型。
5.11运用机器学习结果,预测各个树种的生长情况
导入数据集,提取数据集中No Plot Subplot Species Light_ISF Light_Cat Core Soil Adult Sterile Conspecific Myco SoilMyco PlantDate AMF EMF Phenolics Lignin NSC Census Time Event Harvest元素,输出最后的预测生长状况;
实现代码:
1 import pandas as pd 2 from sklearn.model_selection import train_test_split 3 from sklearn.linear_model import LogisticRegression 4 from sklearn.metrics import classification_report 5 6 # 导入数据集 7 data = pd.read_csv("Cleaned_Tree_Dataset.csv") 8 9 # 提取特征和目标变量 10 features = data[['AMF', 'EMF', 'Phenolics', 'Lignin', 'NSC']] 11 target = data['Species'] 12 13 # 数据集划分为训练集和测试集 14 X_train, X_test, y_train, y_test = train_test_split(features, target, test_size=0.2, random_state=42) 15 16 # 构建模型(逻辑回归) 17 model = LogisticRegression() 18 19 # 模型训练 20 model.fit(X_train, y_train) 21 22 # 预测 23 predictions = model.predict(X_test) 24 25 # 输出预测结果 26 report = classification_report(y_test, predictions) 27 print(report)
程序实验结果:

运用机器学习提高树木生存模型的准确性具有重要的意义。传统的生存分析方法虽然在生物医学领域有广泛应用,但需要满足一些前提假设。然而,随机生存森林方法可以克服这些弱点,它的预测准确度至少等同于或优于传统生存分析方法。
更重要的是,随机生存森林模型不受比例风险假定、对数线性假定等条件的约束。此外,它还能够对变量的重要性进行排名,帮助我们理解哪些因素对树木生存率的影响最大。通过构建机器学习算法的树桉树适宜性评价模型,我们可以更好地预测桉树的适宜性,为科学造林提供依据。
大数据分析所有代码:
1 import pandas as pd 2 3 # 导入数据集 4 data = pd.read_csv('Cleaned_Tree_Dataset.csv') 5 # 数据清洗:去除缺失值或异常值 6 data = data.dropna() # 去除包含缺失值的行 7 # 其他数据清洗操作... 8 # 获取所有树种的唯一值 9 species = data['Species'].unique() 10 # 计算每个树种的数据指标平均值 11 averages = data.groupby(['Species'])[['AMF', 'EMF', 'Phenolics', 'Lignin', 'NSC']].mean() 12 # 输出数据对比表格 13 comparison_table = pd.DataFrame(averages.loc[species], columns=['AMF', 'EMF', 'Phenolics', 'Lignin', 'NSC']) 14 print(comparison_table) 15 16 import pandas as pd 17 from sklearn.model_selection import train_test_split 18 19 # 导入数据集 20 data = pd.read_csv('Cleaned_Tree_Dataset.csv') 21 # 划分特征和目标变量 22 X = data.drop('Species', axis=1) # 特征 23 y = data['Species'] # 目标变量 24 # 数据集划分 25 X_train, X_test, y_train, y_test = train_test_split(X, y, test_size=0.2, random_state=42) 26 # 获取所有树种的唯一值 27 species = data['Species'].unique() 28 # 计算训练集每个树种的数据指标平均值 29 train_averages = X_train.groupby(y_train)[['AMF', 'EMF', 'Phenolics', 'Lignin', 'NSC']].mean() 30 # 输出训练集数据对比表格 31 train_comparison_table = pd.DataFrame(train_averages.loc[species], columns=['AMF', 'EMF', 'Phenolics', 'Lignin', 'NSC']) 32 print("训练集数据对比表格:") 33 print(train_comparison_table) 34 # 计算测试集每个树种的数据指标平均值 35 test_averages = X_test.groupby(y_test)[['AMF', 'EMF', 'Phenolics', 'Lignin', 'NSC']].mean() 36 # 输出测试集数据对比表格 37 test_comparison_table = pd.DataFrame(test_averages.loc[species], columns=['AMF', 'EMF', 'Phenolics', 'Lignin', 'NSC']) 38 print("测试集数据对比表格:") 39 print(test_comparison_table) 40 41 42 from textwrap import wrap 43 import pandas as pd 44 import matplotlib.pyplot as plt 45 import numpy as np 46 47 plt.rcParams['font.sans-serif'] = ['SimHei'] 48 # 导入数据集 49 df = pd.read_csv('Cleaned_Tree_Dataset.csv') 50 # 创建图形 51 fig = plt.figure() 52 # 计算每种类型的数量 53 type_counts = df['Species'].value_counts() 54 # 计算总数量 55 total = type_counts.sum() 56 # 计算占比 57 percentages = (type_counts / total) * 100 58 # 绘制饼图 59 plt.pie(percentages, labels=percentages.index, autopct='%1.1f%%') 60 plt.title('种类总数占比图') 61 plt.show() 62 63 64 from textwrap import wrap 65 import pandas as pd 66 import matplotlib.pyplot as plt 67 import numpy as np 68 69 plt.rcParams['font.sans-serif'] = ['SimHei'] 70 # 导入数据集 71 df = pd.read_csv('Cleaned_Tree_Dataset.csv') 72 # 创建图形 73 fig = plt.figure() 74 # 计算每种类型的数量 75 type_counts = df['Conspecific'].value_counts() 76 # 计算总数量 77 total = type_counts.sum() 78 # 计算占比 79 percentages = (type_counts / total) * 100 80 # 绘制饼图 81 plt.pie(percentages, labels=percentages.index, autopct='%1.1f%%') 82 plt.title('数据同种的总数占比图') 83 plt.show() 84 85 86 import pandas as pd 87 import matplotlib.pyplot as plt 88 89 # 导入数据集 90 data = pd.read_csv('Cleaned_Tree_Dataset.csv') 91 # 指定要提取的树种 92 species_list = ['Acer saccharum', 'Quercus rubra', 'Quercus alba', 'Prunus serotina'] 93 subset = data[data['Species'].isin(species_list)] 94 # 提取所需的数据列 95 columns = ['Species', 'AMF', 'EMF', 'Phenolics', 'Lignin', 'NSC'] 96 subset = subset[columns] 97 # 计算每个数据指标的平均值 98 averages = subset.groupby('Species').mean() 99 # 创建表格 100 fig, ax = plt.subplots() 101 ax.axis('off') 102 table = ax.table(cellText=averages.values, colLabels=averages.columns, rowLabels=averages.index, loc='center') 103 # 设置表格样式 104 table.auto_set_font_size(False) 105 table.set_fontsize(12) 106 table.scale(1.2, 1.2) 107 # 显示表格 108 plt.show() 109 110 111 import pandas as pd 112 import matplotlib.pyplot as plt 113 114 plt.rcParams['font.sans-serif'] = ['SimHei'] 115 # 导入数据集 116 data = pd.read_csv('Cleaned_Tree_Dataset.csv') 117 # 指定要提取的树种 118 species_list = ['Acer saccharum', 'Quercus rubra', 'Quercus alba', 'Prunus serotina'] 119 subset = data[data['Species'].isin(species_list)] 120 # 提取所需的数据列 121 columns = ['Species', 'AMF', 'EMF', 'Phenolics', 'Lignin', 'NSC'] 122 subset = subset[columns] 123 # 计算每个数据指标的平均值 124 averages = subset.groupby('Species').mean().reset_index() 125 # 绘制柱状图 126 x = range(len(averages)) 127 bar_width = 0.15 128 plt.bar(x, averages['AMF'], width=bar_width, label='AMF') 129 plt.bar([i + bar_width for i in x], averages['EMF'], width=bar_width, label='EMF') 130 plt.bar([i + 2 * bar_width for i in x], averages['Phenolics'], width=bar_width, label='Phenolics') 131 plt.bar([i + 3 * bar_width for i in x], averages['Lignin'], width=bar_width, label='Lignin') 132 plt.bar([i + 4 * bar_width for i in x], averages['NSC'], width=bar_width, label='NSC') 133 # 设置图表属性 134 plt.title('2018年5月-6月各树种数据指标的平均值对比图') 135 plt.xlabel('Species') 136 plt.ylabel('Average Value') 137 plt.xticks([i + 2 * bar_width for i in x], averages['Species']) 138 plt.legend() 139 # 显示图表 140 plt.show() 141 142 143 import pandas as pd 144 import matplotlib.pyplot as plt 145 146 # 导入数据集 147 data = pd.read_csv('Cleaned_Tree_Dataset.csv') 148 # 指定要提取的树种和数据指标 149 species_list = ['Prunus serotina', 'Quercus rubra', 'Acer rubrum', 'Populus grandidentata', 'Sterile', 'Acer saccharum', 'Quercus alba'] 150 indicators = ['AMF', 'EMF', 'Phenolics', 'Lignin', 'NSC'] 151 # 提取所需的数据列和指定的树种 152 columns = ['Species'] + indicators 153 subset = data[data['Species'].isin(species_list)][columns] 154 # 绘制散点图 155 for indicator in indicators: 156 plt.figure() 157 for species in species_list: 158 species_data = subset[subset['Species'] == species] 159 x = range(len(species_data)) 160 y = species_data[indicator] 161 plt.scatter(x, y, label=species) 162 plt.xlabel('Data Point') 163 plt.ylabel(indicator) 164 plt.title(f'{indicator} Comparison') 165 plt.legend() 166 # 显示图表 167 plt.show() 168 169 import pandas as pd 170 import matplotlib.pyplot as plt 171 import numpy as np 172 173 plt.rcParams['font.sans-serif'] = ['SimHei'] 174 # 导入数据集 175 data = pd.read_csv('Cleaned_Tree_Dataset.csv') 176 # 指定要提取的数据指标和Sterile类型 177 indicators = ['AMF', 'EMF', 'Phenolics', 'Lignin', 'NSC'] 178 sterile_types = ['Non-Sterile', 'Sterile'] 179 # 提取所需的数据列和指定的Sterile类型 180 columns = ['Species', 'Sterile'] + indicators 181 subset = data[data['Sterile'].isin(sterile_types)][columns] 182 # 计算每个数据指标的平均值 183 averages = subset.groupby(['Species', 'Sterile']).mean().reset_index() 184 # 创建雷达图 185 num_indicators = len(indicators) 186 # 计算角度 187 angles = np.linspace(0, 2 * np.pi, num_indicators, endpoint=False).tolist() 188 angles += angles[:1] 189 # 设置图表属性 190 plt.figure(figsize=(8, 8)) 191 ax = plt.subplot(111, polar=True) 192 ax.set_xticks(angles[:-1]) 193 ax.set_xticklabels(indicators) 194 ax.set_yticklabels([]) 195 # 绘制雷达图 196 for sterile_type in sterile_types: 197 species_data = averages[averages['Sterile'] == sterile_type] 198 values = species_data[indicators].values.tolist()[0] 199 values += values[:1] 200 ax.plot(angles, values, label=sterile_type) 201 ax.fill(angles, values, alpha=0.25) 202 # 添加图例 203 plt.legend() 204 # 显示图表 205 plt.title('2018年5月-6月各树种在有菌和无菌的数据指标值对比雷达图') 206 plt.show() 207 208 import pandas as pd 209 import matplotlib.pyplot as plt 210 211 plt.rcParams['font.sans-serif'] = ['SimHei'] 212 # 导入数据集 213 data = pd.read_csv('Cleaned_Tree_Dataset.csv') 214 # 输入树种和土壤环境 215 species = input("请输入树种: ") 216 soil = input("请输入土壤环境: ") 217 # 指定要提取的数据指标 218 indicators = ['AMF', 'EMF', 'Phenolics', 'Lignin', 'NSC'] 219 # 提取满足条件的数据 220 subset = data[(data['Species'] == species) & (data['Soil'] == soil)][indicators] 221 # 计算每个数据指标的平均值 222 averages = subset.mean() 223 # 打印输出表格 224 print(f"树种: {species}, 土壤环境: {soil}") 225 print("各个数据指标的平均值:") 226 print(averages) 227 # 绘制折线图 228 plt.figure(figsize=(10, 6)) 229 for indicator in indicators: 230 plt.plot(subset[indicator], label=indicator) 231 plt.xlabel('样本') 232 plt.ylabel('数值') 233 plt.title(f"{species}在{soil}条件下各个数据指标的折线图") 234 plt.legend() 235 plt.show() 236 237 238 import pandas as pd 239 import matplotlib.pyplot as plt 240 241 plt.rcParams['font.sans-serif'] = ['SimHei'] 242 # 导入数据集 243 data = pd.read_csv('Cleaned_Tree_Dataset.csv') 244 # 分类提取Sterile的Non-Sterile和Sterile数据 245 sterile_data = data[data['Sterile'] == 'Sterile'] 246 non_sterile_data = data[data['Sterile'] == 'Non-Sterile'] 247 # 获取所有树种的唯一值 248 species = data['Species'].unique() 249 # 计算每个树种的数据指标平均值 250 sterile_averages = sterile_data.groupby('Species')[['AMF', 'EMF', 'Phenolics', 'Lignin', 'NSC']].mean() 251 non_sterile_averages = non_sterile_data.groupby('Species')[['AMF', 'EMF', 'Phenolics', 'Lignin', 'NSC']].mean() 252 # 设置柱状图的宽度和位置 253 bar_width = 0.35 254 index = range(len(species)) 255 opacity = 0.8 256 # 创建图表对象 257 fig, ax = plt.subplots() 258 # 绘制Sterile数据的柱状图 259 sterile_bars = ax.bar(index, sterile_averages.loc[species]['AMF'], bar_width, alpha=opacity, color='b', label='Sterile') 260 # 绘制Non-Sterile数据的柱状图 261 non_sterile_bars = ax.bar([i + bar_width for i in index], non_sterile_averages.loc[species]['AMF'], bar_width, alpha=opacity, color='r', label='Non-Sterile') 262 # 设置图表标题和x轴标签 263 ax.set_title('不同树种的AMF数据对比') 264 ax.set_xlabel('树种') 265 ax.set_xticks([i + bar_width/2 for i in index]) 266 ax.set_xticklabels(species, rotation=45) 267 # 设置y轴标签 268 ax.set_ylabel('平均值') 269 # 添加图例 270 ax.legend() 271 # 显示图表 272 plt.tight_layout() 273 plt.show() 274 # 生成不同树种的EMF、Phenolics、Lignin、NSC数据对比柱状图 275 fig, ax = plt.subplots(2, 2, figsize=(10, 8)) 276 # 绘制EMF数据对比柱状图 277 emf_bars = ax[0, 0].bar(index, sterile_averages.loc[species]['EMF'], bar_width, alpha=opacity, color='b', label='Sterile') 278 ax[0, 0].bar([i + bar_width for i in index], non_sterile_averages.loc[species]['EMF'], bar_width, alpha=opacity, color='r', label='Non-Sterile') 279 ax[0, 0].set_title('EMF数据对比') 280 ax[0, 0].set_xlabel('树种') 281 ax[0, 0].set_ylabel('平均值') 282 ax[0, 0].set_xticks([i + bar_width/2 for i in index]) 283 ax[0, 0].set_xticklabels(species, rotation=45) 284 ax[0, 0].legend() 285 # 绘制Phenolics数据对比柱状图 286 phenolics_bars = ax[0, 1].bar(index, sterile_averages.loc[species]['Phenolics'], bar_width, alpha=opacity, color='b', label='Sterile') 287 ax[0, 1].bar([i + bar_width for i in index], non_sterile_averages.loc[species]['Phenolics'], bar_width, alpha=opacity, color='r', label='Non-Sterile') 288 ax[0, 1].set_title('Phenolics数据对比') 289 ax[0, 1].set_xlabel('树种') 290 ax[0, 1].set_ylabel('平均值') 291 ax[0, 1].set_xticks([i + bar_width/2 for i in index]) 292 ax[0, 1].set_xticklabels(species, rotation=45) 293 ax[0, 1].legend() 294 # 绘制Lignin数据对比柱状图 295 lignin_bars = ax[1, 0].bar(index, sterile_averages.loc[species]['Lignin'], bar_width, alpha=opacity, color='b', label='Sterile') 296 ax[1, 0].bar([i + bar_width for i in index], non_sterile_averages.loc[species]['Lignin'], bar_width, alpha=opacity, color='r', label='Non-Sterile') 297 ax[1, 0].set_title('Lignin数据对比') 298 ax[1, 0].set_xlabel('树种') 299 ax[1, 0].set_ylabel('平均值') 300 ax[1, 0].set_xticks([i + bar_width/2 for i in index]) 301 ax[1, 0].set_xticklabels(species, rotation=45) 302 ax[1, 0].legend() 303 # 绘制NSC数据对比柱状图 304 nsc_bars = ax[1, 1].bar(index, sterile_averages.loc[species]['NSC'], bar_width, alpha=opacity, color='b', label='Sterile') 305 ax[1, 1].bar([i + bar_width for i in index], non_sterile_averages.loc[species]['NSC'], bar_width, alpha=opacity, color='r', label='Non-Sterile') 306 ax[1, 1].set_title('NSC数据对比') 307 ax[1, 1].set_xlabel('树种') 308 ax[1, 1].set_ylabel('平均值') 309 ax[1, 1].set_xticks([i + bar_width/2 for i in index]) 310 ax[1, 1].set_xticklabels(species, rotation=45) 311 ax[1, 1].legend() 312 # 调整子图之间的间距 313 plt.tight_layout() 314 315 # 显示图表 316 plt.show() 317 318 319 import pandas as pd 320 import matplotlib.pyplot as plt 321 from mpl_toolkits.mplot3d import Axes3D 322 323 # 导入数据集 324 data = pd.read_csv("Cleaned_Tree_Dataset.csv") 325 326 # 提取特征和目标变量 327 features = data[['AMF', 'EMF', 'Phenolics']] 328 target = data['Species'] 329 330 # 创建字典以存储每个树种的数据 331 species_data = {} 332 333 # 根据树种分组,将数据存储到字典中 334 for species in target.unique(): 335 species_data[species] = features[target == species] 336 337 # 创建三维视图 338 fig = plt.figure(figsize=(10, 8)) 339 ax = fig.add_subplot(111, projection='3d') 340 341 # 定义不同树种的颜色和标记样式 342 colors = ['r', 'g', 'b', 'c', 'm', 'y', 'k'] 343 markers = ['o', 's', '^', 'v', 'D', 'p', '*'] 344 345 # 绘制每个树种的三维视图 346 for i, species in enumerate(species_data): 347 ax.scatter(species_data[species]['AMF'], species_data[species]['EMF'], species_data[species]['Phenolics'], 348 c=colors[i], marker=markers[i], label=species) 349 350 # 设置坐标轴标签 351 ax.set_xlabel('AMF') 352 ax.set_ylabel('EMF') 353 ax.set_zlabel('Phenolics') 354 355 # 添加图例 356 ax.legend() 357 358 # 显示图形 359 plt.show() 360 361 362 import pandas as pd 363 from sklearn.model_selection import train_test_split 364 from sklearn.linear_model import LogisticRegression 365 from sklearn.metrics import accuracy_score 366 data = pd.read_csv("Cleaned_Tree_Dataset.csv") 367 features = data[['AMF', 'EMF', 'Phenolics', 'Lignin', 'NSC']] 368 target = data['Species'] 369 370 X_train, X_test, y_train, y_test = train_test_split(features, target, test_size=0.2, random_state=42) 371 model = LogisticRegression() 372 model.fit(X_train, y_train) 373 predictions = model.predict(X_test) 374 accuracy = accuracy_score(y_test, predictions) 375 print("基准模型准确率:", accuracy) 376 num_iterations = 39 377 accuracies = [] 378 379 for i in range(num_iterations): 380 X_train, X_test, y_train, y_test = train_test_split(features, target, test_size=0.2, random_state=i) 381 model = LogisticRegression() 382 model.fit(X_train, y_train) 383 predictions = model.predict(X_test) 384 accuracy = accuracy_score(y_test, predictions) 385 accuracies.append(accuracy) 386 387 # 输出每次遍历的准确率 388 for i, accuracy in enumerate(accuracies): 389 print("遍历", i+1, "准确率:", accuracy) 390 391 392 import pandas as pd 393 from sklearn.model_selection import train_test_split 394 from sklearn.linear_model import LogisticRegression 395 from sklearn.metrics import classification_report 396 397 # 导入数据集 398 data = pd.read_csv("Cleaned_Tree_Dataset.csv") 399 400 # 提取特征和目标变量 401 features = data[['AMF', 'EMF', 'Phenolics', 'Lignin', 'NSC']] 402 target = data['Species'] 403 404 # 数据集划分为训练集和测试集 405 X_train, X_test, y_train, y_test = train_test_split(features, target, test_size=0.2, random_state=42) 406 407 # 构建模型(逻辑回归) 408 model = LogisticRegression() 409 410 # 模型训练 411 model.fit(X_train, y_train) 412 413 # 预测 414 predictions = model.predict(X_test) 415 416 # 输出预测结果 417 report = classification_report(y_test, predictions) 418 print(report)
六、大数据分析总结
除了不同演替森林之间的差异外,我发现相同的乔木科,在不同的属种上,所需的生存影响因子元素是不同的。例如植物吸收,微生物动员,酶矿化和土壤氧化还原。吸收植物可能比其他过程更重要,因为植物在生长季节需要大量的磷,因为在此季节它们会快速积累生物量。土壤氧化还原影响铁在湿润热带森林中对磷的吸附能力,并影响磷的吸附和溶解度,造成(丛枝菌根真菌(AMF)和外生菌根真菌(EMF)的数值波动,再加上无菌环境下的无机物丰富,树木所需的生长微生物得到了充分的营养补充,造成数据指标快速增长。
总而言之,我研究土壤、菌类、环境对乔木植物的生存影响数据的预测模型,这可能会增进土壤、菌类、环境对乔木植物的生存影响有了新的认识和亚热带森林巨大生产力和高生物多样性水平的理解。需要进一步的研究来阐明在土壤、菌类、环境对生存影响因子相互作用和各自和协同作用,特别是进行一些可操作的实验以找到更可靠的证据。
在实验中,我对数据集划分:将数据集划分为训练集和测试集,以确保模型在未知数据上的泛化能力。模型优化:根据评估结果对模型进行优化,以提高其性能。局限性分析:分析结果的局限性。有跟深一步的了解和运用。
感谢鄂大伟老师,给我运用Python大数据实践分析报告的机会,如有错误,请老师帮忙批评指正。






【推荐】国内首个AI IDE,深度理解中文开发场景,立即下载体验Trae
【推荐】编程新体验,更懂你的AI,立即体验豆包MarsCode编程助手
【推荐】抖音旗下AI助手豆包,你的智能百科全书,全免费不限次数
【推荐】轻量又高性能的 SSH 工具 IShell:AI 加持,快人一步
· 分享4款.NET开源、免费、实用的商城系统
· 全程不用写代码,我用AI程序员写了一个飞机大战
· MongoDB 8.0这个新功能碉堡了,比商业数据库还牛
· 白话解读 Dapr 1.15:你的「微服务管家」又秀新绝活了
· 记一次.NET内存居高不下排查解决与启示