蛋白质序列位置特异性矩阵(PSSM)的获取
一、下载安装ncbi-blast(一定要是最新版本,在这里吃了苦头)
下载地址:https://ftp.ncbi.nlm.nih.gov/blast/executables/blast+/LATEST/
安装blast:按照提示安装即可
这里我们要用到主要的文件为:rpsblast.exe
二、下载对比数据库
下载地址:ftp://ftp.ncbi.nlm.nih.gov/blast/db/
这里可以选择nr库和swissprot库,因为nr库太大,下载时间长,这里我选择使用swissprot库,下载到本地之后,不需要做成本地数据库,如果需要做成本地数据库,指令为:makeblastdb -in swissprot -dbtype prot -title "swissprot" -out swissprot(这里要用cmd命令切换地址到下载的blast/bin目录下运行该命令)
三、获得PSSM文件
将自己需要的fasta序列准备好,同样要在blast/bin目录下运行:psiblast.exe -db swissprot -query 0.txt -evalue 0.001 -num_iterations 3 -out_ascii_pssm 0.pssm

这里:0.txt文件就是我准备查询的fasta序列, 0.pssm就是输出的pssm文件其它参数都设置为常用数值,需要了解,可查阅其它资料
运行结束得到0.pssm文件,文件内容如下:
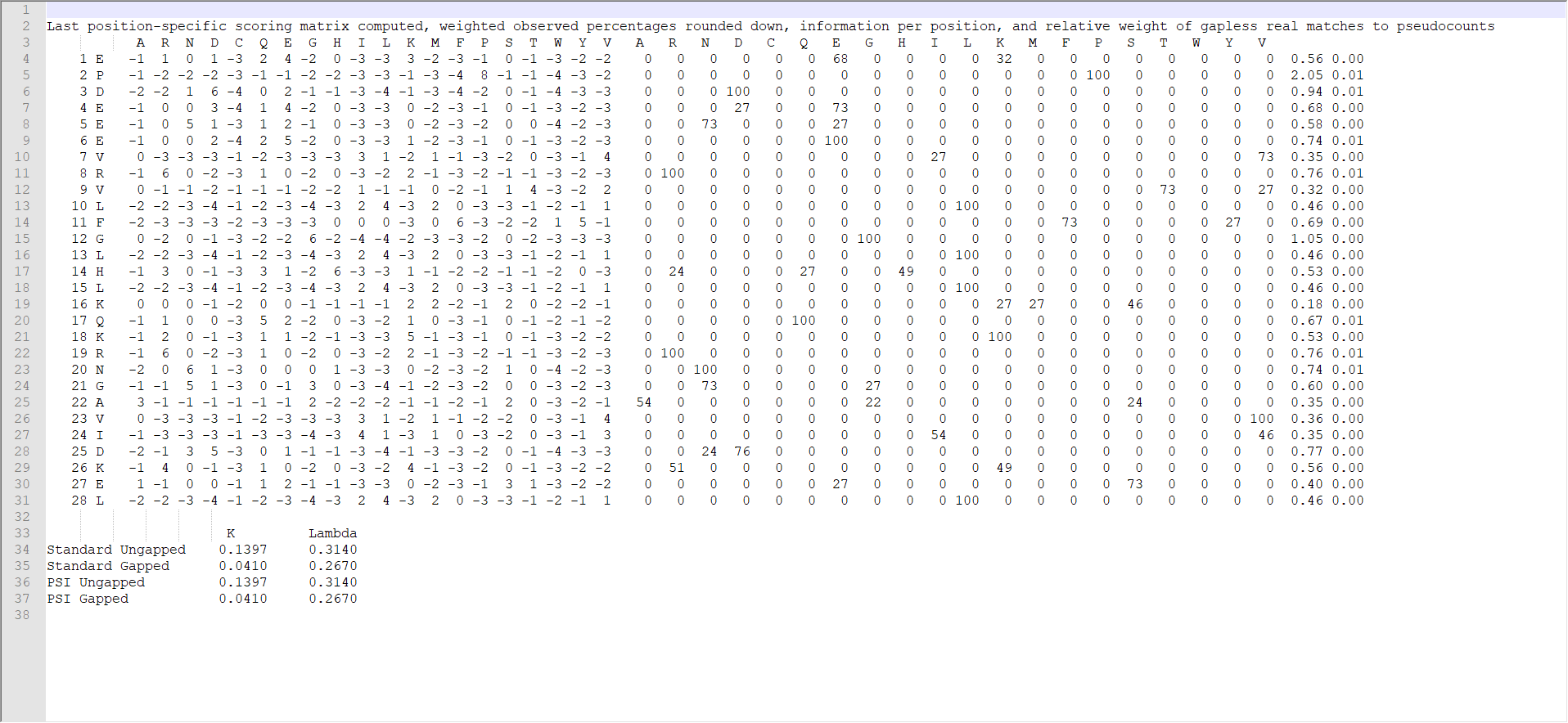
这里我们需要的PSSM矩阵就为L*20(L为所使用的fasta序列的长度,20个氨基酸),如下所示:
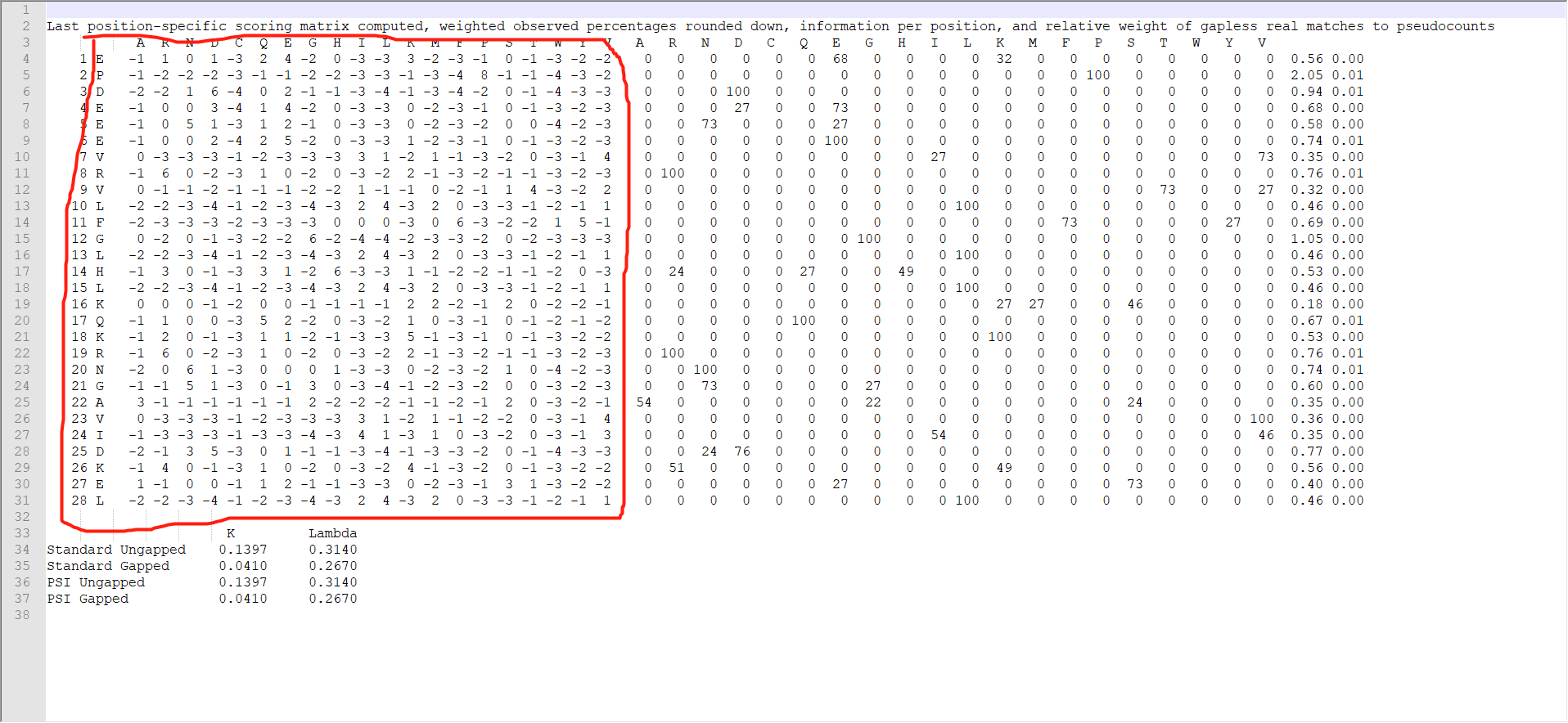
这就是一个fasta序列的位置特异性矩阵(PSSM)的获得。
注意:该指令是一个fasta序列的pssm矩阵,如果批量获取,编写程序,让其批量运行并保存即可。
前期所有fasta序列的处理:https://www.cnblogs.com/cong3Z/p/12775480.html



 浙公网安备 33010602011771号
浙公网安备 33010602011771号