字符串匹配算法在DNA序列查找中的应用
atcaatgatcaacgtaagcttctaagcatgatcaaggtgctcacacagtttatccacaacctgagtggatgacatcaagataggtcgttgtatctccttcctctcgtactctcatgaccacggaaagatgatcaagagaggatgatttcttggccatatcgcaatgaatacttgtgacttgtgcttccaattgacatcttcagcgccatattgcgctggccaaggtgacggagcgggattacgaaagcatgatcatggctgttgttctgtttatcttgttttgactgagacttgttaggatagacggtttttcatcactgactagccaaagccttactctgcctgacatcgaccgtaaattgataatgaatttacatgcttccgcgacgatttacctcttgatcatcgatccgattgaagatcttcaattgttaattctcttgcctcgactcatagccatgatgagctcttgatcatgtttccttaaccctctattttttacggaagaatgatcaagctgctgctcttgatcatcgtttc
以上500字母组成的字符串是从一个大肠杆菌基因组中截取出来的部分序列。
我们能从这段基因中知道什么?
什么也不能知道,因为这只是某一个大肠杆菌的基因序列并不具有普遍性。
我们要做的是从这一段基因中找出隐藏信息,然后在在大量个体中验证我们的猜想。
那这一段基因中有什么隐藏信息呢?
DNA上编码区三个碱基对编码一个氨基酸
如果我们发现了这一段DNA中有大量重复出现的三碱基序列,那么我们便可以针对这个碱基序列展开研究
当然不仅仅是三碱基,数据表明大肠杆菌中有大部分功能蛋白由9个碱基编码
这就引出了一类问题等待我们利用算法知识解决
→寻找字符串中频率最高的短重复序列(字符串匹配问题下的一个分支)
问题抽象:
找出给定字符串j中出现频数最高的长度为k的短序列(k<j)
输入:字符串j,长度k
输出:最频繁短序列q,出现次数n
方法一:朴素的字符串匹配算法
方法描述:
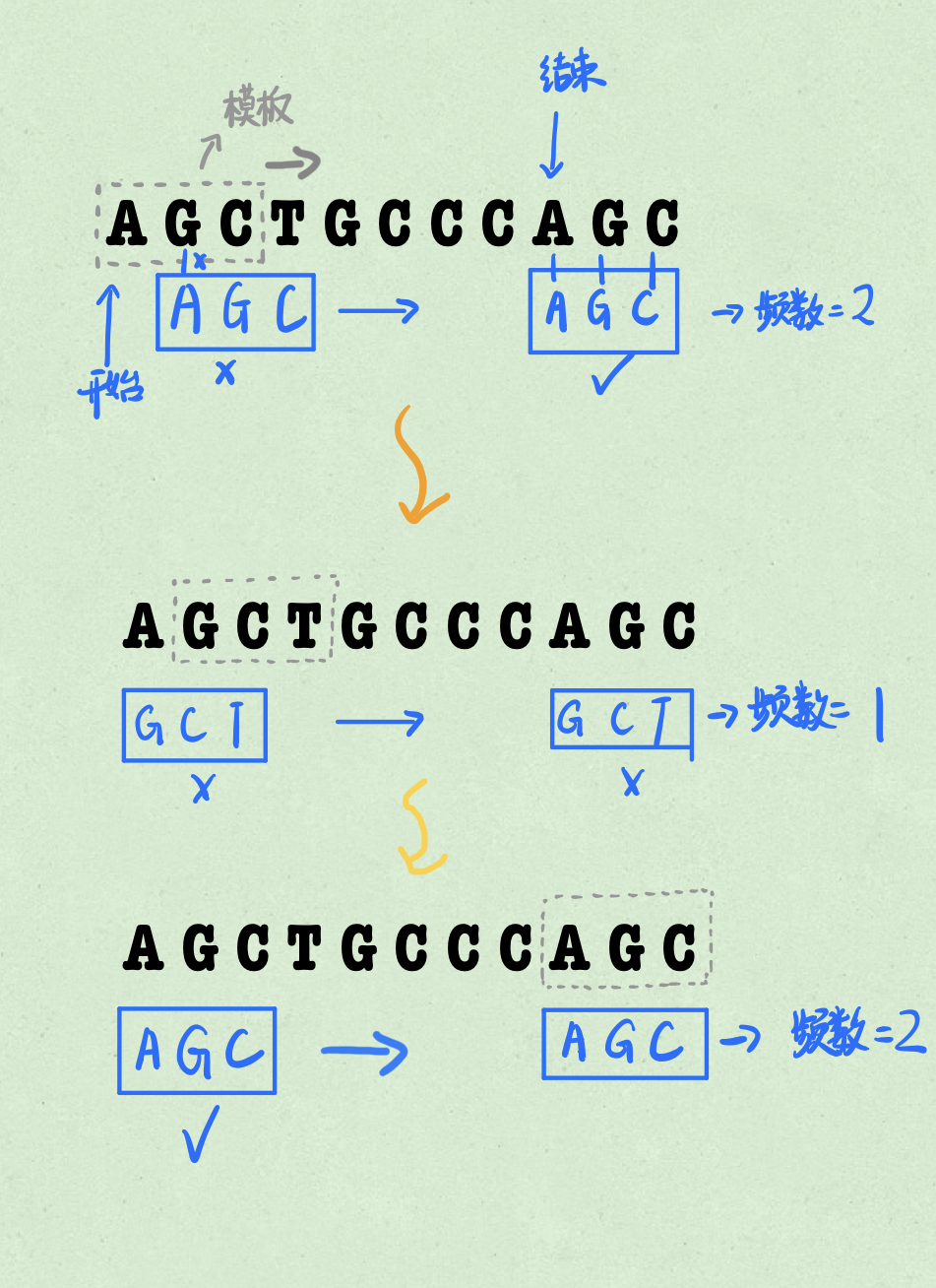
思路很简单,设定模板阅读框(灰色)和校验阅读框(蓝色),长度都为K(假设为3)
模板阅读框读取一个序列作为模板后,校验阅读框从头阅读一遍,一次移动一个字符,如果校验框内容和该位置字符串匹配,则对应字符串频度加1
这里我用了频度数组来储存每一个字符串的频度,注意这里包括了字符串中的重复序列。
频度数组也很好理解:
![]()
数组长度为模板总数→(字符串长度-K+1)
数组中第i个元素即为字符串中以第i个字符打头,长度为K的短序列出现的频数
如上图所示
ps:代码中应用了一个 in_window函数,将指针p后的k个字符读入字符串s中,便于改变校验框,模板框的内容
1 #include <stdio.h> 2 #include <string.h> 3 4 void in_window(char *source,int n,char *des);//将source指向的字符后的n个字符读入des中 5 void Frequent_word(char *p,int k);//频数统计函数 6 7 int main(int argc, char const *argv[]) 8 { 9 printf("Enter the DNA Code\n"); 10 char dna[9999]; 11 scanf("%s",dna); 12 int n; 13 printf("Enter the size of the array\n"); 14 scanf("%d",&n); 15 Frequent_word(dna,n); 16 17 18 return 0; 19 } 20 21 22 void in_window(char *source,int n,char *des){ 23 int i; 24 for (i = 0; i < n; ++i) 25 { 26 *(des+i)=*(source+i); 27 } 28 *(des+i)='\0';//关键是要加/0将其补成字符串而不是字符数组以利用字符串函数进行操作 29 } 30 31 void Frequent_word(char *p,int k){ 32 int frequent_array[strlen(p)-k+1]; 33 //频度数组→储存每个长度为K的短重复序列的频数→共有length-k+1个 34 for (int q = 0; q < strlen(p)-k+1; ++q) 35 { 36 frequent_array[q]=0; 37 } 38 39 //填充频度数组 40 int i; 41 int max_number=0,tag=0;//用来储存最大频数和子序列位置 42 43 char model[k+1]; 44 char sample[k+1];//校验框 45 for (i = 0; i < strlen(p)-k+1; ++i)//外层循环,每一次扫描全DNA时读取框的模板选择 46 { 47 in_window(p+i,k,model); 48 49 for (int j = 0; j < strlen(p)-k+1; ++j)//判断每一个模板的频数 50 { 51 in_window(p+j,k,sample); 52 if(!strcmp(sample,model))//扫描框等于模板框时频数对应加一 53 frequent_array[i]++; 54 } 55 56 if (frequent_array[i]>max_number) 57 {max_number=frequent_array[i]; 58 tag=i;}//储存最大信息 59 } 60 61 char final[k+1]; 62 final[k]='\0'; 63 in_window(p+tag,k,final); 64 printf("The most frequent array at the size of %d is\n%s\nappear %d times",k,final,max_number ); 65 66 }
实验结果:
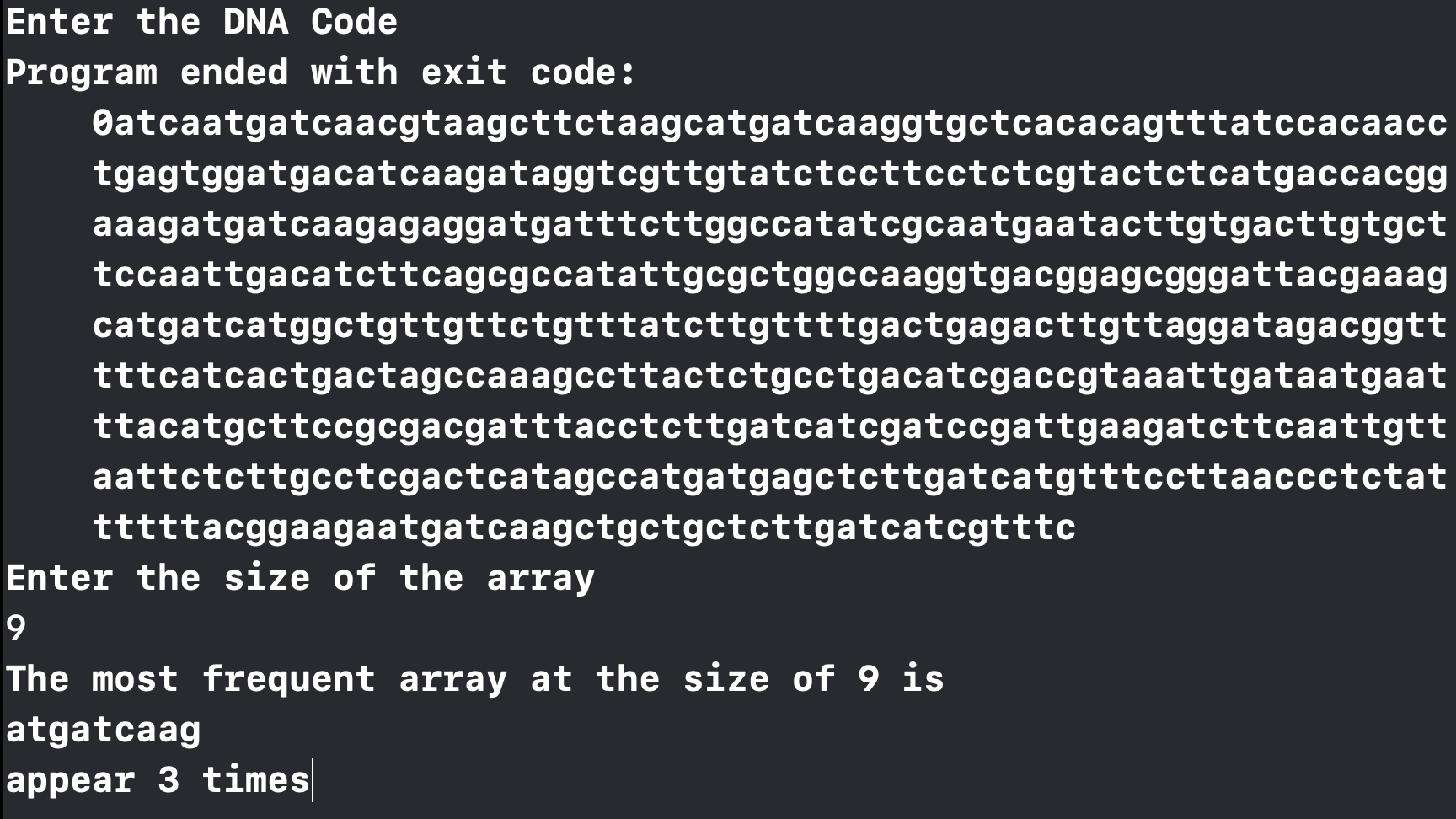
这一种方法有显而易见的缺点,由于嵌套循环,时间复杂度随着字符串长度的增长自然达到了指数级。
方法二:KMP算法
稍后更!



 浙公网安备 33010602011771号
浙公网安备 33010602011771号