11.分类与监督学习,朴素贝叶斯分类算法
1.理解分类与监督学习、聚类与无监督学习。
简述分类与聚类的联系与区别。
聚类分析是研究如何在没有训练的条件下把样本划分为若干类。
在分类中,已知存在哪些类,即对于目标数据库中存在哪些类是知道的,要做的就是将每一条记录分别属于哪一类标记出来。
联系:聚类需要解决的问题是将已给定的若干无标记的模式聚集起来使之成为有意义的聚类,聚类是在预先不知道目标数据库到底有多少类的情况下,希望将所有的记录组成不同的类或者说聚类,并且使得在这种分类情况下,以某种度量(例如:距离)为标准的相似性,在同一聚类之间最小化,而在不同聚类之间最大化。
区别:与分类不同,无监督学习不依赖预先定义的类或带类标记的训练实例,需要由聚类学习算法自动确定标记,而分类学习的实例或数据样本有类别标记。
简述什么是监督学习与无监督学习。
无监督学习:输入数据没有被标记,也没有确定的结果。样本数据类别未知,需要根据样本间的相似性对样本集进行分类(聚类,clustering)试图使类内差距最小化,类间差距最大化。通俗点将就是实际应用中,不少情况下无法预先知道样本的标签,也就是说没有训练样本对应的类别,因而只能从原先没有样本标签的样本集开始学习分类器设计。
监督学习:从给定的训练数据集中学习出一个函数(模型参数),当新的数据到来时,可以根据这个函数预测结果。监督学习的训练集要求包括输入输出,也可以说是特征和目标。训练集中的目标是由人标注的。监督学习就是最常见的分类(注意和聚类区分)问题,通过已有的训练样本(即已知数据及其对应的输出)去训练得到一个最优模型(这个模型属于某个函数的集合,最优表示某个评价准则下是最佳的),再利用这个模型将所有的输入映射为相应的输出,对输出进行简单的判断从而实现分类的目的。也就具有了对未知数据分类的能力。监督学习的目标往往是让计算机去学习我们已经创建好的分类系统(模型)。
2.朴素贝叶斯分类算法 实例
利用关于心脏病患者的临床历史数据集,建立朴素贝叶斯心脏病分类模型。
有六个分类变量(分类因子):性别,年龄、KILLP评分、饮酒、吸烟、住院天数
目标分类变量疾病:
–心梗
–不稳定性心绞痛
新的实例:–(性别=‘男’,年龄<70, KILLP=‘I',饮酒=‘是’,吸烟≈‘是”,住院天数<7)
最可能是哪个疾病?
上传手工演算过程。

|
|
性别 |
年龄 |
KILLP |
饮酒 |
吸烟 |
住院天数 |
疾病 |
|
1 |
男 |
>80 |
1 |
是 |
是 |
7-14 |
心梗 |
|
2 |
女 |
70-80 |
2 |
否 |
是 |
<7 |
心梗 |
|
3 |
女 |
70-81 |
1 |
否 |
否 |
<7 |
不稳定性心绞痛 |
|
4 |
女 |
<70 |
1 |
否 |
是 |
>14 |
心梗 |
|
5 |
男 |
70-80 |
2 |
是 |
是 |
7-14 |
心梗 |
|
6 |
女 |
>80 |
2 |
否 |
否 |
7-14 |
心梗 |
|
7 |
男 |
70-80 |
1 |
否 |
否 |
7-14 |
心梗 |
|
8 |
女 |
70-80 |
2 |
否 |
否 |
7-14 |
心梗 |
|
9 |
女 |
70-80 |
1 |
否 |
否 |
<7 |
心梗 |
|
10 |
男 |
<70 |
1 |
否 |
否 |
7-14 |
心梗 |
|
11 |
女 |
>80 |
3 |
否 |
是 |
<7 |
心梗 |
|
12 |
女 |
70-80 |
1 |
否 |
是 |
7-14 |
心梗 |
|
13 |
女 |
>80 |
3 |
否 |
是 |
7-14 |
不稳定性心绞痛 |
|
14 |
男 |
70-80 |
3 |
是 |
是 |
>14 |
不稳定性心绞痛 |
|
15 |
女 |
<70 |
3 |
否 |
否 |
<7 |
心梗 |
|
16 |
男 |
70-80 |
1 |
否 |
否 |
>14 |
心梗 |
|
17 |
男 |
<70 |
1 |
是 |
是 |
7-14 |
心梗 |
|
18 |
女 |
70-80 |
1 |
否 |
否 |
>14 |
心梗 |
|
19 |
男 |
70-80 |
2 |
否 |
否 |
7-14 |
心梗 |
|
20 |
女 |
<70 |
3 |
否 |
否 |
<7 |
不稳定性心绞痛 |
3.使用朴素贝叶斯模型对iris数据集进行花分类。
尝试使用3种不同类型的朴素贝叶斯:
- 高斯分布型
- 多项式型
- 伯努利型
并使用sklearn.model_selection.cross_val_score(),对各模型进行交叉验证。
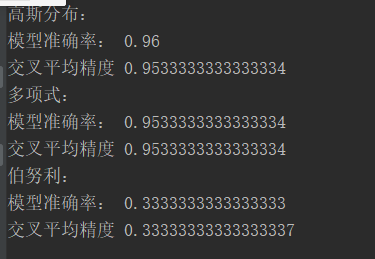
from sklearn.datasets import load_iris from sklearn.naive_bayes import GaussianNB, MultinomialNB, BernoulliNB from sklearn.model_selection import cross_val_score iris = load_iris() # 高斯分布型 gnb = GaussianNB() # 导入模型 gnb.fit(iris.data, iris.target) # 训练模型 y_gnb = gnb.predict(iris.data) # 预 测 GNB_scores = cross_val_score(gnb, iris.data, iris.target, cv=10) # 交叉验证 # print("高斯分布:") # print("模型准确率:", sum(y_gnb == iris.target)/len(iris.data)) # print("交叉平均精度", GNB_scores.mean()) # 多项式 mnb = MultinomialNB() mnb.fit(iris.data, iris.target) y_mnb = mnb.predict(iris.data) MNB_scores = cross_val_score(mnb, iris.data, iris.target, cv=10) # print("多项式:") # print("模型准确率:", sum(y_mnb == iris.target)/len(iris.data)) # print("交叉平均精度", MNB_scores.mean()) # 伯努利型 bnl = BernoulliNB() bnl.fit(iris.data, iris.target) y_bnl = bnl.predict(iris.data) BNL_scores = cross_val_score(bnl, iris.data, iris.target, cv=10) print("高斯分布:") print("模型准确率:", sum(y_gnb == iris.target)/len(iris.data)) print("交叉平均精度", GNB_scores.mean()) print("多项式:") print("模型准确率:", sum(y_mnb == iris.target)/len(iris.data)) print("交叉平均精度", MNB_scores.mean()) print("伯努利:") print("模型准确率:", sum(y_bnl == iris.target)/len(iris.data)) print("交叉平均精度", BNL_scores.mean())