使用vcftools或者gcta计算群体间固定指数(Fixation index,FST)
下列所用到的数据均为千人基因组数据库
1、通过vcftools计算FST
命令行如下:
./vcftools --vcf input_data.vcf --weir-fst-pop population_1.txt --weir-fst-pop population_2.txt --out pop1_vs_pop2
其中,input_data.vcf就是输入的vcf格式
population_1.txt的格式如下:
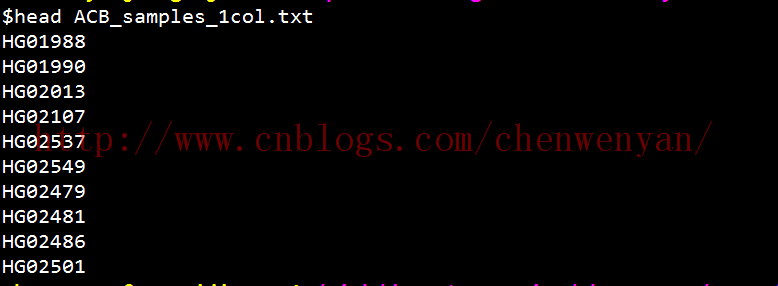
population_2.txt的格式同population_1.txt,只有一列sample名字的信息。
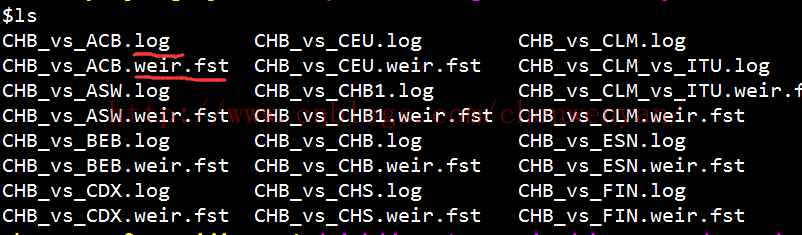
计算之后,会得到两个文件格式,分别为.log和.fst,如图下所示:
2、通过gcta计算FST
命令行如下:
gcta64 --bfile test --fst --sub-popu subpopu.txt --out test
其中,bfile文件W为plink二进制格式文件,后缀分别为.bed、.bim、.fam
subpopu.txt的输入格式如图下所示:
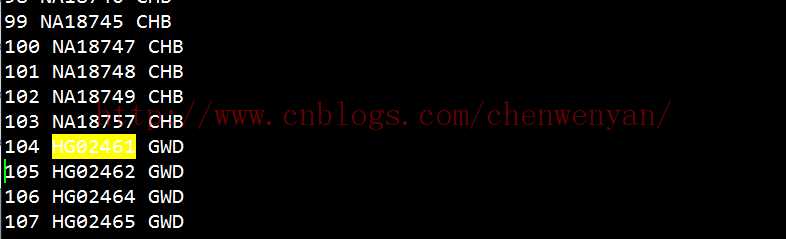
解释下subpopu.txt这三列的意思。第一列为样本数,第二列为样本的ID,第三列为每个样本对应的population(如CHB、GWD)
其中,CHB为其中一个population,GWD为另一个population。
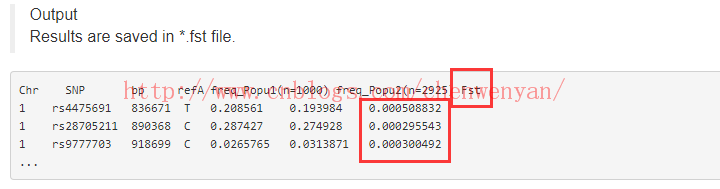
得到的结果文件为.fst格式,如图下所示:
参考链接:
http://cnsgenomics.com/software/gcta/#Fst
http://vcftools.sourceforge.net/documentation.html
本文来自博客园,作者:橙子牛奶糖(陈文燕),转载请注明原文链接:https://www.cnblogs.com/chenwenyan/p/8572537.html




