plink, vcftool计算等位基因频率(allele frequency,vcf)
计算等位基因频率有两种方式,第一种用vcftool计算:
/path/to/vcftools --vcf file.vcf --freq --chr 1 --out filefreq
很简单的一个命令行,file.vcf指的是你要输入的vcf文件,--freq表示计算等位基因频率,--chr后面的1表示你要计算的区域在1号染色体,当然,你也可以选择你想计算的染色体区域,filefreq指的是输出的文件名。
结果如下图所示:
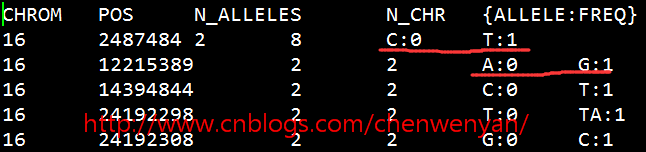
第二种用plink计算:
/path/to/plink-1.07-x86_64/plink --noweb --bfile file --freq --out filefrquency
效果如下图所示:

.frq (basic allele frequency report)
Produced by --freq. Valid input for --read-freq.
A text file with a header line, and then one line per variant with the following six fields:
|
CHR |
Chromosome code |
|
SNP |
Variant identifier |
|
A1 |
Allele 1 (usually minor) |
|
A2 |
Allele 2 (usually major) |
|
MAF |
Allele 1 frequency |
|
NCHROBS |
Number of allele observations |
本文来自博客园,作者:橙子牛奶糖(陈文燕),转载请注明原文链接:https://www.cnblogs.com/chenwenyan/p/7565619.html


 浙公网安备 33010602011771号
浙公网安备 33010602011771号