本周最新文献速递20211226
本周最新文献速递20211226
一、精细解读文献 一
文献题目: Finding genetically-supported drug targets for Parkinson’s disease using Mendelian randomization of the druggable genome
不想看英文题目: 孟德尔随机化鉴定帕金森病的药物靶点
杂志和影响因子: Nat Commun (IF: 14.92; Q1)
研究意义: 帕金森病(PD)属于神经退行性运动障碍疾病,由于药物靶点识别和验证效率低下,目前没有改善该病的治疗方法。在此,作者使用孟德尔随机化(MR)研究 3,000 多个药物蛋白基因,并预测它们作为帕金森病药物靶点的功效;
结论:
- 整合了血液和脑组织的 eQTL 数据以及全基因组关联分析( GWAS )荟萃分析数据。血液和脑组织 eQTL 中分别包含 2786 和 2448 个药物编码基因;

- GWAS 荟萃分析数据包括 13,708 名 PD 患者和 95,282 名对照。在发现队列中,通过 MR 分析发现 31 个基因(11 个在血液中;17 个在脑组织中;三个同时在血液和脑组织中)的表达水平与 PD 风险显着相关;
- 采用重复队列(包括 8036 名 PD 患者和 5803 名对照)复现 31 个基因的结果,发现 12 个基因(BST1、CD38、CHRNB1、CTSB、GPNMB、LGALS3、MAPT、MMRN1、NDUFAF2、PIGF、VKORC1 和 WNT3)在重复队列达到显著水平(FDR<0.05),三个基因(ACVR2A、HSD3B7和MAP3K12)达到名义上的显著水平(P<0.05)。GPNMB 和HSD3B7 在血液和脑组织中均达到显着水平。15 个预防 PD 的潜在药物靶点中,6 个在 PD 的 GWAS 荟萃分析中被报道过,三个已获批准或在临床阶段;
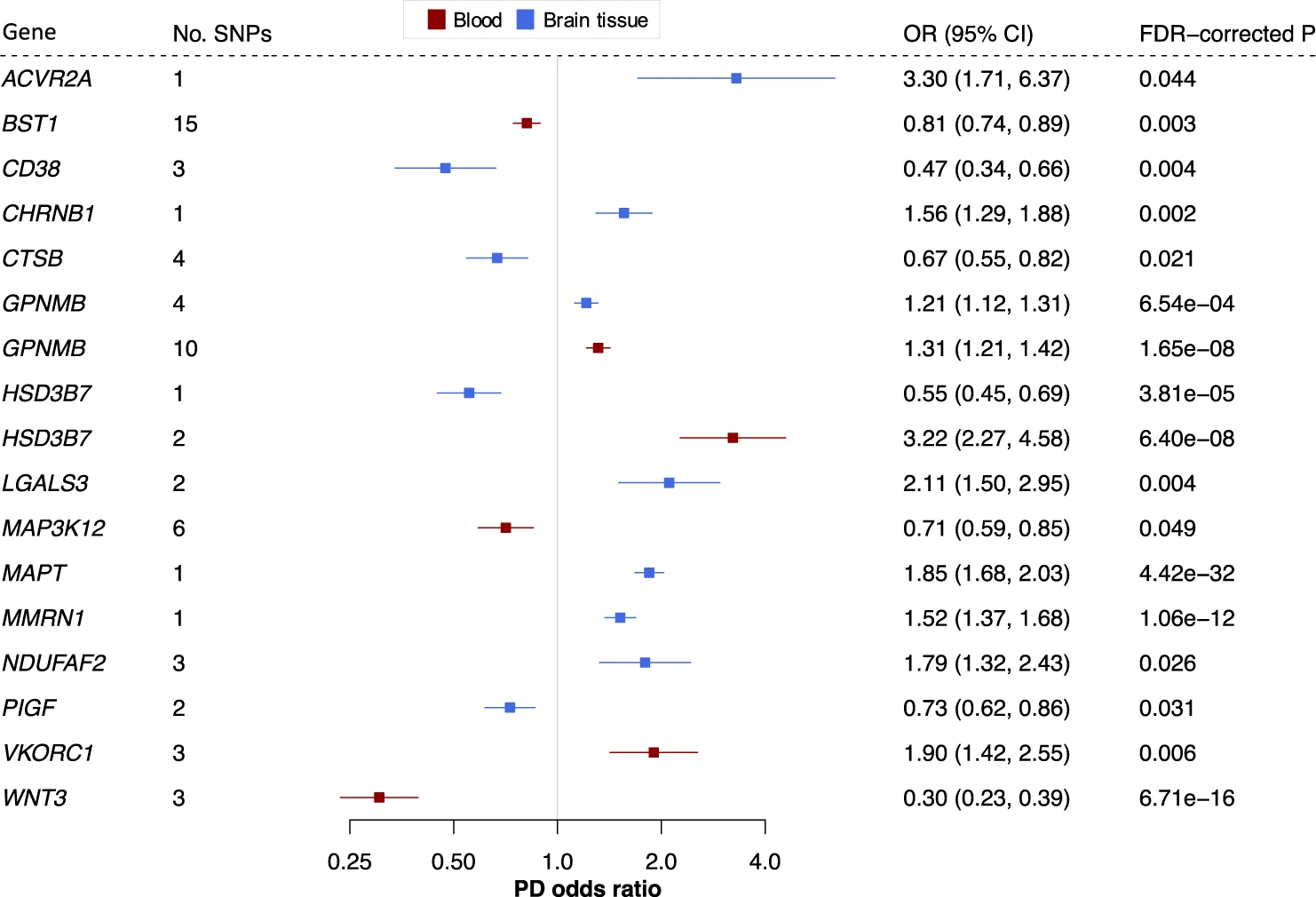
- 对 15 个药物蛋白基因的 eQTL 和 PD GWAS 汇总数据进行共定位,发现 BST1、CD38、GPNMB、HSD3B7、MAPT、MMRN1、VKORC1 和 WNT3 具有强烈的共定位信号。另一项独立的 eQTL 队列同样发现 CD38 和 GPNMB 与 PD 风险位点共定位,说明结合 MR 和共定位分析可有效鉴定 PD 的药物靶点;
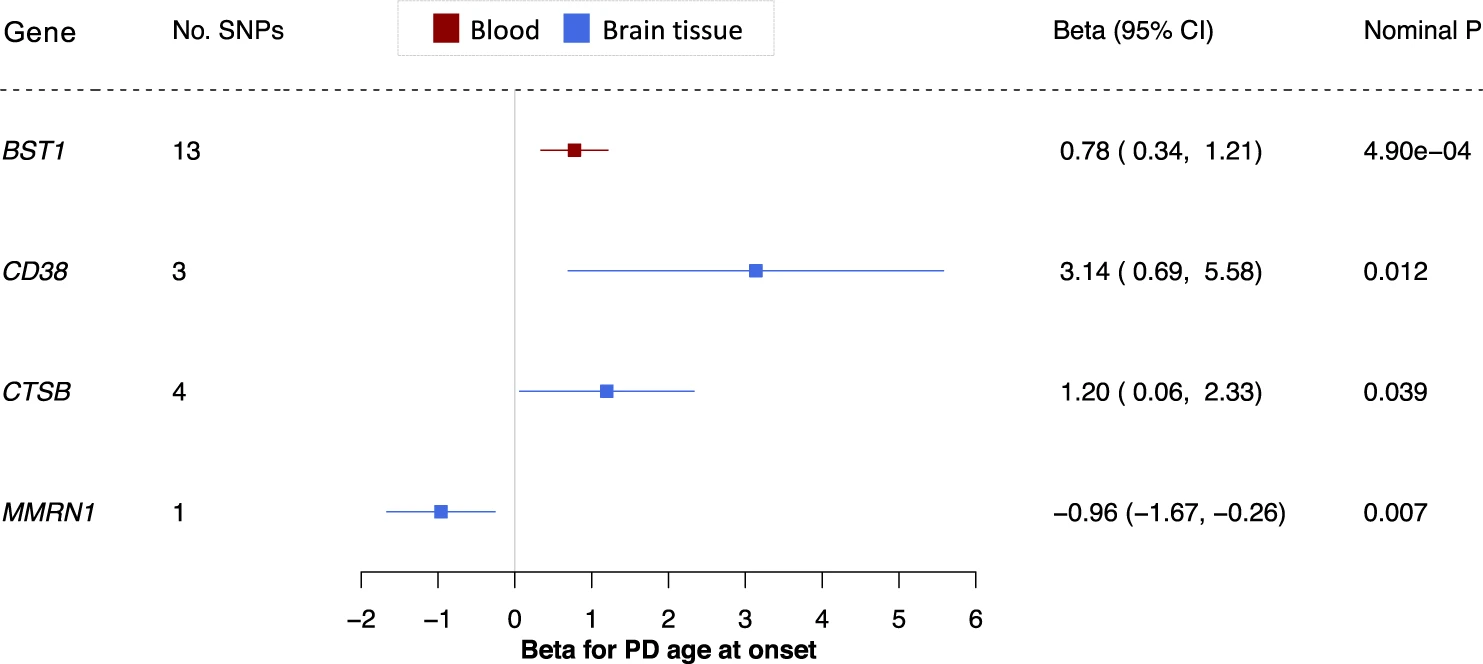
- 研究发现 PD 的遗传风险与 PD 发病年龄相关,为了探究 PD 的风险位点是否也影响 PD 发病年龄,作者对四个 PD 药物靶标基因(CD38、CTSB、GPNMB 和 MAP3K12)进行 PD 发病年龄的 MR 分析,发现 PD 的三个药物靶标基因表达水平与发病年龄正相关,一个基因表达水平与发病年龄负相关;
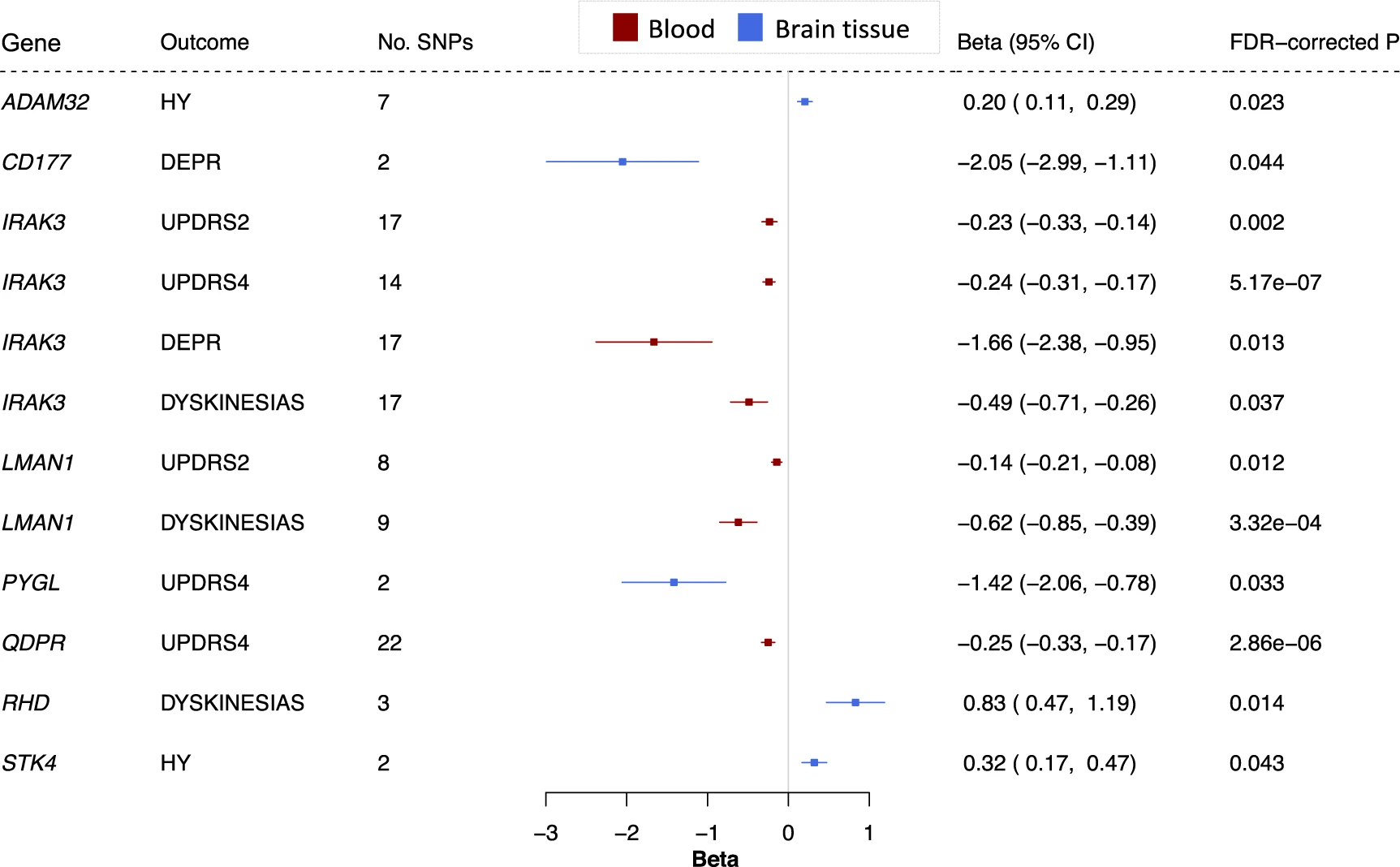
- 为了分析药物靶点是否与 PD 发病进程相关,作者收集了 PD 进展标志物 GWAS 汇总数据进行分析。MR 分析发现 8 个基因在 5 个 PD 进展结果中达到显着性,但这 8 个基因均与 PD 风险和 PD 发病年龄无关;
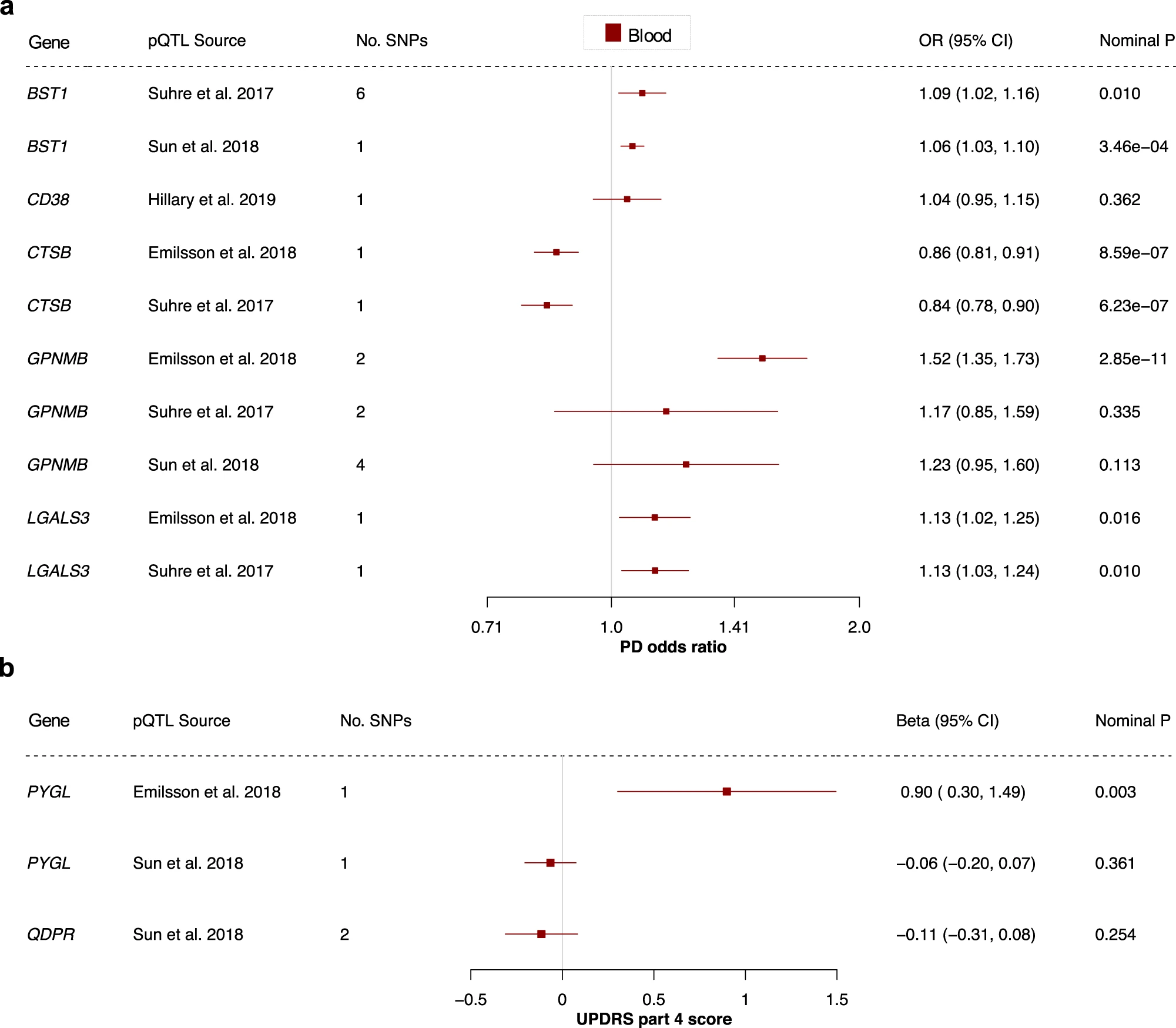
- 整合 PD 相关的药物靶标基因和 pQTL(蛋白质数量性状基因座)数据,发现 BST1、CD38、CTSB、GPNMB 和 LGALS3 的蛋白质水平与 PD 风险相关。除 BST1 外,所有基因的 pQTL 和 eQTL 的效应方向一致,说明整合 MR 、eQTL、pQTL 可有效鉴定药物靶标;
亮点: 系统性地整合药物靶标、GWAS 组学数据和 MR 分析;
局限: 本文的工具变量数量普遍较少,这也是为什么上下文的结果在换了队列数据或者换了组学数据后,大多数基因的作用方向会不一致甚至不显著的情况。当然,这也是 MR 研究普遍存在的问题。
文章链接:
https://www.nature.com/articles/s41467-021-26280-1
公开的资料:
- eQTL 数据:https://eqtlgen.org/; http://resource.psychencode.org/
- 药物靶标基因:https://www.nature.com/articles/s41467-021-26280-1#MOESM1
- PD 发病年龄 GWAS 汇总数据:http://pdgenetics.org/resources;
- PD 进展 GWAS 汇总数据:https://pdgenetics.shinyapps.io/pdprogmetagwasbrowser/
- 代码:https://github.com/catherinestorm/mr_druggable_genome_pd
二、其他文献推荐
下面的文献也挺精彩的,但由于下不到原文,或博主时间有限,没法精细解读,故列出来供各位参阅;
当然,你们有精彩的文献想让我解读的(前提是一周内刚出炉的文献),可给我发pdf(然而可能种种原因,我不一定有时间解读,不要对我抱太高期待);
文献题目: A Species-Level Timeline of Mammal Evolution Integrating Phylogenomic Data
不想看英文题目: 整合系统基因组数据评估哺乳动物进化时间线
杂志和影响因子: Nature (IF: 42.778; Q1)
文章链接:
https://www.nature.com/articles/s41586-021-04341-1
文献题目: Cancer risk across mammals
不想看英文题目: 哺乳动物的癌症风险
杂志和影响因子: Nature (IF: 42.778; Q1)
文章链接:
https://www.nature.com/articles/s41586-021-04224-5
文献题目: Large uncertainty in individual polygenic risk score estimation impacts PRS-based risk stratification
不想看英文题目: 个体多基因风险评分的巨大不确定性影响后续分析(例如基于多基因分险评分的分层分析)
杂志和影响因子: Nat Genet (IF: 38.33; Q1)
文章链接:
https://www.nature.com/articles/s41588-021-00961-5
文献题目: Hidden biases in germline structural variant detection
不想看英文题目: 生殖系结构变异检测中的隐藏偏倚
杂志和影响因子: Genome Biol (IF: 10.81; Q1)
文章链接:
https://genomebiology.biomedcentral.com/articles/10.1186/s13059-021-02558-x
文献题目: Rare coding variants in 35 genes associate with circulating lipid levels—A multi-ancestry analysis of 170,000 exomes
不想看英文题目: 35 个基因中的罕见编码变异与循环脂质水平相关—— 170,000 个外显子组的多祖先分析
杂志和影响因子: Am J Hum Genet (IF: 11.03; Q1)
文章链接:
https://pubmed.ncbi.nlm.nih.gov/34932938/
文献题目: Role of Polygenic Risk Score in the Familial Transmission of Bipolar Disorder in Youth
不想看英文题目: 多基因风险评分在青少年双相情感障碍家族传递中的作用
杂志和影响因子: JAMA Psychiatry (IF: 21.6; Q1)
文章链接:
https://pubmed.ncbi.nlm.nih.gov/34935868/
文献题目: Integration of high-resolution promoter profiling assays reveals novel, cell type-specific transcription start sites across 115 human cell and tissue types
不想看英文题目: 整合高分辨率启动子数据集揭示新型细胞类型特异性转录起始位点
杂志和影响因子: Genome Res (IF: 9.04; Q1)
文章链接:
https://pubmed.ncbi.nlm.nih.gov/34949670/
文献题目: Locus-specific chromatin profiling of evolutionarily young transposable elements
不想看英文题目: 进化中的年轻转座子的位点特异性染色质分析
杂志和影响因子: Nucleic Acids Res (IF: 16.97; Q1)
文章链接:
https://academic.oup.com/nar/advance-article/doi/10.1093/nar/gkab1232/6462377
文献题目: Mutation and selection explain why many eukaryotic centromeric DNA sequences are often A + T rich
不想看英文题目: 突变和选择解释了为什么许多真核着丝粒 DNA 序列通常富含 A+T
杂志和影响因子: Nucleic Acids Res (IF: 16.97; Q1)
文章链接:
https://academic.oup.com/nar/advance-article/doi/10.1093/nar/gkab1219/6470688
三、工具或资源类介绍
文献题目: CCPE: cell cycle pseudotime estimation for single cell RNA-seq data
不想看英文题目: CCPE:单细胞 RNA-seq 数据的细胞周期伪时间估计
杂志和影响因子: Nucleic Acids Res (IF: 16.97; Q1)
文章链接:
https://academic.oup.com/nar/advance-article/doi/10.1093/nar/gkab1236/6471855
文献题目: HiCRes: a computational method to estimate and predict the genomic resolution of Hi-C libraries
不想看英文题目: HiCRes:一种估计和预测 Hi-C 文库基因组分辨率的计算方法
杂志和影响因子: Nucleic Acids Res (IF: 16.97; Q1)
文章链接:
https://academic.oup.com/nar/advance-article/doi/10.1093/nar/gkab1235/6470675
文献题目: CoV-Spectrum: Analysis of Globally Shared SARS-CoV-2 Data to Identify and Characterize New Variants
不想看英文题目: CoV-Spectrum:分析全球共享的 SARS-CoV-2 数据以识别和表征新变异
杂志和影响因子: Bioinformatics (IF: 5.61; Q1)
文章链接:
https://academic.oup.com/bioinformatics/advance-article/doi/10.1093/bioinformatics/btab856/6483076
文献题目: DeepSVP: Integration of genotype and phenotype for structural variant prioritization using deep learning
不想看英文题目: DeepSVP:使用深度学习整合基因型和表型数据以进行结构变异优先排序
杂志和影响因子: Bioinformatics (IF: 5.61; Q1)
文章链接:
https://academic.oup.com/bioinformatics/advance-article/doi/10.1093/bioinformatics/btab859/6482742
文献题目: AMC: accurate mutation clustering from single-cell DNA sequencing data
不想看英文题目: AMC:从单细胞 DNA 测序数据中进行精准的突变聚类
杂志和影响因子: Bioinformatics (IF: 5.61; Q1)
文章链接:
致谢橙子牛奶糖(陈文燕),请用参考模版:We thank the blogger (orange_milk_sugar, Wenyan Chen) for XXX
感谢小可爱们多年来的陪伴, 我与你们一起成长~

本文来自博客园,作者:橙子牛奶糖(陈文燕),转载请注明原文链接:https://www.cnblogs.com/chenwenyan/p/15734431.html





