本周最新文献速递20211107(论怎么在信号少、样本量少的情况下发到NG上)
本周最新文献速递20211107
一、精细解读文献 一
文献题目: The context-specific role of germline pathogenicity in tumorigenesis
不想看英文题目: 生殖系致病性突变在肿瘤发生中的特定作用
杂志和影响因子: Nat Genet (IF: 38.33; Q1)
研究意义: 人类癌症是由环境、遗传和体细胞多种因素共同驱动的结果,但这些因素在肿瘤中如何相互作用却知之甚少。
结论:
- 为了了解生殖系致病性突变在肿瘤发生中的作用,作者收集了 17,152 名癌症患者的测序数据,涵盖 55 种癌症类型和 413 种组织亚型;
- 随后整合了突变类型、功能类别、等位基因频率等信息,对 6,009 名癌症患者的测序数据进行机器学习,用于预测 468 个癌症相关基因的致病性突变/拷贝数变异的功能性影响;
- 该模型总共鉴定了 183 个基因上的 1,499 个致病性突变/拷贝数变异。根据外显率对致病性突变进行三分类:高、中、低。与欧洲 (6.9%)、非洲 (5.9%) 或亚洲 (5.2%) 血统的患者相比,德系犹太人患者的高/中外显率变异比例(11.4%)更高;
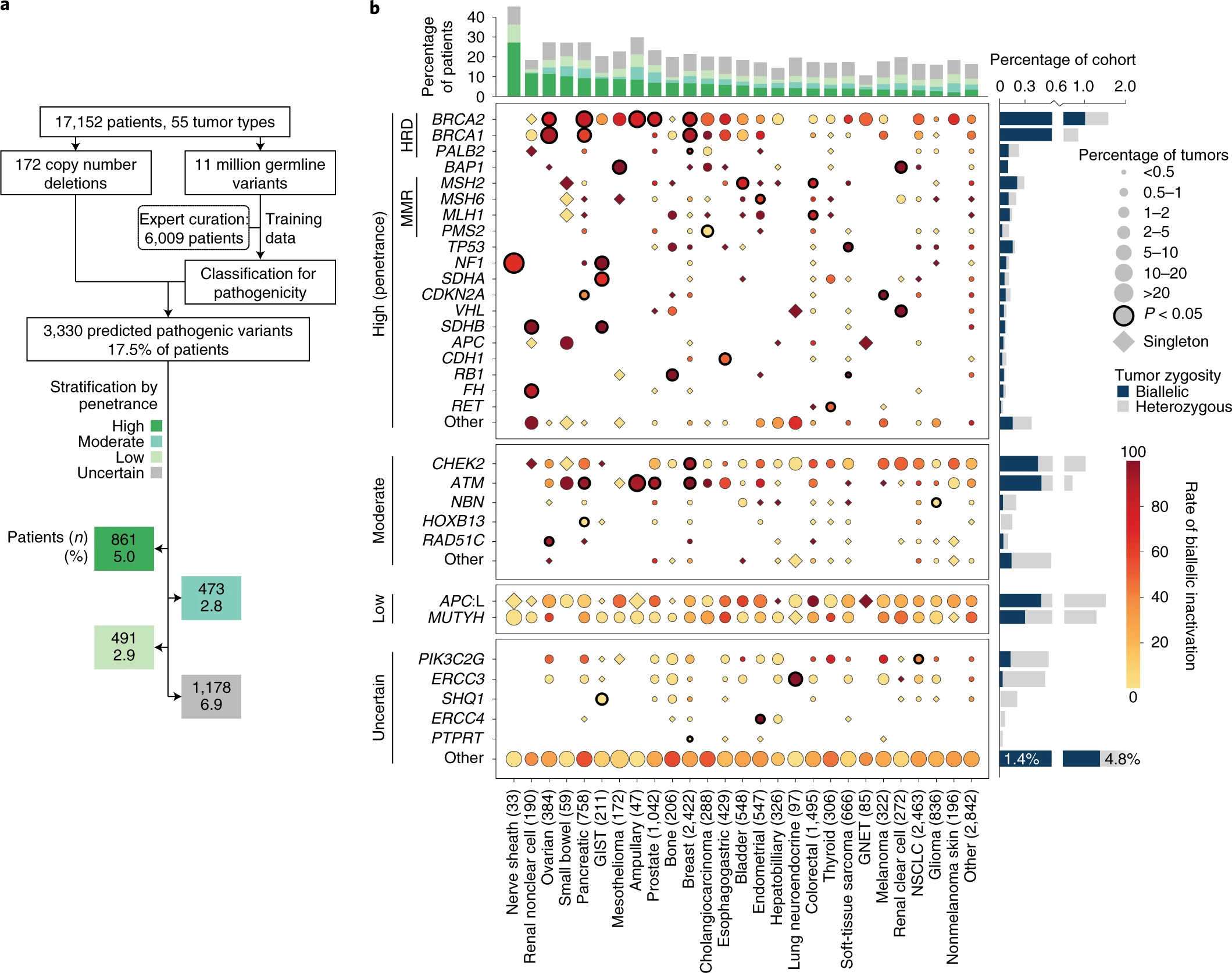
- 野生型等位基因 (WT) 缺失是肿瘤发生的关键因素,作者用体细胞拷贝数杂合性丢失 (LOH)或第二体细胞突变评估肿瘤特异性体细胞双等位基因失活率,作为衡量肿瘤形成和进展的指标。总共挑选了 176 个抑癌基因中的 57 个基因(超过5位患者均有该基因的致病性突变)进行评估,体细胞致病性突变双等位基因失活率为 40%,显着高于良性变异的双等位基因失活率(22.1%;P = 5.7 × 10-76);
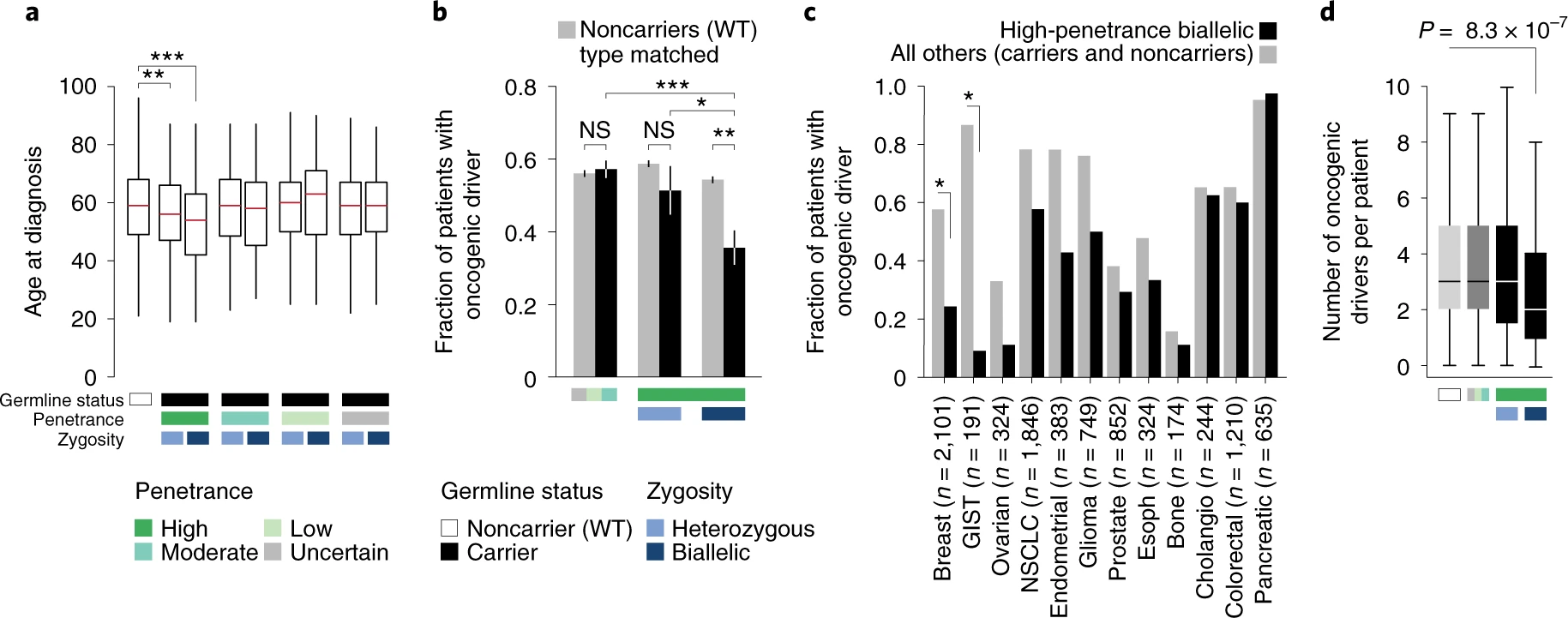
- 鉴于肿瘤患者中双等位基因失活的普遍性和可变性,作者随后考察了影响双等位基因失活的可能原因。首先考虑不同外显率水平的影响,对外显率进行分层分析,发现高外显率基因的肿瘤特异性双等位基因失活率最高(致病变异为 65%,良性变异为 23%,P = 2.8 × 10-125)、低外显率患者的双等位基因失活率较低。除了外显率,作者推测肿瘤谱系也影响双等位基因失活的选择压力,因此,根据肿瘤与致病性突变发生率的相关性进行肿瘤谱系的分层,同样观察到相关性高的肿瘤类型其双等位基因失活率(85%)远高于相关性低的肿瘤类型 (43%,P = 2.6 × 10-21),说明外显率和肿瘤谱系均与双等位基因失活率有关;

- 前面观察到外显率和肿瘤谱系均与双等位基因失活率相关,作者进一步分析了携带双等位基因失活患者的临床表型,结果表明具有双等位基因失活的携带者发病年龄较低(P = 6 × 10 -14),且携带的体细胞驱动突变数量较少(P = 8.3 × 10-7);
体细胞驱动突变: 包括 gain-of-function 和 loss-of-function Mutations
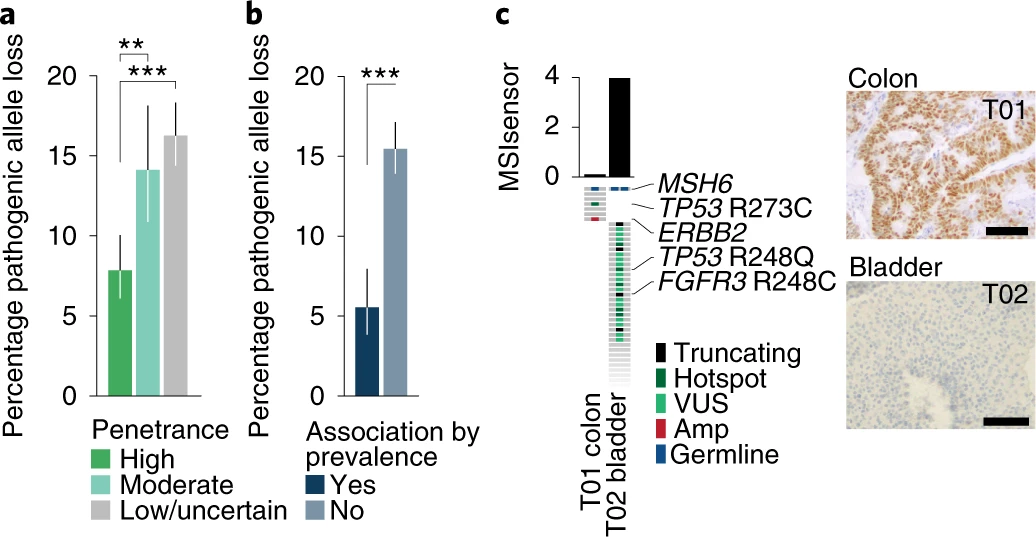
- 为了进一步探索体细胞双等位基因失活对生殖系疾病的驱动作用,作者以林奇综合征 (LS) 作为研究对象。LS 患者的种系错配修复(MMR)基因容易发生微卫星不稳定 (MSI),MSI 是一种与肿瘤无关的生物标志物。结果观察到在发生体细胞双等位基因失活的MMR携带者中,发生MSI的比例大大提升,说明体细胞双等位基因失活可影响 MSI 表型;

- 以上结果表明体细胞双等位基因失活对肿瘤的发生和进展起着重要作用,作者随后分析了生殖系突变对肿瘤的影响,结果发现13.6% 生殖系致病性突变在肿瘤进展中发生丢失,尤其是外显率低的肿瘤。此外,生殖系致病性突变的丢失率在非相关癌症类型中显着更高 (P = 1.5 × 10-9)。这些结果表明,在某些患者中,他们的癌症在肿瘤进展过程中失去了对生殖系致病性突变的依赖,或者他们的肿瘤发生与遗传缺陷无关;
亮点: 将外显率、体细胞双等位基因失活率、生殖系致病性突变、肿瘤类型等进行分层分析,发现体细胞突变和生殖系突变共同驱动肿瘤的发生和进展,并回答了以下几个问题:
- 为什么不同肿瘤的外显率会不一样;
- 为什么有些患者发病年龄很早;
- 在携带生殖系突变的患者中,体细胞突变对肿瘤的作用;
文章链接:
https://www.nature.com/articles/s41588-021-00949-1
公开的资料:
代码:https://github.com/taylor-lab/somatic-germline
二、精细解读文献 二
文献题目: Genome-wide association analyses highlight etiological differences underlying newly defined subtypes of diabetes
不想看英文题目: 全基因组关联分析重新定义糖尿病亚型的遗传差异
杂志和影响因子: Nat Genet (IF: 38.33; Q1)
研究意义: 2 型糖尿病 (T2D) 被认为是一种高度异质性的疾病,患者存在不同临床特征、疾病进展、药物反应和并发症风险。T2D可分为五种亚型:自身免疫性糖尿病(SAID,6% 的患者)、胰岛素缺乏型糖尿病(SIDD,18% 的患者)、胰岛素抵抗性糖尿病(SIRD,15% 的患者)、肥胖相关糖尿病(MOD,22% 的患者)、年龄相关糖尿病(MARD,39% 的患者);然而,这些亚型间的病因差异尚不清楚;
结论:
- 收集了 SAID ( n = 450 )、SIDD ( n = 1,186)、SIRD ( n = 1,125)、MOD ( n = 1,372) 和 MARD ( n = 2,853) 五种亚型进行关联分析,发现 MOD (87.8%) 和 SIDD (83.7%) 亚型的一级或二级亲属有更高的比例具有 T2D 家族史,而 SAID (72.8%)的比例则较低;
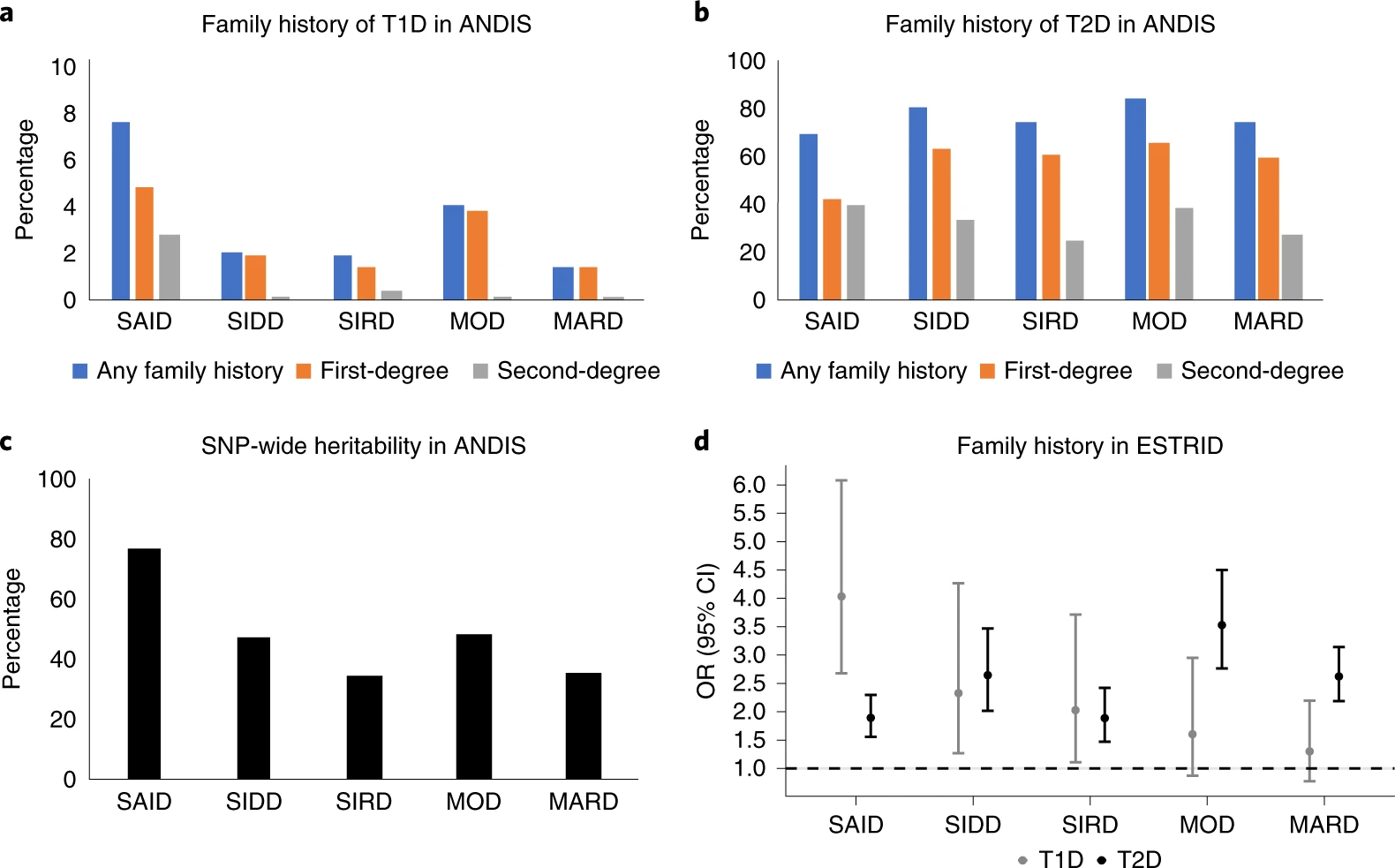
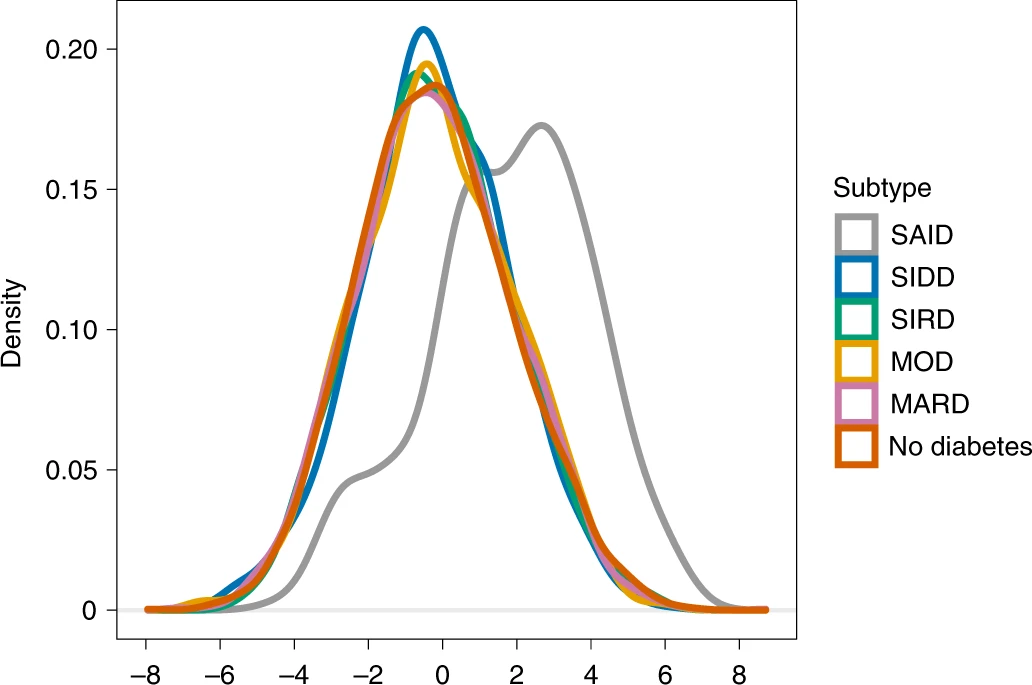
- 对所有的基因型构建 T2D 多基因风险评分 (T2D gPRS),T2D gPRS 显示所有糖尿病亚型的遗传风险增加,其中 SIDD 和 MOD 的遗传风险最高,而 SAID、SIRD 和 MARD 的风险较低;
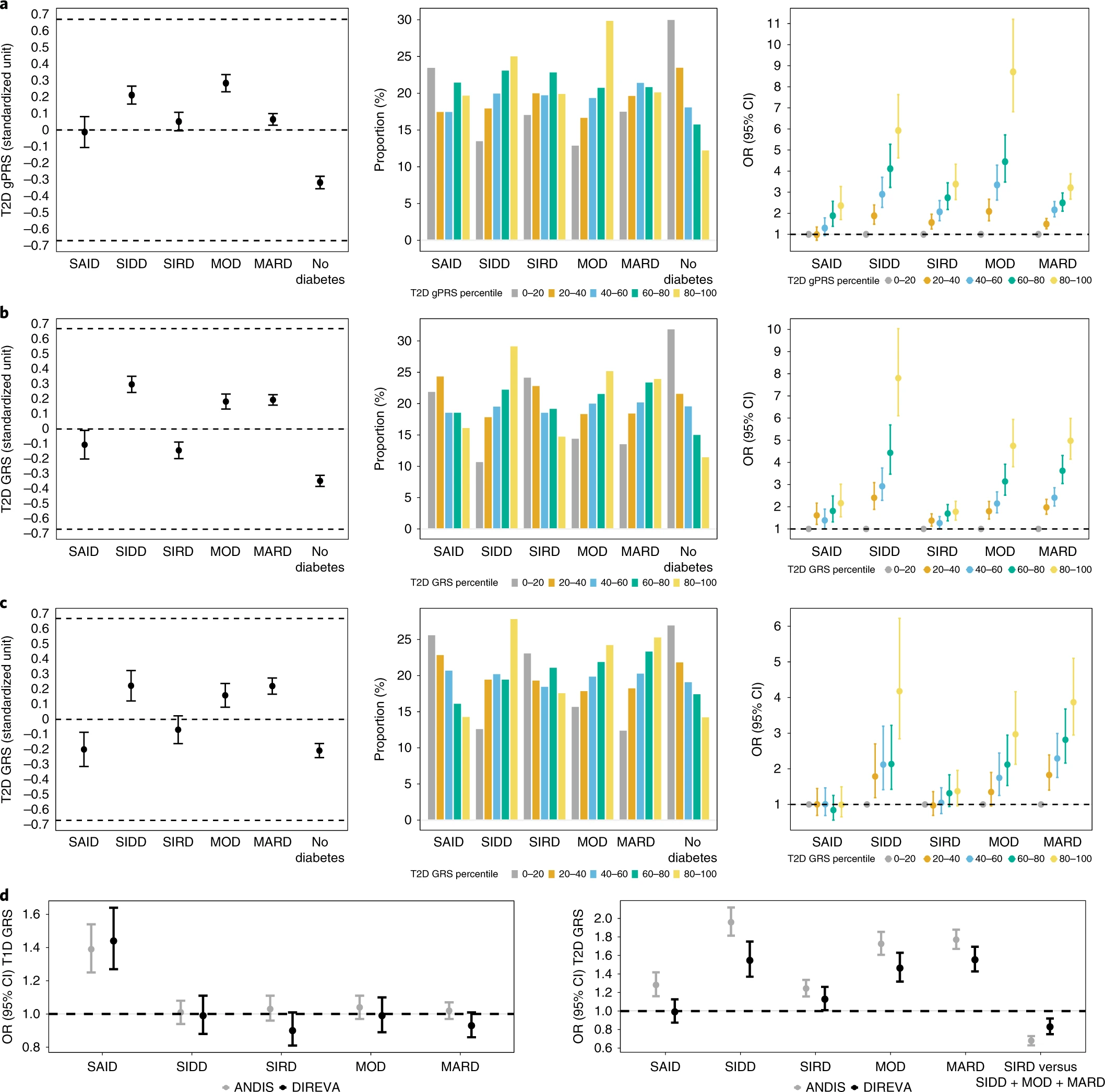
- 对所有的基因型构建 T1D 多基因风险评分 (T1D gPRS), 发现 T1D GRS 仅与自身免疫性 SAID 相关(P ≤ 3.36 × 10-8);
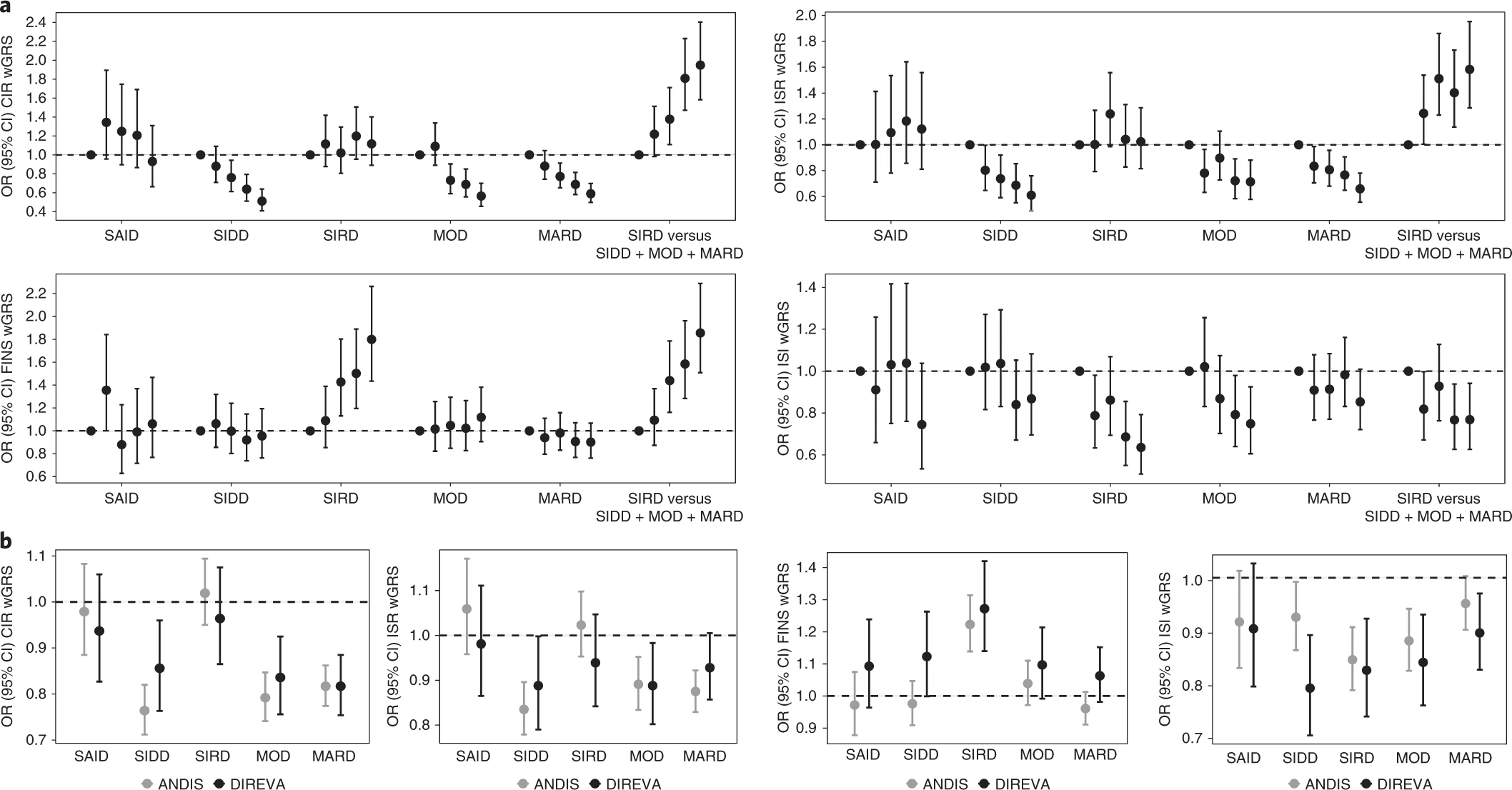
- 分别对胰岛素分泌和胰岛素敏感性进行加权,构建 T2D 相关位点的加权遗传风险评分 (T2D wGRS),结果发现胰岛素反应遗传风险评分 (CIR wGRS ) 与 SIDD (OR80–100 = 0.51, P = 3.9 × 10-9)、MOD (OR80–100 = 0.56, P = 1.6 × 10-7) 、 MARD (OR80–100 = 0.59, P = 7.6 × 10-10) 显著相关,但与 SIRD 没有关联(P > 0.5)。空腹胰岛素遗传风险评分 (FINS wGRS)仅与 SIRD 相关(P = 3.10 × 10-8),与其他亚型无显着相关(P > 0.1)。胰岛素敏感性指数评分 (ISI wGRS) 与 SIRD ( P = 2.93 × 10-6 ) 和 MOD ( P = 1.87 × 10-4) 显著相关;
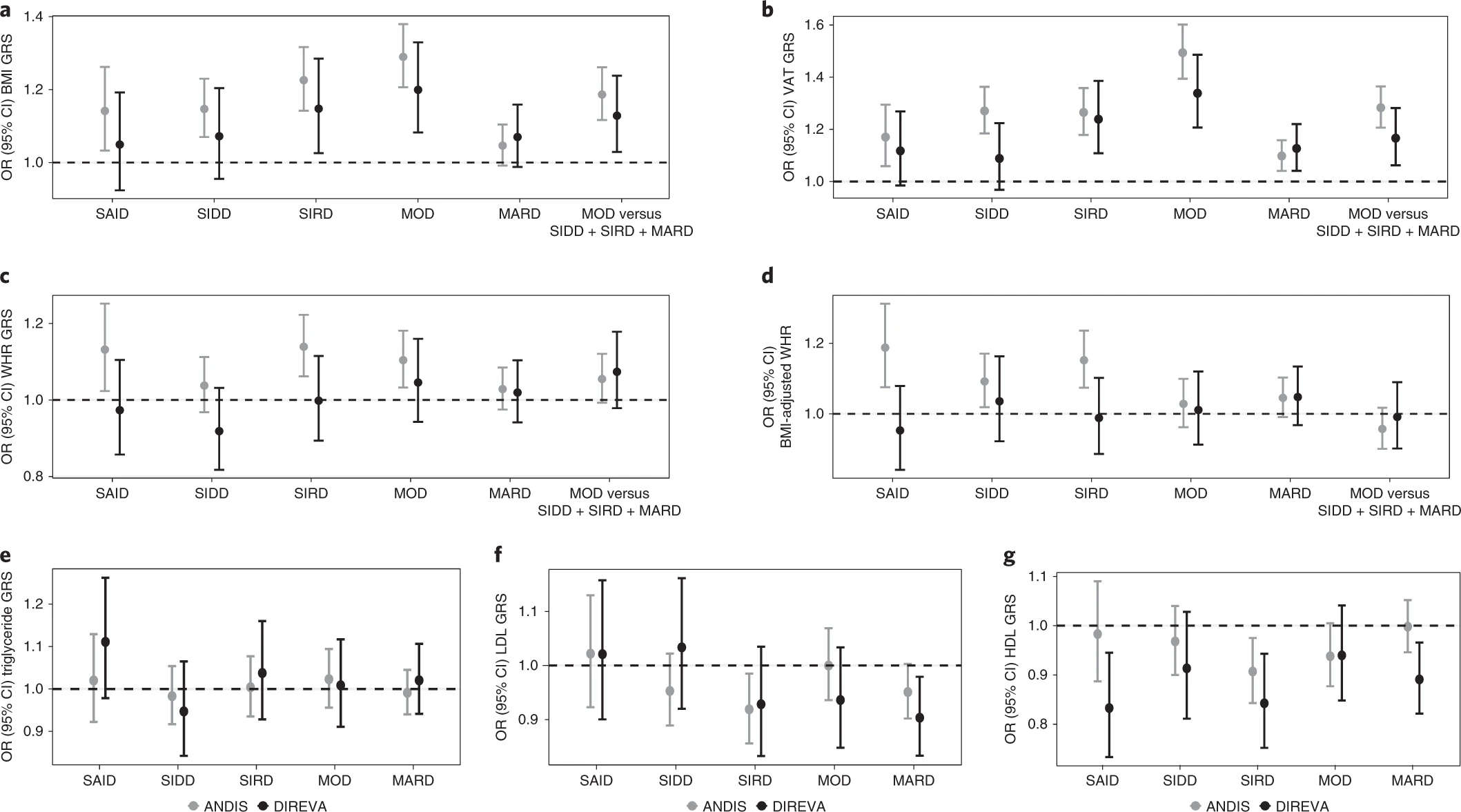
- 对脂质和体重相关表型构建 GRS,结果表明 BMI GRS 与 MOD 的相关性最强(OR = 1.29,95% CI = 1.206–1.379,P = 9.80 × 10-14),而 MARD 没有显示相关性(P = 0.099)。腰臀比 (WHR) 的 GRS 与 SIRD ( P = 2.89 × 10-4 ) 和 MOD ( P = 3.84 × 10-3 ) 相关。BMI 调整的 WHR 的 GRS 与 SIRD 的关联最强(P = 7.59 × 10-5),但与 MOD 没有关联;
亮点: 通过遗传风险评分解析五种 T2D 亚型的遗传背景差异;
- 这篇文章用传统思路分析,只能发二三区(样本少,power低,显著信号少),所以作者不走常规路,而是通过解析疾病异质性背后的遗传差异拔高文章的高度,有同样问题的朋友们可以借鉴这篇文章哦:论怎么在信号少、样本量少的情况下发到NG上;
文章链接:
https://www.nature.com/articles/s41588-021-00948-2
公开的资料:
- GWAS Catalog (www.ebi.ac.uk/gwas/):no. GCST90026412-7
- GRS 结果:
https://www.pgscatalog.org/score/PGS000024/;
http://www.pgscatalog.org/:PGS000832-64;
三、其他文献推荐
下面的文献也挺精彩的,但由于下不到原文,或博主时间有限,没法精细解读,故列出来供各位参阅;
当然,你们有精彩的文献想让我解读的(前提是一周内刚出炉的文献),可给我发pdf(然而可能种种原因,我不一定有时间解读,不要对我抱太高期待);
文献题目: Refining models of archaic admixture in Eurasia with ArchaicSeeker 2.0
不想看英文题目: ArchaicSeeker 2.0 模型重塑欧亚大陆与远古人类的基因交流
杂志和影响因子: Nat Commun (IF: 14.92; Q1)
文章链接:
https://www.nature.com/articles/s41467-021-26503-5
文献题目: Disease variant prediction with deep generative models of evolutionary data
不想看英文题目: 利用进化数据生成的模型进行疾病变异预测
杂志和影响因子: Nature (IF: 42.778; Q1)
文章链接:
https://www.nature.com/articles/s41586-021-04043-8
文献题目: MC3R links nutritional state to childhood growth and the timing of puberty
不想看英文题目: 营养状态通过 MC3R 信号影响儿童成长和青春期
杂志和影响因子: Nature (IF: 42.778; Q1)
文章链接:
https://www.nature.com/articles/s41586-021-04088-9
文献题目: A generalized linear mixed model association tool for biobank-scale data
不想看英文题目: 开发了基于广义线性混合模型 (GLMM) 的全基因组关联分析工具:fastGWA-GLMM,该工具速度更快、更好的控制极端比例的二元表型
杂志和影响因子: Nat Genet (IF: 38.33; Q1)
文章链接:
https://www.nature.com/articles/s41588-021-00954-4
文献题目: Genome-wide analysis of 53,400 people with irritable bowel syndrome highlights shared genetic pathways with mood and anxiety disorders
不想看英文题目: 对 53,400 名肠易激综合征患者的全基因组关联分析发现了与情绪和焦虑症的共同遗传途径
杂志和影响因子: Nat Genet (IF: 38.33; Q1)
文章链接:
https://www.nature.com/articles/s41588-021-00950-8
文献题目: Synonymous mutations reveal genome-wide levels of positive selection in healthy tissues
不想看英文题目: 同义突变揭示了健康组织整个基因组的阳性选择水平
杂志和影响因子: Nat Genet (IF: 38.33; Q1)
文章链接:
https://www.nature.com/articles/s41588-021-00957-1
文献题目: Pairwise effects between lipid GWAS genes modulate lipid plasma levels and cellular uptake
不想看英文题目: 脂质 GWAS 基因的成对累加效应 (AE) 调节脂质血浆水平和细胞摄取
杂志和影响因子: Nat Commun (IF: 14.92; Q1)
文章链接:
https://www.nature.com/articles/s41467-021-26761-3
文献题目: Multi-omics analysis identifies therapeutic vulnerabilities in triple-negative breast cancer subtypes
不想看英文题目: 多组学分析鉴定三阴性乳腺癌亚型的治疗漏洞
杂志和影响因子: Nat Commun (IF: 14.92; Q1)
文章链接:
https://www.nature.com/articles/s41467-021-26502-6
四、工具或资源类介绍
文献题目: GreeNC 2.0: a comprehensive database of plant long non-coding RNAs
不想看英文题目: GreeNC 2.0:植物长非编码 RNA 的综合数据库
杂志和影响因子: Nucleic Acids Res (IF: 16.97; Q1)
文章链接:
https://academic.oup.com/nar/advance-article/doi/10.1093/nar/gkab1014/6415108
文献题目: CoCoPRED: coiled-coil protein structural feature prediction from amino acid sequence using deep neural networks
不想看英文题目: CoCoPRED:使用深度神经网络从氨基酸序列预测卷曲螺旋蛋白结构特征
杂志和影响因子: Bioinformatics (IF: 5.61; Q1)
文章链接:
https://doi.org/10.1093/bioinformatics/btab744
文献题目: Over 1000 tools reveal trends in the single-cell RNA-seq analysis landscape
不想看英文题目: 超过 1000 个工具揭示单细胞 RNA-SEQ 的趋势
杂志和影响因子: Genome Biol (IF: 10.81; Q1)
文章链接:
https://genomebiology.biomedcentral.com/articles/10.1186/s13059-021-02519-4
致谢橙子牛奶糖(陈文燕),请用参考模版:We thank the blogger (orange_milk_sugar, Wenyan Chen) for XXX
感谢小可爱们多年来的陪伴, 我与你们一起成长~

本文来自博客园,作者:橙子牛奶糖(陈文燕),转载请注明原文链接:https://www.cnblogs.com/chenwenyan/p/15521524.html



