本周最新文献速递20210516
本周最新文献速递20210516
一
文献题目: Landscape of allele-specific transcription factor binding in the human genome
不想看英文题目: 人类基因组中等位基因特异性转录因子结合图谱
杂志和影响因子: Nat Commun (12.121 1区)
研究意义: 基因调控区域变异位点可改变基因表达,并引起细胞和生物学表型发生改变。基因调控区域变异位点一般通过与转录因子特异性结合影响基因转录,本文通过ChIP-Seq数据研究杂合染色体基因座中等位基因特异性结合转录因子的功能效应。
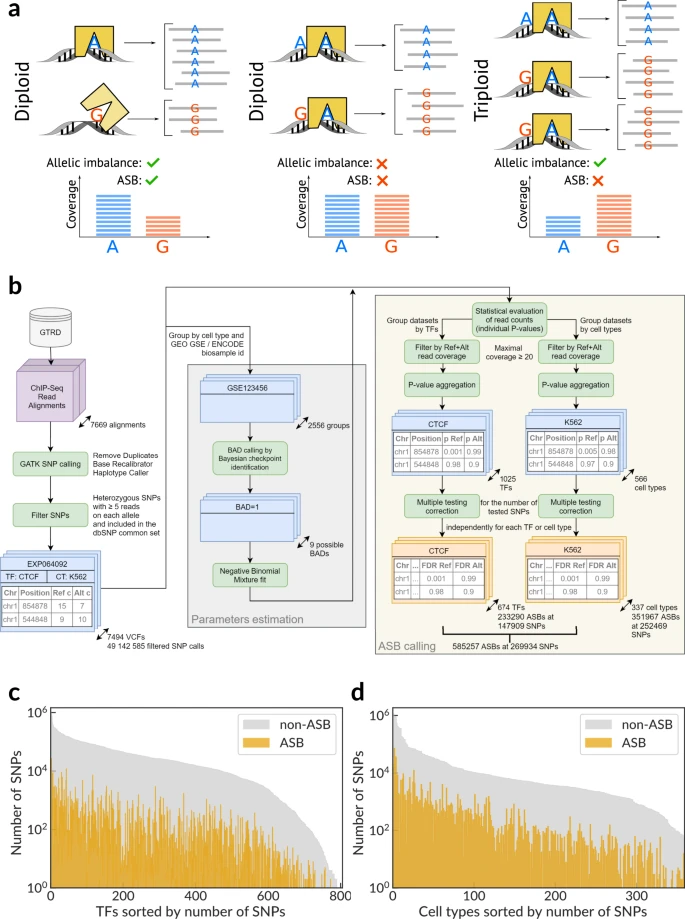
概念:
ASB:等位基因特异性结合事件;
BAD: 背景等位基因剂量,特定基因组区域的两个等位基因拷贝数比例。举个例子,当 BAD = 1,指的是两个等位基因具有相同的拷贝数,比如 1:1(二倍体)、2:2、3:3;
结论:
1)作者提出了一个新的检测 ASB 框架,从 1025 个转录因子( TFs )、566 个细胞类型中检测到 20 万个 ASB;
2)转录因子中,CTCF call 出的 ASB 数量最多,细胞类型中,K562 细胞call 出的 ASB 数量最多;
3)根据统计检验显著性,把 ASB 分为显著 ASB(FDR<0.05)和 候选 ASB ( 原始 P < 0.05)。与 所有 SNV 和候选 ASB 相比,显着 ASB 更多富集在增强子和启动子区域;
4)对序列进行注释,发现 ASB 区域与 TF 基序具有较高的重合和一致性,说明 ASB 检测的准确性较高;
5)使用机器学习预测 ASB(任务 1 :所有 TF 或细胞类型下 ASB 的检测性能;任务 2 :特定 TF 或细胞类型下 ASB 的检测性能),结果发现,对于任务 1 ,TF 和细胞类型的 auROC 分别为 0.74 和 0.73 ,auPRC 分别为 0.44 和 0.56。对于任务 2,CTCF(TFs)和 HepG2(细胞类型)的 auROC 最高,分别为 0.72 和 0.81 ,CTCF(TFs)和 A549(细胞类型)的 auPRC 最高,分别为 0.35 和 0.64;
6)通过比较 ASB 和非 ASB 位点的差异,发现 ASB 更多富集在 eQTL 以及疾病相关的变异位点上,说明 ASB 可作为因果变异位点推断的一个重要信息;
亮点:
全基因组关联研究(GWAS) 找到的变异位点大多位于非编码区,对于变异位点如何影响基因功能,一直没有很好解答。这是第一篇基于 ASB 的方法通过转录因子特异性结合进行非编码变异位点功能注释。
文章链接:
https://pubmed.ncbi.nlm.nih.gov/33980847/
doi: 10.1038/s41467-021-23007-0
公开的资料:
ADASTRA database:http://adastra.autosome.ru/
ChIP-Seq data sets: http://adastra.autosome.ru/soos/downloads
代码:https://github.com/autosome-ru/
二
文献题目: An atlas of alternative polyadenylation quantitative trait loci contributing to complex trait and disease heritability
不想看英文题目: 可变多聚腺苷酸化数量性状基因座图谱可解释复杂性状/疾病遗传力
杂志和影响因子: Nat Genet(27.602 1区)
研究意义: GWAS 已经鉴定出多个人类特征/疾病相关的非编码变异位点。然而这些变异位点的功能解释仍是一项重大挑战。
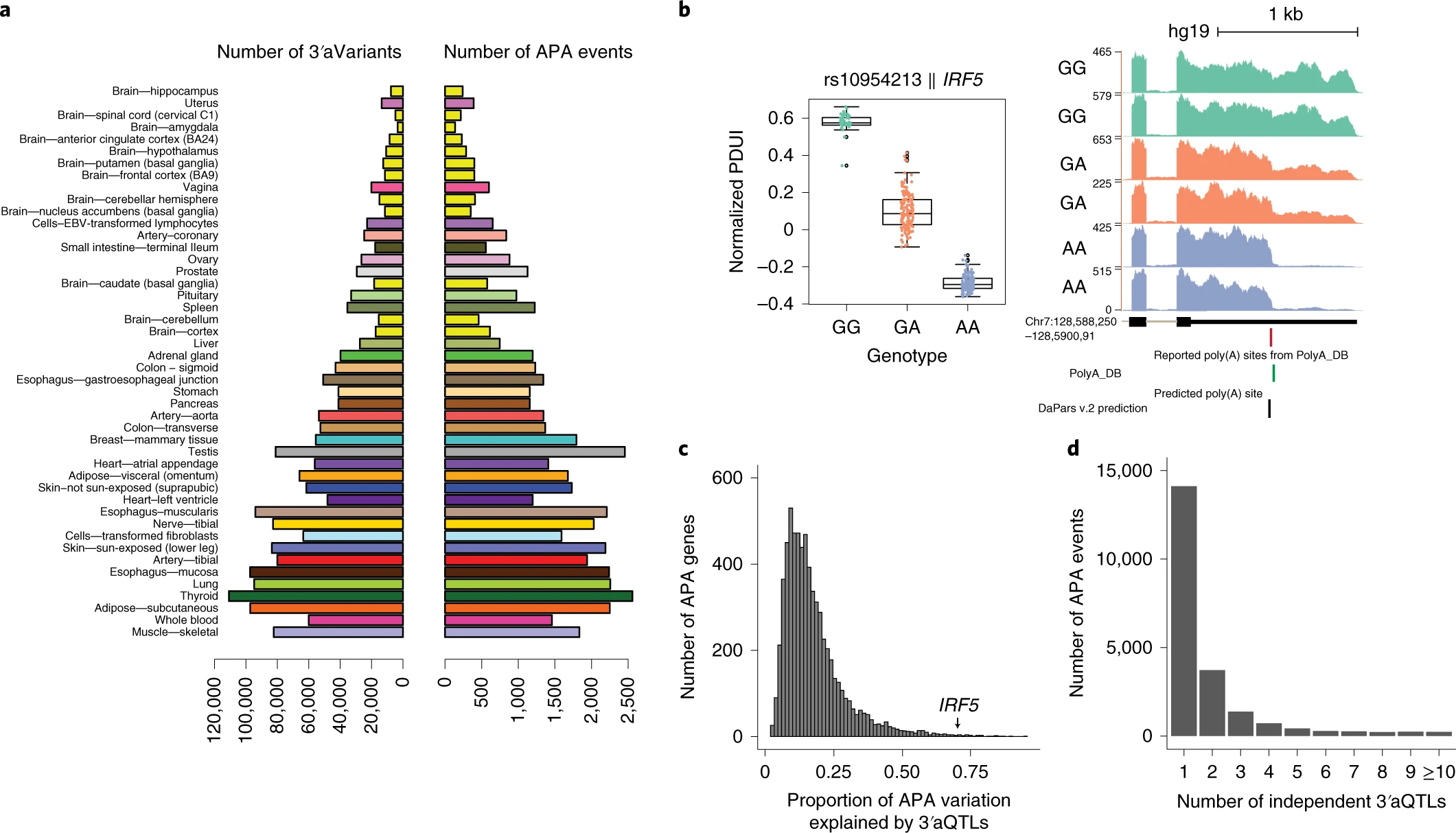
概念:
APA:alternative polyadenylation, 可变多聚腺苷酸化,属于转录后调控,为真核细胞mRNA成熟过程中针对前体 mRNA 3′ 端的一种加工修饰方式。APA通过调控3′非翻译区(3′UTR)长度影响 mRNA 稳定性、翻译效率以及定位;
3’QTLs:3’-UTR 可变多聚腺苷酸化数量性状基因座;
3′aQTLs:3’QTLs相关的变异位点;
3′ aGenes : 3′aQTL相关的基因;
3′aVariants:与3′ aGenes 相关的显著变异位点;
结论:
1)作者开发了检测 APA 的算法(DaPars v.2.0),从 GTEx v.7 的 8,277 个 RNA-seq 样本、46 个组织中鉴定了403,215 个 3′aVariants 、11,613 个 3′aGenes;
2)根据 3′aQTLs 遗传效应方向,可将组织分为脑组织和非脑组织。此外,3′aQTLs 比 eQTLs 更具有组织特异性;
3)从基因组区域上看,3′aQTLs 显著富集在 3′UTRs 或者基因下游 5 kb 以内;3′aQTLs 富集在保守区域而非转录因子结合区域;
4)从转录后调控的角度上看,3'aQTL 可通过调控 poly(A)信号基序、RNA 结合蛋白的结合位点、RNA 二级结构等改变3’-UTR 使用偏向性;
5)通过结合 GWAS 数据,发现 3′aQTLs 可解释人类复杂特征/疾病的部分遗传度,说明 3′aQTLs 可作为独立的分子表型影响复杂特征/疾病;
6)83.7% (1,019/1,218) 的 3′aQTL 共定位基因与 eQTL 共定位基因无重合,说明大部分表型共定位的 3'aQTLs 与基因表达无关;
亮点:
开发了从大规模 RNA-seq 中检测 APA 的新算法;
提出了一个新的可解释人类特征/疾病遗传度的分子表型 3′aQTLs ,有助于解释非编码变异位点对人类复杂特征/疾病的遗传贡献;
文章链接:
https://pubmed.ncbi.nlm.nih.gov/33986536/
doi: 10.1038/s41588-021-00864-5
公开的资料:
GTEx RNA-seq:
https://www.ncbi.nlm.nih.gov/projects/gap/cgi-bin/study.cgi?study_id=phs000424.v7.p2
3′aQTLs等:
https://doi.org/10.7303/syn22236281
PAC-seq data for the LARP4-depletion experiment:
https://www.ncbi.nlm.nih.gov/geo/query/acc.cgi?acc=GSE139548
proteomics data:
https://massive.ucsd.edu/ProteoSAFe/QueryMSV?id=MSV000087000
3′aQTLs 可视化网站:
https://wlcb.oit.uci.edu/3aQTL/index.php
DaPars v.2.0 代码:
https://github.com/3UTR/DaPars2
三 (在线工具介绍)
文献题目: CoffeeProt: an online tool for correlation and functional enrichment of systems genetics data
不想看英文题目: CoffeeProt:遗传数据相关性和功能富集分析的在线工具
杂志和影响因子: Nucleic Acids Res( 11.503 1区 )
一句话介绍: CoffeeProt 通过转录组学、蛋白质组学数据,以及结合 eQTL、 pQTL 、GWAS 数据等,对输入的数据进行蛋白质-蛋白质相互作用、亚细胞定位、药物关联分析等。

网站:
输入数据:
- Proteomics/expression data
- pQTL/eQTL data
- GWAS/molQTL data
分析:
- Correlation analysis
- Database enrichment
- SNP-protein analysis
- Network
- Bait network
输出数据:
- Tables
- Static plots
- Interactive plots
文章链接:
https://pubmed.ncbi.nlm.nih.gov/33978718/
doi: 10.1093/nar/gkab352
公开的代码:
https://github.com/JeffreyMolendijk/CoffeeProt
本人陈文燕(橙子牛奶糖)必须在此说明,如果有任何重大的错误,无论是文字上或意义上的,完全都是我的责任。然而虽然我非常欢迎批评,但是我想那只会浪费你宝贵的时间。── 摘自《正见》

本文来自博客园,作者:橙子牛奶糖(陈文燕),转载请注明原文链接:https://www.cnblogs.com/chenwenyan/p/14773870.html



 浙公网安备 33010602011771号
浙公网安备 33010602011771号