一个R包(IntAssoPlot),LocusZoom图、连锁不平衡图和基因结构图一步到位
最近,看到Front. Genet.发了一篇文章“IntAssoPlot: An R Package for Integrated Visualization of Genome-Wide Association Study Results With Gene Structure and Linkage Disequilibrium Matrix”,主要推出了一个R包IntAssoPlot,该包整合了LocusZoom图、连锁不平衡图(LD)和基因结构信息。
往常画LocusZoom图和连锁不平衡图(LD)时需要提取相同的起始、终止位置,再分别用LocusZoom网站和haploview软件画。步骤多,画起来麻烦,关键画完以后还需要将他们拼在一起(看起来就不是很专业)。
IntAssoPlot包的优点是直接一步出图。
下载、安装很简单:
install.packages(c("ggplot2","ggrepel","reshape2"))
if (!requireNamespace("BiocManager", quietly = TRUE))
install.packages("BiocManager")
BiocManager::install(c("SNPRelate","gdsfmt"))
library(remotes) # version 2.1.0
#install_github("whweve/IntAssoPlot")
install_github("whweve/IntAssoPlot",build=TRUE,build_vignettes = TRUE)
下面是IntAssoPlot包的示例数据展示:
library("IntAssoPlot")
example("IntGenicPlot")
example("IntRegionalPlot")
画出来的基因结构、LD图、关联图如下所示:
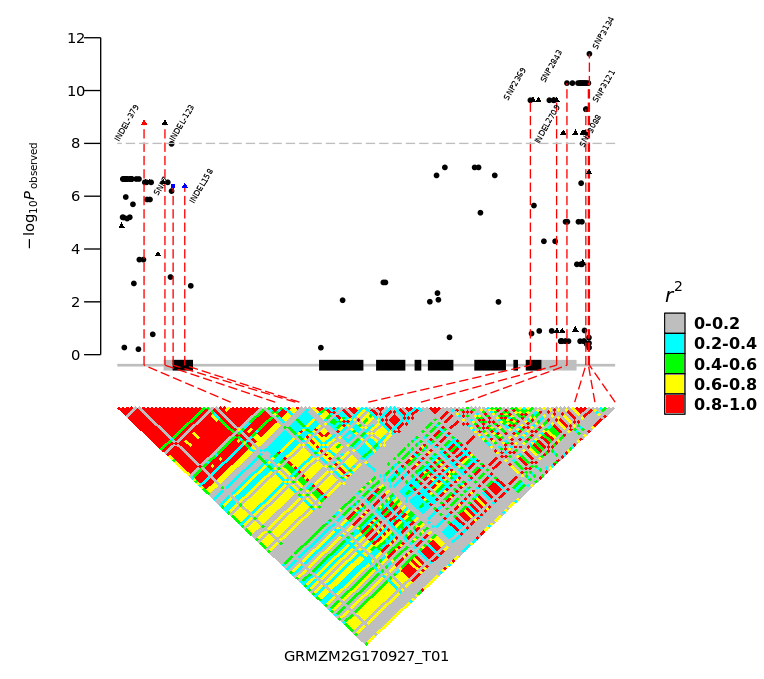
LD图和LocusZoom图:
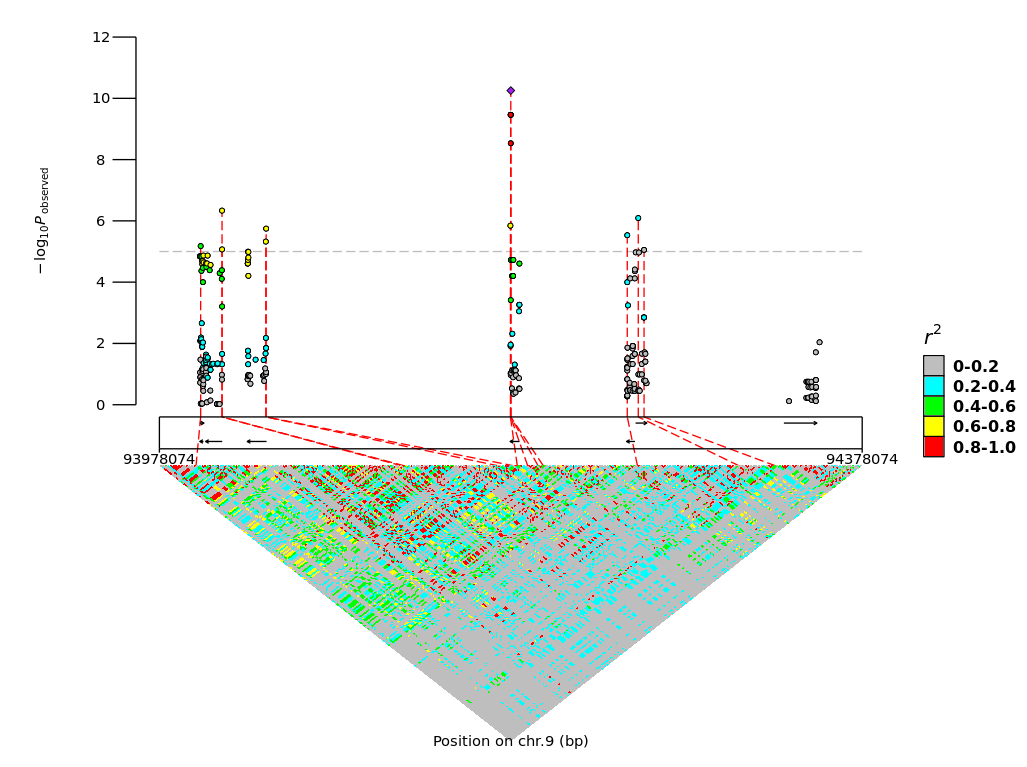
下面是这个包的代码参数使用,作者很贴心的写好了:

以下是上面代码画出来的效果图:

写完这篇推文的时候,挺感慨的。有需求、有想法就可以成就一个包、一篇文献。

本文来自博客园,作者:橙子牛奶糖(陈文燕),转载请注明原文链接:https://www.cnblogs.com/chenwenyan/p/13492692.html



