GWAS后续分析:LocusZoom图的绘制
LocusZoom图几乎是GWAS文章的必备图形之一,其主要作用是可以快速可视化GWAS找出来的信号在基因组的具体信息:比如周围有没有高度连锁的位点,高度连锁的位点是否也显著。
下面是locuszoom的示例图:
下面具体讲讲如何实现Locuszoom的绘制
1、进入Locuszoom的主页
http://locuszoom.org/
2、进入Locuszoom的主页后,点击single plot
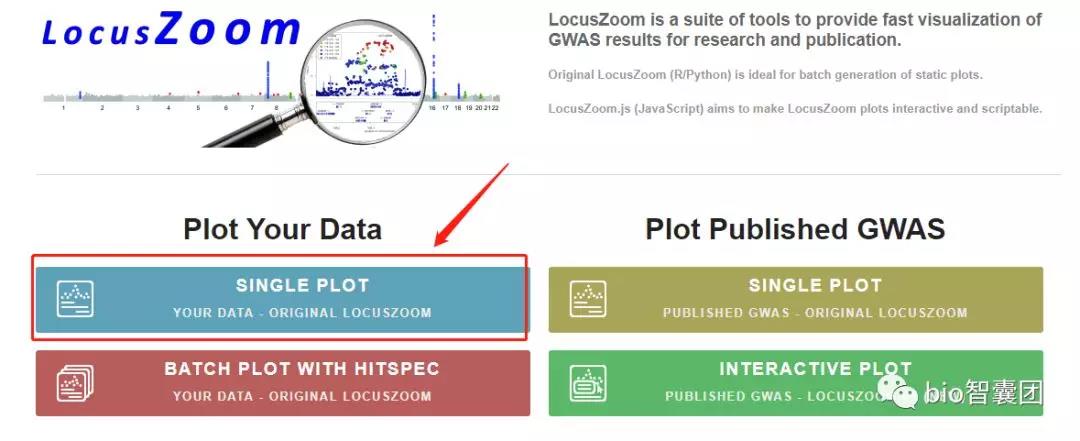
3、按如下图操作
第一步:上传关联分析结果的文件,plink格式的话是assoc.logistic或者assoc.linear。注意:这个文件必须是只含SNP位点的GWAS结果,要把协变量的结果去除掉。
第二步:选择P值所在列的名称,以Plink格式为例,P值所在列的名称为P,也可以点击右方的PLINK data
第三步:同第二步的设置,填写SNP所在的列的名称。

4、指定展示的区域,三选一,可以是SNP,也可以是基因,也可以是区域。

5、选择你所研究的群体的基因组版本(hg18/hg19)
EUR表示欧洲,ASN表示亚洲,AMR表示美洲,AFR表示非洲。
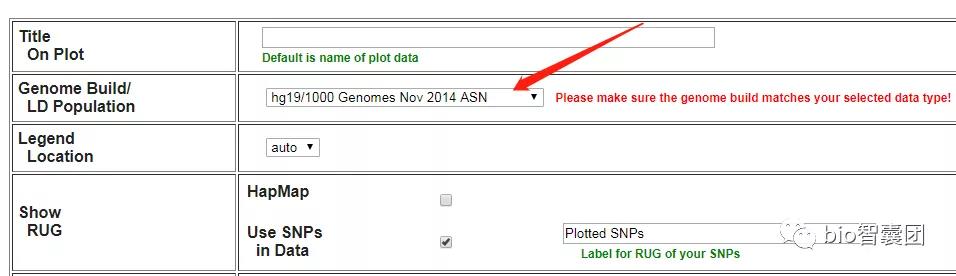
6、最后,点击Plot data,然后就等着收图了。

本文来自博客园,作者:橙子牛奶糖(陈文燕),转载请注明原文链接:https://www.cnblogs.com/chenwenyan/p/10575408.html


