知识图谱-生物信息学-医学论文(BMC Bioinformatics-2022)-挖掘阿尔茨海默病相关KG来确定潜在的相关语义三元组用于药物再利用
论文标题: Mining On Alzheimer’s Diseases Related Knowledge Graph to Identity Potential AD-related Semantic Triples for Drug Repurposing
论文地址: https://arxiv.org/pdf/2202.08712
论文期刊: BMC Bioinformatics 2022
9.(2022.2.17)BMC Bioinformatics-挖掘阿尔茨海默病相关KG来确定潜在的相关语义三元组用于药物再利用
摘要
背景:
迄今为止,大多数神经退行性疾病都没有有效的治疗方法。知识图可以为异构数据提供全面的语义表示,并已成功地应用于包括药物再利用在内的许多生物医学应用中。我们的目标是从文献中构建一个知识图谱,研究阿尔茨海默病(AD)与化学物质、药物和膳食补充剂之间的关系,以确定预防或延缓神经退行性发展的机会。我们收集生物医学注释,并通过SemMedDB使用SemRep提取它们之间的关系。在数据预处理过程中,我们使用了基于BERT的分类器和基于规则的方法来排除噪声,同时保留大多数与AD相关的语义三元组。筛选的1,672,110个三元组使用知识图补全算法(即TransE、DistMult和ComplEx)进行训练,以预测可能有助于AD治疗或预防的候选基因。
结果:
在三种KGC模型中,TransE的补全效果优于其他两种模型(MR=13.45, Hits@1=0.306)。我们利用时间切片技术进一步评估预测结果。我们发现了支持我们模型预测的大多数排名靠前的候选词的证据,这表明我们的方法可以提供可靠的新知识。
结论:
本文表明,我们的图挖掘模型可以预测AD与其他实体(如膳食补充剂、化学品和药物)之间可靠的新关系。构建的知识图谱有助于数据驱动下的知识发现和新假设的生成。
1.背景
神经退行性疾病是一组异质性疾病,其特征是中枢神经系统或周围神经系统的结构和功能逐渐退化。常见的神经退行性疾病,如阿尔茨海默病(AD)和相关痴呆症(ADRD),通常是无法治愈和不可逆转的,很难阻止。
AD/ADRD是一种多因素复杂的神经退行性疾病,以逐渐记忆丧失和严重痴呆伴神经精神症状为特征。到2020年,估计有580万65岁及以上的美国人(12.6%)患有AD/ADRD,预计到2050年这一数字将达到1380万。AD/ADRD的高患病率造成了巨大的医疗和社会负担。2020年,所有患有AD/ADRD的美国人的医疗保健、长期护理和医院服务的总成本估计为3050亿美元。AD/ADRD药物开发的高失败率加剧了人口和财政挑战。鉴于这种疾病日益流行,迫切需要找到创新的方法来开发有效的药物。其中药物再利用是一种用于确定已批准或正在研究的药物的新用途的策略,这些药物不在其原始医学适应症的范围内。与开发全新药物相比,这一策略有几种优势,包括降低失败风险和未知副作用/并发症的风险、有效利用开发资金和缩短开发时间。高通量筛选技术的发展使得计算药物再利用成为最有吸引力的药物发现方法之一,因为海量的可用数据可能会导致药物再利用的新线索,而这些线索是个别项目不可能发现的。然而,知识/数据的复杂性对于理解AD/ADRD的后端机制非常重要,这使得支持疗法发现具有挑战性。
知识图可以为异质数据提供全面的语义表示,这已成功地应用于包括药物再利用在内的许多生物医学应用。例如,最近的一些研究集中在使用基于KG的方法来为新冠肺炎进行药物重定位。有人将KGE方法应用于罕见疾病的药物再利用。有人利用KG的语义特性对常染色体显性多囊肾病(ADPKD)的候选药物进行了优先排序。然而,就我们所知,基于KG的方法很少被应用于AD/ADRD药物的再利用。
本文的目的是使用基于KG的方法研究AD与膳食补充剂、化学物质和药物之间的潜在关系。研究表明,一些药物、化学物质或膳食补充剂可能与预防或延缓神经退化和认知衰退有关。然而,需要进一步的研究来更好地了解后端机制,并揭示与临床和药物(代谢)动力学因素的潜在相互作用。在本文中,我们通过文献挖掘将生物医学概念及其丰富的关系编码成KG。文献挖掘是一种数据挖掘技术,它从文献中识别诸如基因、疾病和化学物质等实体,发现整体趋势,并促进基于现有知识的假设生成。文献挖掘使研究人员能够快速研究大量文献,揭示人工分析难以发现的实体之间的隐藏关系。更具体地说,我们引入了一个生物医学KG,专门关注AD/ADRD,并发现化学品、药物、膳食补充剂和AD/ADRD之间的潜在关系。有关如何构建KG以及如何利用图嵌入方法来预测带有评分的候选者的更多细节将在方法部分进行描述。我们还提供了几种候选者排名以及不同图嵌入算法的比较。
2.方法
为了构建知识图,我们直接从SemMedDB获得三元组,这是一个通过SemRep使用NLP工具自动从生物医学文献(摘要和主题)中提取三元组的数据库。主体和客体的参数被标准化为在具有唯一标识符(CUI)的统一医学语言系统(UMLS)中定义的概念。三元组以“主体-谓语-客体”的形式出现。我们在所有三元组中排除了不相关的概念和关系。有关如何设置这些规则的更多细节将在预处理部分中详细说明。由于三元组是使用SemRep提取的,据报道,SemRep的精确率为0.69,因此我们使用了基于Bert的模型PubMedBERT来进一步提高精度。各步骤的概述如图1所示。
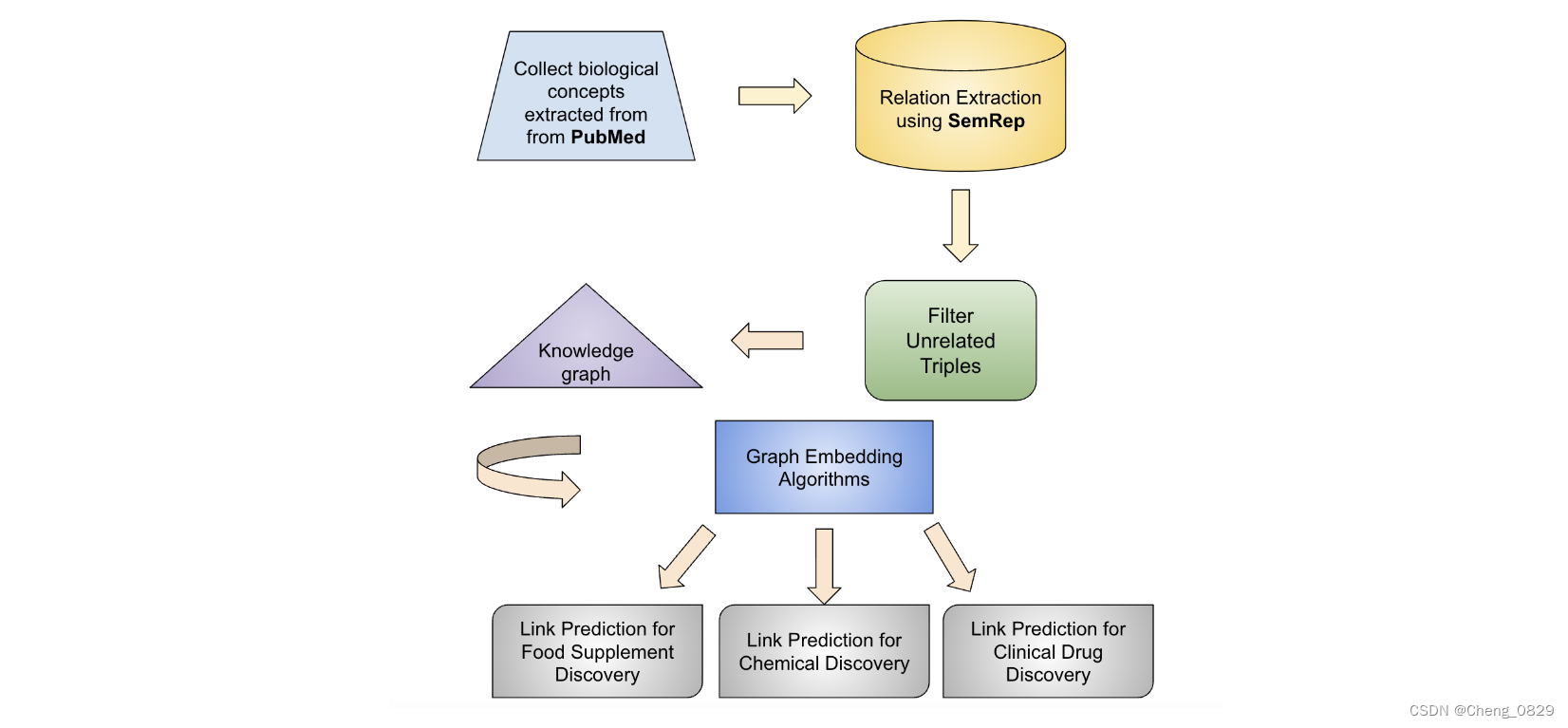
图1-通用步骤:PubMed文献中的生物学概念是使用NLP工具提取的,并使用“主体-关系-客体”三元组构建成知识图。利用图嵌入算法发现潜在候选者并完成知识图。
2.1 预处理
直接从SemMedDB获得的原始数据包含大量的三元组,但并不是所有的数据都对寻找AD或ADRD的治疗/预防的候选方案有用。我们应用一定的规则来排除不相关的主体/客体和谓词类型。更具体地说,我们消除了涉及通用生物医学概念的三元组,如活动和行为、概念和想法、对象、职业、组织和现象。其余的三元组根据其程度中心性\((A_{in},A_{out})\)和指示主体和客体之间联系强度的\(G^2\)分数被剔除。具体地说,用邻接矩阵\(\mathrm{M}\)计算程度中心性\((A_{in},A_{out})\)如下:
\(G^2\)分数是根据两个列联表之间的统计关系计算得出的:观察表和期望表。
列联表(contingency table)是观测数据按两个或更多属性(定性变量)分类时所列出的频数表。它是由两个以上的变量进行交叉分类的频数分布表。
其中,\(O_{ijk}\)表示观察表中的项,并且\(E_{ijk}=\frac{\sum_iO_{ijk}\sum_jO_{ijk}\sum_iO_{ijk}}{(\sum O_{ijk})^2}\)表示期望表中的项。为了确保与AD相关的三元组包括在知识图谱中,我们使用上述标准在三元组消除过程中将与AD相关的所有三元组保留在UMLS中。最后,将三个模型得到的分数归一化到[0,1]之间,并求和得到最终分数。为了将KG保持在图嵌入算法可以处理的合理大小,我们只保留了大约280万个三元组。
2.2 使用PubMedBERT校准
我们利用了来自先前研究的大约6,000个注释,并将它们用作PubMedBERT微调的训练数据。这些注释被手动标记为1或0,其中1表示三元组及其关系确实存在并且是正确的(标记为1的三元组);0表示三元组不存在或不正确(标记为0的三元组)。PubMedBERT接受主体、客体、谓词类型的文本输入以及从中提取这些内容的句子。该模型在验证集上的F1得分为0.82,Recall为0.91,Precision为0.75;在这些用作训练数据的注释上,F1得分为0.83,Recall为0.89,Precision为0.78。
2.3 图嵌入算法
KGE是一种很热门的KGC方法。它将实体和关系嵌入到向量空间中,通过一个评分函数来评估给定的三元组\((h,r,t)\)为真的概率。我们利用了三种流行的KGE方法TransE、DistMult和ComplEx来完成我们的KGC任务。为了训练这个知识图,这三个模型通过将三元组\((h,r,t)\)破坏为\((h',r,t)\)或\((h,r,t')\)形式来进行负采样,其中\(h'\)和\(t'\)是负样本。因此,设\(y=±1\)是正负三元组的标签,\(f\)是得分函数,则逻辑斯蒂损失计算如下:


