PDB文件解读
1. 在PDB文件中,ATOM模块是记录蛋白质分子中原子坐标信息的模块,每一行代表一个原子的坐标信息。
如下图所示是pdb文件的部分内容:
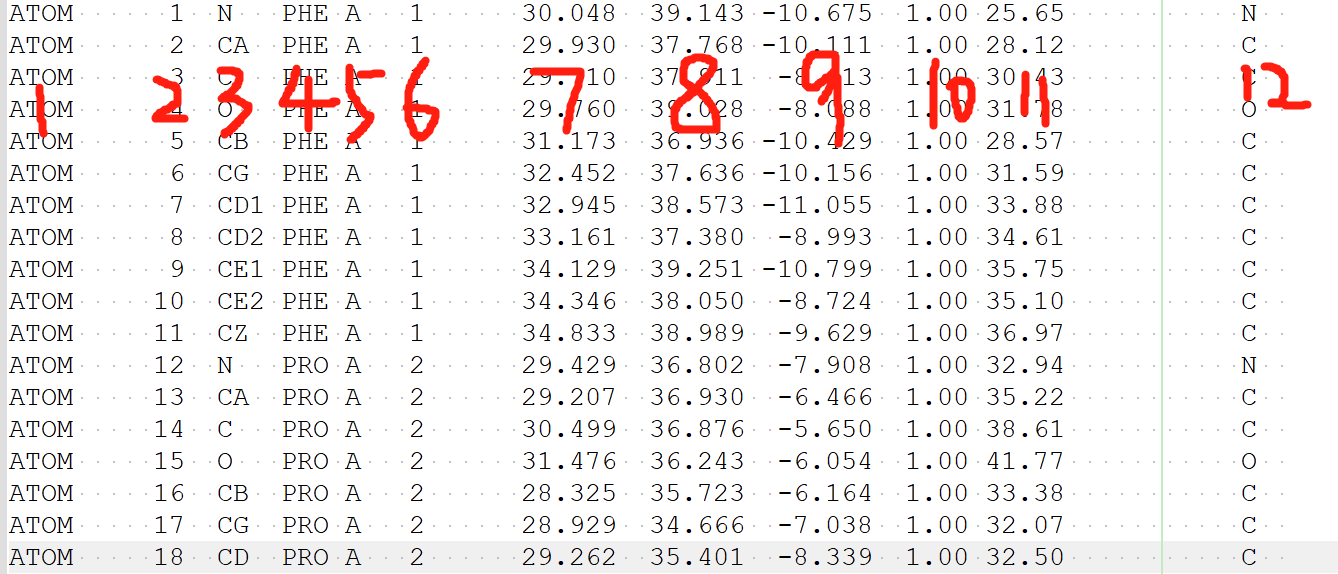
第一列:记录原子的标识号;第二列:原子序号;第三列:原子名称;第四列:残基名称;第五列:链ID;第六列:残基序号;第7-9列:原子坐标;第10列:occupancy,原子占位率;第11列:tempFactor,又称“b-factor”;第12列:原子的元素
2. 以色氨酸(W)残基为例解读每个原子的位置

以氨基酸骨架上的N原子(蓝色)为起始,与之相连的C原子定义为CA,与O原子相连的C定义为C,其他的C原子则根据与CA的距离分别定义为CB,CG,CD,CE,CZ,CH。
分类:
蛋白结构



【推荐】国内首个AI IDE,深度理解中文开发场景,立即下载体验Trae
【推荐】编程新体验,更懂你的AI,立即体验豆包MarsCode编程助手
【推荐】抖音旗下AI助手豆包,你的智能百科全书,全免费不限次数
【推荐】轻量又高性能的 SSH 工具 IShell:AI 加持,快人一步
· 震惊!C++程序真的从main开始吗?99%的程序员都答错了
· winform 绘制太阳,地球,月球 运作规律
· 【硬核科普】Trae如何「偷看」你的代码?零基础破解AI编程运行原理
· 上周热点回顾(3.3-3.9)
· 超详细:普通电脑也行Windows部署deepseek R1训练数据并当服务器共享给他人