【数据处理】PCA(主成分分析)python+matlab代码
一、PCA(Principal Component Analysis)介绍
PCA是数据处理中的一个常用方法,用于数据降维,特征提取等,实际上是将在原有的特征空间中分布的数据映射到新的特征空间(或者说,将原有到正交坐标系进行旋转,使得在旋转后的坐标系下,在某几根坐标轴上数据分布的方差比较大。在这些特征上,不同数据区分度比较高,最高的我们称为第一主成份,按照大小顺序依次第二主成份,第三主成份)。对于高维数据,我们只需要保留k个主要的投影结果,即模最大也就是对应奇异值最大的投影方向,因此投影矩阵也只需要保留前k个主成分对应的特征向量,故 V 也只需要n×k维即可。对数据降维。经过PCA分解以后的分量都是正交的,因此是不相关的,但不一定独立。经过重新映射后的数据不再具有最初的物理意义(都打乱了)。
思考:我们如何得到这些包含最大差异性的主成分方向呢?
答案:计算数据矩阵去均值后的协方差矩阵,得到协方差矩阵的特征值、特征向量,选择特征值最大(即方差最大)的k个特征所对应的特征向量组成的矩阵(映射矩阵),将映射矩阵转置后乘以去均值的数据矩阵得到降维的数据。
二、数学推导
得到协方差矩阵的特征值和特征向量有两种方法:1特征值分解协方差矩阵 。2奇异值分解协方差矩阵。
所以,PCA算法有两种实现方法:基于特征值分解协方差矩阵实现PCA算法、基于SVD分解协方差矩阵实现PCA算法。(矩阵论有关知识)
步骤:
输入:数据集 ![]() ,需要降到k维。
,需要降到k维。
1) 去平均值(即去中心化),即每一位特征减去各自的平均值。
2) 计算协方差矩阵![]() (注:这里除或不除样本数量n或n-1,其实对求出的特征向量没有影响)。
(注:这里除或不除样本数量n或n-1,其实对求出的特征向量没有影响)。
3) 用特征值分解/SVD分解方法求协方差矩阵的特征值与特征向量。
4) 对特征值从大到小排序,选择其中最大的k个。对应的k个特征向量分别作为行向量组成特征向量矩阵P。
5) 将数据转换到k个特征向量构建的新空间中,即去中心化的数据*协方差矩阵的特征向量。
注释:
1 协方差矩阵
我们可以将矩阵看作作用于所有数据并朝向某个方向的力。同时我们还知道了变量间的相关性可以由方差和协方差表达,并且我们希望保留最大方差以实现最优的降维。因此我们希望能将方差和协方差统一表示,并且两者均可以表示为内积的形式,而内积又与矩阵乘法密切相关。因此我们可以采用矩阵乘法的形式表示。若输入矩阵 X 有两个特征 a 和 b,且共有 m 个样本,那么有:
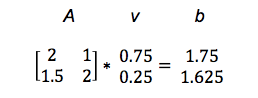
如果我们用 X 左乘 X 的转置,那么就可以得出协方差矩阵
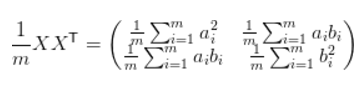
这个矩阵对角线上的两个元素分别是两特征的方差,而其它元素是 a 和 b 的协方差。两者被统一到了一个矩阵的,因此我们可以利用协方差矩阵描述数据点之间的方差和协方差,即经验性地描述我们观察到的数据。
2 PCA降维之前为什么要先标准化?
PCA(主成分分析)所对应的数学理论SVD(矩阵的奇异值分解本身是完全不需要对矩阵中的元素做标准化或者去中心化的。
但是对于机器学习,我们通常会对矩阵(也就是数据)的每一列先进行标准化。
PCA通常是用于高维数据的降维,它可以将原来高维的数据投影到某个低维的空间上并使得其方差尽量大。如果数据其中某一特征(矩阵的某一列)的数值特别大,那么它在整个误差计算的比重上就很大,那么可以想象在投影到低维空间之后,为了使低秩分解逼近原数据,整个投影会去努力逼近最大的那一个特征,而忽略数值比较小的特征。因为在建模前我们并不知道每个特征的重要性,这很可能导致了大量的信息缺失。为了“公平”起见,防止过分捕捉某些数值大的特征,我们会对每个特征先进行标准化处理,使得它们的大小都在相同的范围内,然后再进行PCA。
此外,从计算的角度讲,PCA前对数据标准化还有另外一个好处。因为PCA通常是数值近似分解,而非求特征值、奇异值得到解析解,所以当我们使用梯度下降等算法进行PCA的时候,我们最好先要对数据进行标准化,这是有利于梯度下降法的收敛。
三、PCA实现
1.python
(1)PCA的Python实现:
##Python实现PCA import numpy as np def pca(X,k):#k is the components you want #mean of each feature n_samples, n_features = X.shape mean=np.array([np.mean(X[:,i]) for i in range(n_features)]) #normalization norm_X=X-mean #scatter matrix scatter_matrix=np.dot(np.transpose(norm_X),norm_X) #Calculate the eigenvectors and eigenvalues eig_val, eig_vec = np.linalg.eig(scatter_matrix) eig_pairs = [(np.abs(eig_val[i]), eig_vec[:,i]) for i in range(n_features)] # sort eig_vec based on eig_val from highest to lowest eig_pairs.sort(reverse=True) # select the top k eig_vec feature=np.array([ele[1] for ele in eig_pairs[:k]]) #get new data data=np.dot(norm_X,np.transpose(feature)) return data #输入数据 X = np.array([[-1, 1], [-2, -1], [-3, -2], [1, 1], [2, 1], [3, 2]]) print(pca(X,1))
结果:
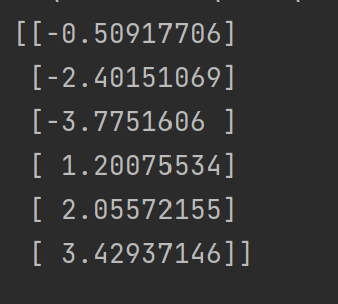
(2)sklearn的PCA实现:
1.sklearn.decomposition.PCA参数介绍
1)n_components:指定PCA降维后的特征维数。n_components是一个(0,1]之间的数。将参数设置为"mle", 此时PCA类会用MLE算法根据特征的方差分布情况自己去选择一定数量的主成分特征来降维。默认值为:n_components=min(样本数,特征数)。
2)whiten :判断是否进行白化。所谓白化,就是对降维后的数据的每个特征进行归一化,让方差都为1.对于PCA降维本身来说,一般不需要白化。如果你PCA降维后有后续的数据处理动作,可以考虑白化。默认值是False,即不进行白化。
3)svd_solver:指定奇异值分解SVD的方法。一般的PCA库都是基于SVD实现的。有4个可以选择的值:{‘auto’, ‘full’, ‘arpack’, ‘randomized’}。randomized一般适用于数据量大,数据维度多同时主成分数目比例又较低的PCA降维,它使用了一些加快SVD的随机算法。 full则是传统意义上的SVD,使用了scipy库对应的实现。arpack和randomized的适用场景类似,区别是randomized使用的是scikit-learn自己的SVD实现,而arpack直接使用了scipy库的sparse SVD实现。默认是auto,即PCA类会自己去在前面讲到的三种算法里面去权衡,选择一个合适的SVD算法来降维。一般来说,使用默认值就够了。
explained_variance_:它代表降维后的各主成分的方差值。方差值越大,则说明越是重要的主成分。
explained_variance_ratio_:它代表降维后的各主成分的方差值占总方差值的比例,这个比例越大,则越是重要的主成分。
另:
fit(): Method calculates the parameters μ and σ and saves them as internal objects.
解释:求得训练集X的均值,方差,最大值,最小值,这些训练集X固有的属性。
transform(): Method using these calculated parameters apply the transformation to a particular dataset.
解释:在fit的基础上,进行标准化,降维,归一化等操作(看具体用的是哪个工具,如PCA,StandardScaler等)。
fit_transform(): joins the fit() and transform() method for transformation of dataset.
解释:fit_transform是fit和transform的组合,既包括了训练又包含了转换。
transform()和fit_transform()二者的功能都是对数据进行某种统一处理(比如标准化~N(0,1),将数据缩放(映射)到某个固定区间,归一化,正则化等)
fit_transform(trainData)对部分数据先拟合fit,找到该part的整体指标,如均值、方差、最大值最小值等等(根据具体转换的目的),然后对该trainData进行转换transform,从而实现数据的标准化、归一化等等。
2.程序
##用sklearn的PCA import numpy as np from sklearn.decomposition import PCA X = np.array([[-1, -1], [-2, -1], [-3, -2], [1, 1], [2, 1], [3, 2]]) pca = PCA(n_components=1) newX = pca.fit_transform(X) invX = pca.inverse_transform(X) # 将降维后的数据转换成原始数据 print(newX) print(invX) print(pca.explained_variance_ratio_)
结果:
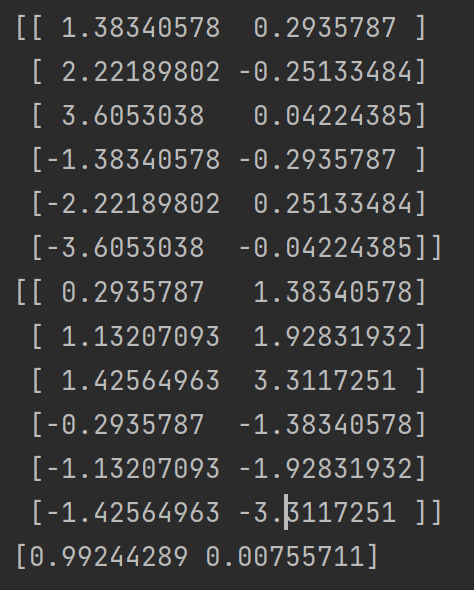
对比结果:
sklearn中的PCA是通过svd_flip函数实现的,sklearn对奇异值分解结果进行了一个处理,因为ui*σi*vi=(-ui)*σi*(-vi),也就是u和v同时取反得到的结果是一样的,而这会导致通过PCA降维得到不一样的结果(虽然都是正确的)。具体了解可以分析一下sklearn中关于PCA的源码。
出现问题:路径种含有中文
解决方法:windows10系统修改c盘user文件夹下的计算机名称
2.matlab
(1)PCA的matlab实现:
注意:直接调用PCA函数时不包括对数据标准化。
load('data.mat');
X=traces(1:1000,:);%1000X6000
[m,n]=size(X); %计算矩阵的行m和列n
%-------------第一步:标准化矩阵-----------------%
mv=mean(X); %计算各变量的均值
st=std(X); %计算各变量的标准差
X=(X-repmat(mv,m,1))./repmat(st,m,1); %标准化矩阵X
%z=zscore(X)
%-------------第二步:计算相关系数矩阵-----------------%
% R1=X'*X/(m-1); %方法一:协方差矩阵计算公式
% R2=cov(X); %方法二:协方差矩阵计算函数
R=corrcoef(X); %方法三:相关系数矩阵函数
%-------------第三步:计算特征向量和特征值-----------------%
[V,D]=eig(R); %计算矩阵R的特征向量矩阵V和特征值矩阵D,特征值由小到大
V=(rot90(V))'; %将特征向量矩阵V从大到小排序
D=rot90(rot90(D)); %将特征值矩阵由大到小排序
E=diag(D); %将特征值矩阵转换为特征值向量
%-------------第四步:计算贡献率和累计贡献率-----------------%
ratio=0; %累计贡献率
for k=1:n
r=E(k)/sum(E); %第k主成份贡献率
ratio=ratio+r; %累计贡献率
if(ratio>=0.9) %取累计贡献率大于等于90%的主成分
break;
end
end
%-------------第五步:计算得分-----------------%
S=X*V;
load('atk_data.mat');
X=atk_traces;%能量迹条数 eps一个很小的正数:2.2204e-16
meanValue=ones(size(X,1),1)*mean(X);
X=X-meanValue;%每个维度减去该维度的均值
C=X'*X/(size(X,1)-1);%计算协方差矩阵
%计算特征向量,特征值
[V,D]=eig(C);
%将特征向量按降序排序
[dummy,order]=sort(diag(-D));
V=V(:,order);%将特征向量按照特征值大小进行降序排列
d=diag(D);%将特征值取出,构成一个列向量
newd=d(order);%将特征值构成的列向量按降序排列
T=V(:,1:600);%取前cols个特征向量,构成变换矩阵T
newX=X*T;%用变换矩阵T对X进行降维
(2)直接调用PCA的matlab实现:
load('data.mat');
X=traces(1:1000,:);
[coeff, score, latent, tsquared, explained, mu]=pca(X);
a=cumsum(latent)./sum(latent); % 计算特征的累计贡献率
% explained和latent均可用来计算降维后取多少维度能够达到自己需要的精度,且效果等价。
% explained=100*latent./sum(latent);
idx=find(a>0.9); % 将特征的累计贡献率不小于0.9的维数作为PCA降维后特征的个数
k=idx(1);
Feature=score(:,1:k); % 取转换后的矩阵score的前k列为PCA降维后特征
(3)fastPCA的matlab实现:
function [pcaA, V] = fastPCA(A,k)
% 快速PCA
% 输入:A --- 样本矩阵,每行为一个样本
% k --- 降维至 k 维
% 输出:pcaA --- 降维后的 k 维样本特征向量组成的矩阵,每行一个样本,列数 k 为降维后的样本特征维数
% V --- 主成分向量
[r, ~] = size(A);
% 样本均值
meanVec = mean(A);
% 计算协方差矩阵的转置 covMatT
Z = (A-repmat(meanVec, r, 1));
covMatT = Z * Z';
% 计算 covMatT 的前 k 个本征值和本征向量
[V, ~] = eigs(covMatT, k);
% 得到协方差矩阵 (covMatT)' 的本征向量
V = Z' * V;
% 本征向量归一化为单位本征向量
for i=1:k
V(:,i)=V(:,i)/norm(V(:,i));
end
% 线性变换(投影)降维至 k 维
pcaA = Z * V;
% 保存变换矩阵 V 和变换原点 meanVec
四、遇到的问题
1 如果出现NaN(Not-a-Number的简写,中文译为“非数”,表示计算结果为不定),可能是因为分母为0,可以在数据后面加上 eps,不影响结果。
X=traces+ eps;%能量迹条数 eps一个很小的正数:2.2204e-16
2 未解决:调用函数是会莫名奇妙的小于想要的维数,用自定义才能求出来。 导致原因应该是协方差的计算方法不一样。
% R1=X'*X/(size(x,1)-1); %方法一:协方差矩阵计算公式 % R2=cov(X); %方法二:协方差矩阵计算函数 % R=corrcoef(X); %方法三:相关系数矩阵函数
3.eig排序到底是从大到小还是从小到大 ????
[V,D]=eig(R); %计算矩阵R的特征向量矩阵V和特征值矩阵D
文献来源
1.理论:主成分分析(PCA)原理详解_Microstrong0305的博客-CSDN博客_pca
2.代码:12-.ipynb · ni1o1/pygeo-tutorial - Gitee.com



 浙公网安备 33010602011771号
浙公网安备 33010602011771号