聚类--KMeans
1、什么是K均值聚类
-
k均值聚类是最简单也是最常用的聚类算法之一。它试图找到代表数据特定区域的簇中心(Cluster Center)
-
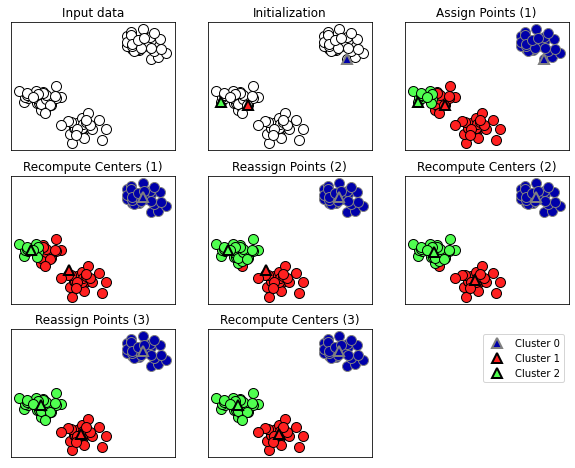
K-means算法过程
1.随机布置K个特征空间内的点作为初始的聚类中心
2.对于根据每个数据的特征向量,从K个聚类中心中寻找距离最近的一个,并且把该数据标记为从属这个聚类中心
3.在所有的数据都被标记过聚类中心之后,根据这些数据新分配的类簇,重新对K个聚类中心做计算
4.如果一轮下来,所有的数据点从属的聚类中心与上一次的分配的类簇没有变化,那么可以迭代停止,否者回到第2步继续循环mglearn.plots.plot_kmeans_algorithm()
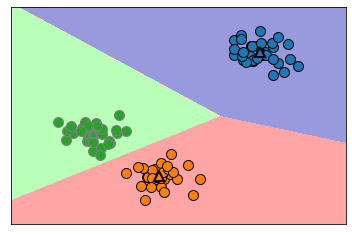
mglearn.plots.plot_kmeans_boundaries()
2、KMeans应用于模拟数据
#获取数据集
X, y = make_blobs(n_samples=100,n_features=2,random_state=42)
#Kmeans模型实例化
kmeans = KMeans(n_clusters=3,random_state=0)
kmeans=kmeans.fit(X)
#打印kmeans.labels_属性
print("labels:{}".format(kmeans.labels_))

📣
-
①算法运行期间,为 X 中的每个训练数据点分配一个簇标签。
- 在 kmeans.labels_ 属性中找到这些标签:
-
②也可以用 predict 方法为新数据点分配簇标签。
- 预测时会将最近的簇中心分配给每个新数据点,但现有模型不会改变。
- 对训练集运行 predict 会返回与 labels_ 相同的结果。
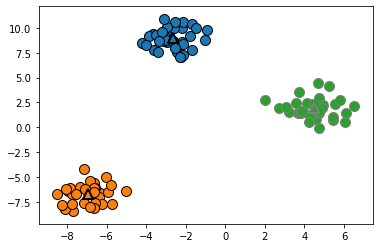
将上述的分类结果可视化
mglearn.discrete_scatter(X[:,0],X[:,1],kmeans.labels_,markers='o')
mglearn.discrete_scatter(kmeans.cluster_centers_[:,0],kmeans.cluster_centers_[:,1],[0,1,2],markers='^',markeredgewidth=2)
#使用更多或更少的簇中心
fig ,axes = plt.subplots(1,2,figsize=(10,5))
clusters=[2,4,5]
for c, axe in zip(clusters,axes):
kmeans = KMeans(n_clusters=c)
kmeans = kmeans.fit(X)
labels = kmeans.labels_
mglearn.discrete_scatter(X[:,0],X[:,1],labels,ax=axe)
3、K均值失败的案例
对于k均值,即使给定算法正确的簇的个数,也无法保证一定可以找到正确的簇。
- 因为每个簇仅仅是由其中心定义的,着意味着每个簇都是凸的(比如圆形)。所以k均值只能找到相对简单的形状。
- k 均值还假设所有簇在某种程度上具有相同的“直径”,它总是将簇之间的边界刚好画 在簇中心的中间位置。
3.1 簇的密度不同
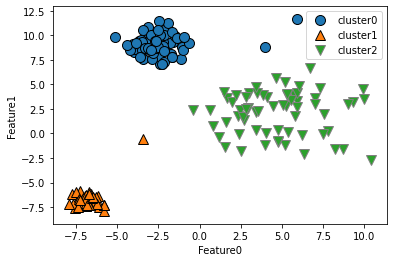
#K均值失败的案例(密度不同or非球形簇很难识别)
# 密度差别大的簇
X_varied, y_varied = make_blobs(n_samples=200,
cluster_std=[1.0,2.5,0.5],
random_state=42)
y_pred = KMeans(n_clusters=3,random_state=42).fit_predict(X_varied)
mglearn.discrete_scatter(X_varied[:,0],X_varied[:,1],y_pred)
plt.legend(["cluster0","cluster1","cluster2"],loc='best')
plt.xlabel("Feature0")
plt.ylabel("Feature1")
📣
我们直观地会认为,左下方的密集区域是第一个簇,右上方的密集区域是第二个,中间密度较小的区域是第三个。
但事实上,簇 0 和簇 1 都包含一些远离簇中其他点的点。
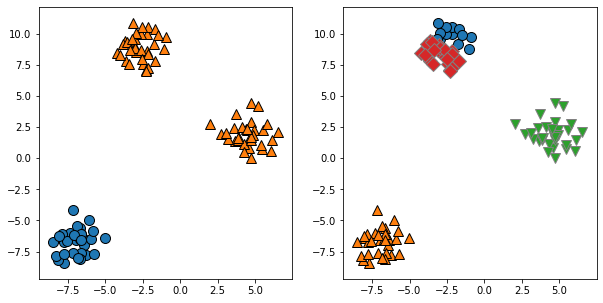
3.2 非球形簇
k 均值还假设所有方向对每个簇都同等重要。这使得它无法处理如下被沿着对角线拉长的数据集:
#非球形簇
import numpy as np
#生成随机分组的数据
X ,y = make_blobs(random_state=170,n_samples=600)
rng = np.random.RandomState(74)
#变换数据,将其拉长
transformation = rng.normal(size=(2,2))
X = np.dot(X,transformation)
#Kmeans
kmeans = KMeans(n_clusters=3,random_state=0).fit(X)
y_pred = kmeans.predict(X)
#画出分配的簇和簇中心
mglearn.discrete_scatter(X[:,0],X[:,1],y_pred)
mglearn.discrete_scatter(kmeans.cluster_centers_[:,0],kmeans.cluster_centers_[:,1],[0,1,2],markeredgewidth=2)
plt.xlabel("Feature0")
plt.ylabel("Feature1")

📣
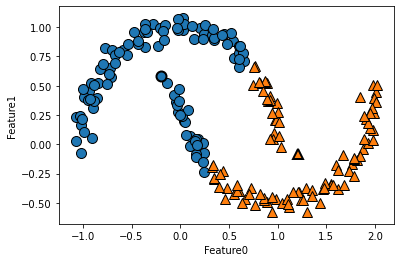
由于 k 均值仅考虑到最近簇中心的距离,所以它对上述数据集进行了错误的分类,它无法处理非球形的簇。
3.3 形状复杂的簇
如果簇的形状更加复杂,比如two_moons (sklearn.datasets自带数据集)数据,那么 k 均值的表现也很差:
from sklearn.datasets import make_moons
#形状复杂的数据
#生成随机分组的数据
X ,y = make_moons(random_state=0,n_samples=200,noise=0.05)
#Kmeans
kmeans = KMeans(n_clusters=2,random_state=0).fit(X)
y_pred = kmeans.predict(X)
#画出分配的簇和簇中心
mglearn.discrete_scatter(X[:,0],X[:,1],y_pred)
mglearn.discrete_scatter(kmeans.cluster_centers_[:,0],kmeans.cluster_centers_[:,1],[0,1],markeredgewidth=2)
plt.xlabel("Feature0")
plt.ylabel("Feature1")
4、矢量量化(把KMeans看作分解)
虽然 k 均值是一种聚类算法,但在k均值和分解方法(PCA,NMF)之间存在一些相似之处。
- PCA 试图找到数据中方差最大的方向
- NMF 试图找到累加的分量
- PCA,NMF通常对应于数据的“极值”或“部分”。试图将数据点表示为一些分量之和。
与之相反,
- k 均值则尝试利用簇中心来表示每个数据点。
- 你可以将其看作仅用一个分量来表示每个数据点,该分量由簇中心给出。
- 这种 观点将 k 均值看作是一种分解方法,其中每个点用单一分量来表示,这种观点被称为矢量量化(vector quantization)
4.1并排比较PCA、NMF、KMeans
-
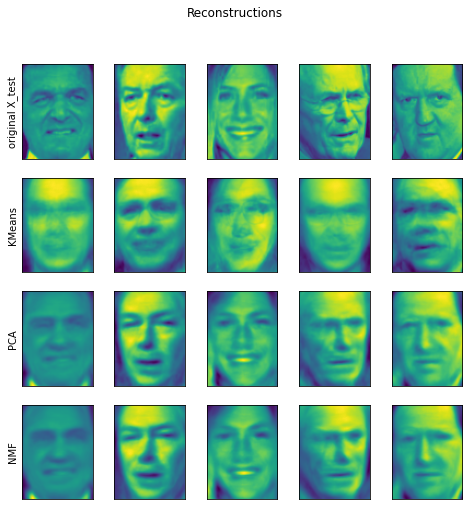
分别显示提取的100个分量;并对测试集中的人脸进行重建,
-
对于KMeans就是将测试集中的数据点找到它们最靠近的簇中心(这簇中心由训练集进行fit得到)
#矢量量化 #PCA试图找到数据中心方差最大的方向 #NMF试图将数据点看做为几个分量之和 #Kmeans看作是一种分解方法,每个点用簇中心来表示 #比较和显示PCA,NMF,KMeans提取的分量,利用100个分量对测试集中的人脸重建 from sklearn.decomposition import PCA from sklearn.decomposition import NMF from sklearn.datasets import fetch_lfw_people people = fetch_lfw_people(min_faces_per_person=20,resize=0.7) image_shape = people.images[0].shape # 降低数据偏斜,每个人最多取50张图像 mask = np.zeros(people.target.shape,dtype=np.bool) for target in np.unique(people.target): mask[np.where(people.target==target)[0][:50]]=1 #按条件查找数组元素并返回索引——np.where() X_people = people.data[mask] #mask是一个bool列表,将显示True的行选出来做X_people y_people = people.target[mask] X_people=X_people / 255 #把灰度值缩放到0-1之间 X_train,X_test,y_train,y_test=train_test_split(X_people,y_people,random_state=0) #实例化各模型,并进行拟合 nmf = NMF(n_components=100,random_state=0).fit(X_train) pca = PCA(n_components=100,random_state=0).fit(X_train) kmeans = KMeans(n_clusters=100,random_state=0).fit(X_train) #分别对测试集进行数据转化,然后重建 X_reconstruction_pca = pca.inverse_transform(pca.transform(X_test)) X_reconstruction_nmf = np.dot(nmf.transform(X_test),nmf.components_) #为啥要点乘? X_reconstruction_kmeans = kmeans.cluster_centers_[kmeans.predict(X_test)] fig, axes = plt.subplots(3,5,figsize=(8,8),subplot_kw={'xticks':(),'yticks':()}) fig.suptitle("Extracted Components") for ax, comp_kmeans, comp_pca, comp_nmf in zip( axes.T,kmeans.cluster_centers_,pca.components_,nmf.components_): ax[0].imshow(comp_kmeans.reshape(image_shape)) ax[1].imshow(comp_pca.reshape(image_shape)) ax[2].imshow(comp_nmf.reshape(image_shape)) axes[0,0].set_ylabel("KMeans簇中心的图像化") axes[1,0].set_ylabel("PCA") axes[2,0].set_ylabel("NMF") fig, axes = plt.subplots(4,5,figsize=(8,8),subplot_kw={'xticks':(),'yticks':()}) fig.suptitle("Reconstructions") for ax, orig, rec_kmeans, rec_pca, rec_nmf in zip( axes.T,X_test,X_reconstruction_kmeans,X_reconstruction_pca,X_reconstruction_nmf): ax[0].imshow(orig.reshape(image_shape)) ax[1].imshow(rec_kmeans.reshape(image_shape)) ax[2].imshow(rec_pca.reshape(image_shape)) ax[3].imshow(rec_nmf.reshape(image_shape)) axes[0,0].set_ylabel("original X_test") axes[1,0].set_ylabel("KMeans") axes[2,0].set_ylabel("PCA") axes[3,0].set_ylabel("NMF")

4.2利用KMeans的许多簇来表示复杂数据集中的变化(维数提高)
让我们回到 two_moons 数据。如果我们使用更多的簇中心,我们可以用 k 均值找到一种更具表现力的表示:
#用KMeans的transform来给数据提升维度,得到一种表现力更强的数据表示
#以make_moons为数据集
from sklearn.datasets import make_moons
X,y = make_moons(n_samples=200,noise=0.05,random_state=0)
kmeans = KMeans(n_clusters=10).fit(X)
y_pred = kmeans.predict(X)
#画图表示
plt.scatter(X[:,0],X[:,1],c=y_pred,s=60,cmap='Paired')
plt.scatter(kmeans.cluster_centers_[:,0],kmeans.cluster_centers_[:,1],
c=range(kmeans.n_clusters),s=60,linewidths=2,cmap='Paired')

📣
现在我们使用了10个簇中心,换言之,每个点现在都被分配了0到9之间的一个数字。于是我们可以构建10个新的特征 。
- 利用这个 10 维表示,现在可以用线性模型来划分两个半月形,而利用原始的两个特征是无法做到这一点的。
👍
将到每个簇中心的距离作为特征, 还可以得到一种表更好的数据表示。
- 可以利用 kmeans 的 transform 方法来完成这一点。
5、参考文献
《python机器学习基础教程》P130-P139


 浙公网安备 33010602011771号
浙公网安备 33010602011771号