降维、特征提取与流形学习--主成分分析
主成分分析(PCA)是一种旋转数据集的方法,旋转后的特征在统计上不相关。
1、什么是PCA
(1)、理解概念
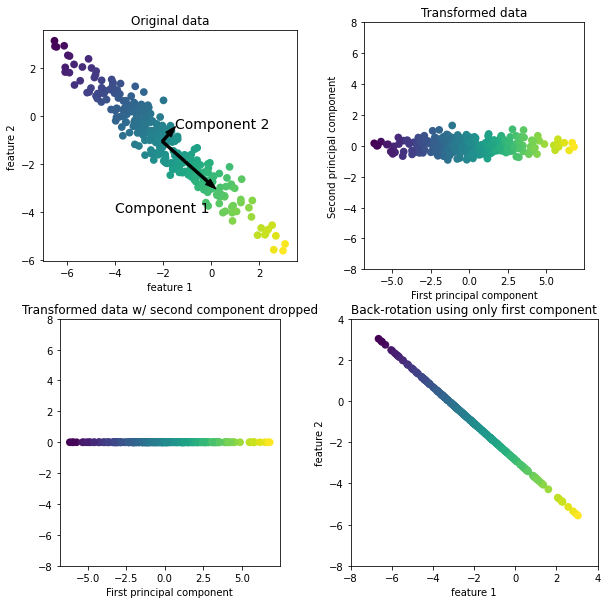
下图展示了PCA对于一个模拟二维数据集的作用
-
图一:
算法在原始数据点集中,找到方差最大的方向(包含最多信息),标记为‘成分1’。
找到与“成分1”正交(成直角)且包含最多信息的方向,标记为“成分2”。- 利用这一过程找到的方向被称为主成分(重建的最佳方向),一般主成分个数和原始特征数相同。
-
图二:从数据中减去平均值,使得变换后的数据以0为中心。
-
图三:仅保留一部分主成分(第一主成分)进行PCA降维。
-
图四:反向旋转并将平均值重新加到数据中。
(2)、主要应用
- 高维数据集可视化
- 特征提取
3、将PCA应用于cancer数据集
- 观察一下,cancer数据集中,每个特征中肿瘤类别的频次

(1)应用PCA变换
-
代码
from sklearn.preprocessing import StandardScaler X_train, X_test,y_train,y_test=train_test_split(cancer.data,cancer.target,random_state=40) #应用pca之前,对数据进行标准化 scaler = StandardScaler().fit(X_train) X_train_scaled = scaler.transform(X_train) #调用PCA进行降维 pca = PCA(n_components=2).fit(X_train_scaled) #用训练集进行拟合 print("the shape of pca components:{}".format(pca.components_.shape)) # 可以发现主成分的shape为(2,30)说明有2个主成分,每个主成分包含所有原始特征(30个)的权重信息,权重有正有负 X_train_pca = pca.transform(X_train_scaled) #对训练集进行降维,主成分为2 print("Original shape:{}".format(X_train.shape)) print("Reduced shape:{}".format(X_train_pca.shape)) -
输出

(2)变换后得到的主成分
-利用前两个主成分绘制乳腺癌数据集的二维散点图
plt.figure(figsize=(8,8))
mglearn.discrete_scatter(X_train_pca[:,0],X_train_pca[:,1],y_train)
plt.legend(cancer.target_names,loc="best")
#plt.gca().set_aspect("equal")
plt.xlabel("First component")
plt.ylabel("second component")
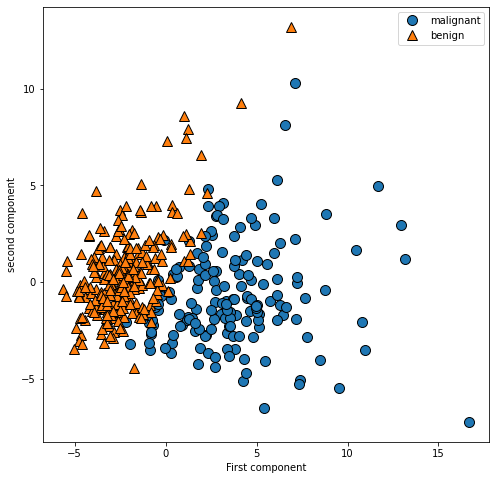
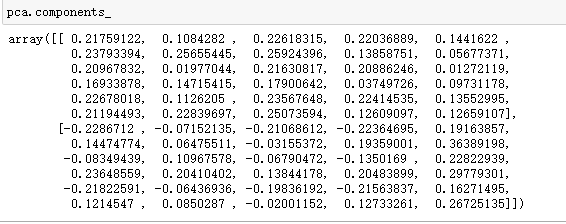
- 观察应用PCA变换后的两个主成分的属性components
-
利用热图将components进行可视化
#利用热图将pca.components 进行可视化 plt.matshow(pca.components_,cmap='viridis') #matplotlib.pyplot.matshow()画图 #热图的每行,代表每个成分,有两行表示两个主成分 #热图的每列是不同颜色的小方块,颜色深浅代表每个特征的权重大小 #添加坐标轴信息 plt.xticks(np.arange(len(cancer.feature_names)),cancer.feature_names,rotation=60,ha='left') plt.yticks([0,1],["component1","conmponent2"])

4、特征提取特征脸
特征提取:可以找到一种数据表示,比给定的原始数据更适合于分析。
像素值:图片的尺寸大小。
灰度值:指的是单个像素点的亮度。灰度值越大表示越亮。范围一般从0到255,白色为255 ,黑色为0。
(1)先了解一下数据集
- Wild数据集中的人脸图像
- 这个数据集有3023张图片,每张87*65像素,分别属于62个人,每个人照片数量不等。



- 通过上述操作,我们得到了这个数据集的基本情况,并发现这个数据集有些偏斜
(2)用PCA提取特征脸,运用KNN进行评估
目标:预测某个人脸是否属于数据库中的某个已知人物。
- 方法:
- 构建一个分类器,每个人是一个类别,但是类别的训练数据不足(同一个人图像不足),难添加新人物。
- 使用单一近邻分类器(寻找与要分类的人脸最为相同的一个)
mask = np.zeros(people.target.shape,dtype=np.bool)#降低数据偏斜,每个人最多取50张图像
for target in np.unique(people.target):
mask[np.where(people.target==target)[0][:50]]=1 #按条件查找数组元素并返回索引——np.where()
X_people = people.data[mask] #mask是一个bool列表,将显示True的行选出来做X_people
y_people = people.target[mask]
X_people=X_people / 255 #把灰度值缩放到0-1之间
-
没有使用pca
from sklearn.neighbors import KNeighborsClassifier X_train,X_test,y_train,y_test = train_test_split(X_people,y_people,random_state=0) knn = KNeighborsClassifier(n_neighbors=1).fit(X_train,y_train) print("test score:{}".format(knn.score(X_test,y_test)))
-输出
test score:0.2145748987854251
📣
精度较低,因为计算原始像素空间中的距离时,如果使用像素距离,将人脸右移一个像素将会发生巨大变化,得到一个完全不同的表示。
用像素比较两张图片是,比较的是每个像素灰度值与另一张图像对应位置的像素灰度值。
于是进行处理,沿着主成分方向的距离可以提高精度↓
-
使用PCA
启用PCA的白化选项(旋转缩放数据),将主成分缩放到相同的尺度。变换后与使用StandardScaler(每个特征的平均值为0,方差为1)相同。pca = PCA(n_components=100,whiten=True,random_state=0) pca.fit(X_train) #pca降维 X_train_pca = pca.transform(X_train) X_test_pca = pca.transform(X_test) #对降维后的数据使用knn knn = KNeighborsClassifier(n_neighbors=1).fit(X_train_pca,y_train) print("test score:{}".format(knn.score(X_test_pca,y_test))) -
输出
test score:0.27125506072874495
📣
主成分可能提供了一种更好地数据表示
5、参考文献
《Pyhton机器学习基础教程》P107-P119


 浙公网安备 33010602011771号
浙公网安备 33010602011771号