python sklearn PCA源码阅读:参数n_components的设置(设为‘mle’出错的原因)
在介绍n_components参数之前,首先贴一篇PCA参数详解的文章:http://www.cnblogs.com/akrusher/articles/6442549.html。
按照文章中对于n_components的介绍,我对一个1000x9000的array进行了主成分分析,n_components选择为"mle“,即自动选择(因为刚接触PCA,并不知道咋设置( ˇˍˇ )),尝试几次,每次都会报出下面的错误.
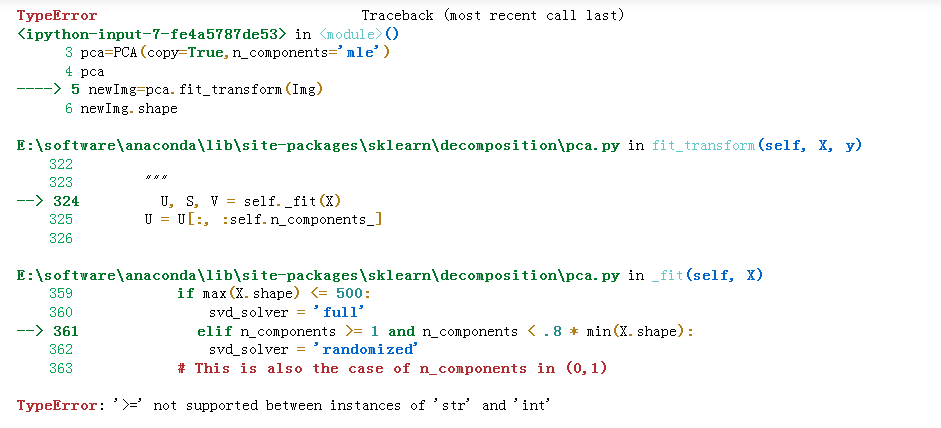
百思不得其解,终于通过阅读源码找到了原因。

就是因为svd_solver同样设置为了自动选择‘auto’,而它在选择的过程中,需要比较n_components,1,0.8*min(X.shape)的大小关系,所以会报错,所以n_components该如何设置?
n_components的设置与参数svd_solver的设置是相关联的,而它们的设置又都跟输入数据x的维度相关,通过阅读源码和说明文档总结如下。
svd_solver的设置有四种情况:'auto','fill','arpack','randomized',自动选择 'auto'的选择机制如下: x.shape>500 并且 1<=n_components<0.8*min(x.shape)时,svd_solver=‘randomsize',否则,svd_solver=’full‘。
n_components是要保留的成分,int 或者 string,缺省时默认为None,所有成分被保留,但是这三种设置并不适用于所有情况,下面的表格说明了两个参数之间的关联:
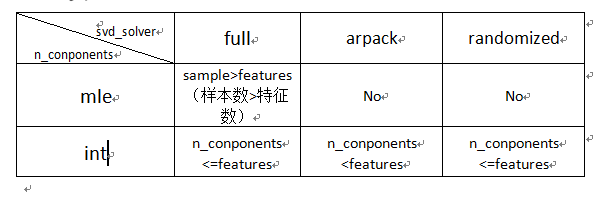
表格说明了n_components设置为‘mle’或整数时需要满足的条件,其中,“No”表示不能设置为该值。所以我们看到,只有输入数据的样本数多于特征数,并且svd_solver设置为‘full'时,才可以将n_components设置为’mle';同时注意当svd_solve设置为‘arpack’时,保留的成分必须少于特征数,即不能保留所有成分。另外,当n_components缺省时,所有成分被保留,最终保留的成分数为min(sample,features),原理在这里不做说明。


