Spark Locality Sensitive Hashing (LSH)局部哈希敏感
1、概念
LSH是一类重要的散列技术,通常用于聚类,近似最近邻搜索和大型数据集的异常检测。
LSH的一般思想是使用一个函数族(“ LSH族”)将数据点散列(hash)到存储桶中,以便彼此靠近的数据点很有可能位于同一存储桶中,而彼此相距很远的情况很可能在不同的存储桶中。
在度量空间(M,d)中,M是集合,d是M上的距离函数,LSH族是满足以下属性的函数h族:

该LSH系列称为(r1,r2,p1,p2)敏感。 在Spark中,不同的LSH系列在单独的类(例如MinHash)中实现,并且类似近似相似联接和近似最近邻居的模型在每个类中提供了用于特征转换的API。 在LSH中,我们将假阳性定义为一对距离较远输入特征(d(p,q)≥r2)散列到同一存储桶中,我们将假阴性定义为散布到不同存储桶中的一对邻近特征(d(p,q)≤r1)。
2、LSH操作
我们描述了LSH可以用于的主要操作类型。拟合的LSH模型具有用于每个操作的方法。
2.1、Feature Transformation 特征转换
特征转换是将哈希值添加为新列的基本功能。这对于降低数据维度很有用。用户可以通过设置inputCol和outputCol来指定输入和输出列的名称。 LSH还支持多个LSH哈希表。用户可以通过设置numHashTables来指定哈希表的数量。这也用于近似相似连接和近似最近邻中的OR-amplification。散列表的数量增加将提高准确性,但也会增加通信成本和运行时间。 outputCol的类型为Seq [Vector],其中数组的维数等于numHashTables,向量的维数当前设置为1。在将来的版本中,我们将实现AND-amplification,以便用户可以指定这些向量的维数。
2.2、Approximate Similarity Join 近似相似联接
近似相似联接采用两个数据集,并近似返回数据集中距离小于用户定义阈值的行对。近似相似联接既支持联接两个不同的数据集,也支持自联接。自连接会产生一些重复的对。 近似相似性联接接受已转换和未转换的数据集作为输入。如果使用未转换的数据集,它将被自动转换。在这种情况下,哈希签名将创建为outputCol。 在合并的数据集中,可以在数据集A和数据集B中查询原始数据集。距离列将添加到输出数据集中,以显示返回的每对行之间的真实距离。
2.3、Approximate Nearest Neighbor Searc 近似最近邻搜索
近似最近邻居搜索采用(特征向量的)数据集和键(单个特征向量),并近似返回数据集中最接近向量的指定行数。 近似最近邻搜索将已转换和未转换的数据集都接受为输入。如果使用未转换的数据集,它将被自动转换。在这种情况下,哈希签名将创建为outputCol。 距离列将添加到输出数据集中,以显示每个输出行和搜索到的键之间的真实距离。 注意:如果哈希存储桶中没有足够的候选者,则近似最近邻居搜索将返回少于k行的结果。
3、LSH算法
3.1 Bucketed Random Projection for Euclidean Distance 欧式距离分桶随机投影
分桶随机投影是欧氏距离的LSH族。欧几里得距离的定义如下:
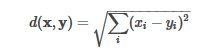
它的LSH系列将特征向量x投影到随机单位向量v上,并将投影的结果分配到哈希桶中:
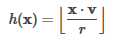
其中r是用户定义的存储区长度。存储桶长度可用于控制哈希存储桶的平均大小(以及存储桶的数量)。较大的存储桶长度(即,较少的存储桶)增加了将特征散列到同一存储桶的可能性(增加了真假性确定数)。 分桶随机投影接受任意矢量作为输入特征,并支持稀疏矢量和密集矢量。
3.2、BucketedRandomProjectionLSH code
package com.home.spark.ml import org.apache.spark.SparkConf import org.apache.spark.ml.feature.BucketedRandomProjectionLSH import org.apache.spark.ml.linalg.Vectors import org.apache.spark.sql.SparkSession import org.apache.spark.sql.functions.col /** * @Description: BucketedRandomProjectionLSH局部敏感哈希 * 为欧几里得距离度量实现局部敏感哈希函数 * * 输入是密集或稀疏向量,每个向量代表欧几里得距离空间中的一个点。 输出将是可配置尺寸的向量。 相同维中的哈希值由相同的哈希函数计算。 **/ object Ex_BucketedRandomProjectionLSH { def main(args: Array[String]): Unit = { val conf: SparkConf = new SparkConf(true).setMaster("local[2]").setAppName("spark ml") val spark = SparkSession.builder().config(conf).getOrCreate() val dfA = spark.createDataFrame(Seq( (0, Vectors.dense(1.0, 1.0)), (1, Vectors.dense(1.0, -1.0)), (2, Vectors.dense(-1.0, -1.0)), (3, Vectors.dense(-1.0, 1.0)) )).toDF("id", "features") val dfB = spark.createDataFrame(Seq( (4, Vectors.dense(1.0, 1.0)), (5, Vectors.dense(-1.0, 0.0)), (6, Vectors.dense(0.0, 1.0)), (7, Vectors.dense(0.0, -1.0)) )).toDF("id", "features") val key = Vectors.dense(1.0, 0.0) val brp = new BucketedRandomProjectionLSH().setInputCol("features").setOutputCol("hashes") //增大参数降低假阴性率,但以增加计算复杂性为代价 .setNumHashTables(3) //每个哈希存储桶的长度(较大的存储桶可降低假阴性) .setBucketLength(2.0) val model = brp.fit(dfA) // Feature Transformation println("The hashed dataset where hashed values are stored in the column 'hashes':") model.transform(dfA).show(false) // Compute the locality sensitive hashes for the input rows, then perform approximate similarity join. // We could avoid computing hashes by passing in the already-transformed dataset, // e.g. `model.approxSimilarityJoin(transformedA, transformedB, 2.5)` println("Approximately joining dfA and dfB on Euclidean distance smaller than 2.5:") model.approxSimilarityJoin(dfA, dfB, 2.5, "EuclideanDistance") .select(col("datasetA.id").alias("idA"), col("datasetB.id").alias("idB"), col("EuclideanDistance")).show() // Compute the locality sensitive hashes for the input rows, then perform approximate nearest neighbor search. // We could avoid computing hashes by passing in the already-transformed dataset, // e.g. `model.approxNearestNeighbors(transformedA, key, 2)` println("Approximately searching dfA for 2 nearest neighbors of the key:") model.approxNearestNeighbors(dfA, key, 2).show(false) spark.stop() } }
+---+-----------+-----------------------+
|id |features |hashes |
+---+-----------+-----------------------+
|0 |[1.0,1.0] |[[0.0], [0.0], [-1.0]] |
|1 |[1.0,-1.0] |[[-1.0], [-1.0], [0.0]]|
|2 |[-1.0,-1.0]|[[-1.0], [-1.0], [0.0]]|
|3 |[-1.0,1.0] |[[0.0], [0.0], [-1.0]] |
+---+-----------+-----------------------+
Approximately joining dfA and dfB on Euclidean distance smaller than 2.5:
+---+---+-----------------+
|idA|idB|EuclideanDistance|
+---+---+-----------------+
| 0| 6| 1.0|
| 0| 5| 2.23606797749979|
| 1| 5| 2.23606797749979|
| 1| 7| 1.0|
| 3| 5| 1.0|
| 3| 6| 1.0|
| 2| 7| 1.0|
| 2| 5| 1.0|
| 3| 4| 2.0|
| 0| 4| 0.0|
+---+---+-----------------+
Approximately searching dfA for 2 nearest neighbors of the key:
+---+----------+-----------------------+-------+
|id |features |hashes |distCol|
+---+----------+-----------------------+-------+
|0 |[1.0,1.0] |[[0.0], [0.0], [-1.0]] |1.0 |
|1 |[1.0,-1.0]|[[-1.0], [-1.0], [0.0]]|1.0 |
+---+----------+-----------------------+-------+
3.3、MinHash for Jaccard Distance
杰卡德距离(Jaccard Distance) 是用来衡量两个集合差异性的一种指标,它是杰卡德相似系数的补集,被定义为1减去Jaccard相似系数。而杰卡德相似系数(Jaccard similarity coefficient),也称杰卡德指数(Jaccard Index),是用来衡量两个集合相似度的一种指标。

MinHash是Jaccard距离的LSH系列,其中输入特征是自然数集。两组的Jaccard距离由其交集和并集的基数定义:
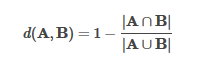
MinHash将随机哈希函数g应用于集合中的每个元素,并采用所有哈希值中的最小值:
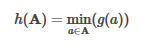
MinHash的输入集表示为二进制向量,其中向量索引表示元素本身,向量中的非零值表示该元素在集合中的存在。虽然同时支持密集和稀疏向量,但通常建议使用稀疏向量以提高效率。
例如,Vectors.sparse(10,Array [(2,1.0),(3,1.0),(5,1.0)])表示空间中有10个元素。该集合包含elem 2,elem 3和elem5。所有非零值都被视为二进制“ 1”值。 注意:MinHash不能转换空集,这意味着任何输入向量必须至少具有1个非零条目。
3.4、code MinHashLSH
package com.home.spark.ml import org.apache.spark.SparkConf import org.apache.spark.ml.feature.MinHashLSH import org.apache.spark.ml.linalg.Vectors import org.apache.spark.sql.SparkSession import org.apache.spark.sql.functions.col /** * @Description: LSH class for Jaccard distance. **/ object Ex_MinHash { def main(args: Array[String]): Unit = { val conf: SparkConf = new SparkConf(true).setMaster("local[2]").setAppName("spark ml") val spark = SparkSession.builder().config(conf).getOrCreate() val dfA = spark.createDataFrame(Seq( (0, Vectors.sparse(6, Seq((0, 1.0), (1, 1.0), (2, 1.0)))), (1, Vectors.sparse(6, Seq((2, 1.0), (3, 1.0), (4, 1.0)))), (2, Vectors.sparse(6, Seq((0, 1.0), (2, 1.0), (4, 1.0)))) )).toDF("id", "features") val dfB = spark.createDataFrame(Seq( (3, Vectors.sparse(6, Seq((1, 1.0), (3, 1.0), (5, 1.0)))), (4, Vectors.sparse(6, Seq((2, 1.0), (3, 1.0), (5, 1.0)))), (5, Vectors.sparse(6, Seq((1, 1.0), (2, 1.0), (4, 1.0)))) )).toDF("id", "features") dfA.show(false) dfB.show(false) val key = Vectors.sparse(6, Seq((1, 1.0), (3, 1.0))) val mh = new MinHashLSH() .setNumHashTables(5) .setInputCol("features") .setOutputCol("hashes") val model = mh.fit(dfA) // Feature Transformation println("The hashed dataset where hashed values are stored in the column 'hashes':") model.transform(dfA).show(false) // Compute the locality sensitive hashes for the input rows, then perform approximate similarity join. // We could avoid computing hashes by passing in the already-transformed dataset, // e.g. `model.approxSimilarityJoin(transformedA, transformedB, 0.6)` println("Approximately joining dfA and dfB on Jaccard distance smaller than 0.6:") model.approxSimilarityJoin(dfA, dfB, 0.6, "JaccardDistance") .select(col("datasetA.id").alias("idA"), col("datasetB.id").alias("idB"), col("JaccardDistance")).show(false) // Compute the locality sensitive hashes for the input rows, then perform approximate nearest neighbor search. // We could avoid computing hashes by passing in the already-transformed dataset, // e.g. `model.approxNearestNeighbors(transformedA, key, 2)` // It may return less than 2 rows when not enough approximate near-neighbor candidates are found. println("Approximately searching dfA for 2 nearest neighbors of the key:") model.approxNearestNeighbors(dfA, key, 2).show(false) spark.stop() } }
The hashed dataset where hashed values are stored in the column 'hashes':
+---+-------------------------+---------------------------------------------------------------------------------+
|id |features |hashes |
+---+-------------------------+---------------------------------------------------------------------------------+
|0 |(6,[0,1,2],[1.0,1.0,1.0])|[[2.25592966E8], [6.5902527E7], [2.82845246E8], [4.95314097E8], [7.01119548E8]] |
|1 |(6,[2,3,4],[1.0,1.0,1.0])|[[2.25592966E8], [4.98143035E8], [4.76528358E8], [1.247220523E9], [1.64558731E8]]|
|2 |(6,[0,2,4],[1.0,1.0,1.0])|[[2.25592966E8], [6.5902527E7], [2.82845246E8], [4.95314097E8], [6.65063373E8]] |
+---+-------------------------+---------------------------------------------------------------------------------+
Approximately joining dfA and dfB on Jaccard distance smaller than 0.6:
+---+---+---------------+
|idA|idB|JaccardDistance|
+---+---+---------------+
|1 |4 |0.5 |
|0 |5 |0.5 |
|1 |5 |0.5 |
|2 |5 |0.5 |
+---+---+---------------+
Approximately searching dfA for 2 nearest neighbors of the key:
+---+-------------------------+---------------------------------------------------------------------------------+-------+
|id |features |hashes |distCol|
+---+-------------------------+---------------------------------------------------------------------------------+-------+
|1 |(6,[2,3,4],[1.0,1.0,1.0])|[[2.25592966E8], [4.98143035E8], [4.76528358E8], [1.247220523E9], [1.64558731E8]]|0.75 |
+---+-------------------------+---------------------------------------------------------------------------------+-------+
4、LSH 是一项有大量应用方向的多功能技术
近似重复的检测: LSH 通常用于对大量文档,网页和其他文件进行去重处理。
全基因组的相关研究:生物学家经常使用 LSH 在基因组数据库中鉴定相似的基因表达。
大规模的图片搜索: Google 使用 LSH 和 PageRank 来构建他们的图片搜索技术VisualRank。
音频/视频指纹识别:在多媒体技术中,LSH 被广泛用于 A/V 数据的指纹识别。



 浙公网安备 33010602011771号
浙公网安备 33010602011771号