[学习笔记] MRF 入门
MRF马尔可夫随机场入门
Intro
MRF是一种广泛应用于图像分割的模型,当然我看到MRF的时候并不是因为分割,而是在图像生成领域,有的paper利用MRF模型来生成图像,因此入门一下MRF,并以分割模型为例记一下代码。
Model
Target
在图像分割中,我们的任务是给定一张图像,输出每个像素的标签。因此我们就是要得到在给定图片特征之下,标签概率最大化时所对应的标签。
因此可以这么建模:
其中w表示标签,f表示图像特征,求最大后验概率。
根据贝叶斯理论,上式右边可以写成:
其中,P(f)不含参,对数据集而言是常量。因此,上式只取决于分子部分。分子又可以表达为\(P(f,\omega)\),所以我们直接建模的其实是这个部分,计算的也是这个部分,这是与CRF不同的一点(MRF是直接对左边建模,不分解为右边,所以没个样本都要算一遍后验概率,然后乘起来最大化,MRF其实是通过对等式右边分子建模"曲线救国")。最终的建模量是联合概率。
因此,我们的任务中只需要对分子的两个部分进行定义即可。
Neighbors
像素Neighbors的定义很简单,就是这个像素周围的其他像素。
举例而言,下图分别是中心点像素的四邻域和八邻域。

Hammersley-Clifford Theorem
定理的内容为:
如果一个分布\(P(x)>0\)满足无向图\(G\)中的局部马尔可夫性质,当且仅当\(P(x)\)可以表示为一系列定义在最大团上的非负函数的乘积形式,即:
其中\(C\)为\(G\)中最大团集合,也就是所有的最大团组成的集合,\(\phi(x_c) \ge 0\)是定义在团\(c\)上的势能函数,Z是配分函数,用来将乘积归一化为概率的形式。
无向图模型与有向图模型的一个重要区别就是配分函数Z。
Hammersley-Clifford Theorem表明,无向图模型和吉布斯分布是一致的,所以将\(P(\omega)\)定义下式:
其中,Z作为normalization项,\(Z = \sum exp(-U(\omega))\),U定义为势能,而等号最右边将U变成了V的求和,在后面我们会说到,这里其实是每个原子团的势能的求和。
Clique
Clique就是我们上面提到的“团”的概念。集合\(c\)是\(S\)的原子团当且仅当c中的每个元素都与该集合中的其他元素相邻。那么Clique就是所有\(c\)的并集。
举例而言:
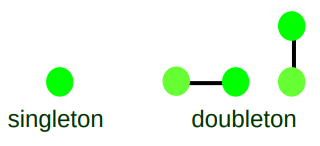
一个像素的四邻域及他自己组成的集合的原子团可以分为singleton和doubleton如图所示。
Clique Potential
翻译过来就是势能,用\(V(w)\)表示,描述的是一个Clique的能量。
那么,一个像素的领域的势能就是每个团的能量的和。
其中c表示原子团,c表示Clique,V是如何定义的呢?
在图像分割中,可以以一阶团为例,
到这里,\(P(\omega)\)的所有变量解释完了,下一步是计算\(P(f|\omega)\)
\(P(f|\omega)\)的计算
\(P(f|\omega)\)被认为是服从高斯分布的,也就是说,如果我们知道了这个像素的标签是什么,那么他的像素值应该服从这个标签下的条件概率的高斯分布。其实他服从高斯分布还是很好理解的,我们已知这个像素点的label比如说是A,那么我们去统计一下所有标签是A的点的像素值的均值和方差,显然以这个均值和方差为参数的高斯分布更加契合这里的条件分布。
计算每个类别的像素均值和方差,带入公式,即得条件概率。
最后,就是最大化\(P(\omega)P(f|\omega)\),以对数形式转化为求和的形式去优化,最大化\(log(P(\omega)) + log(P(f|\omega))\).
Coding
import numpy as np
import cv2 as cv
import copy
class MRF():
def __init__(self,img,max_iter = 100,num_clusters = 5,init_func = None,beta = 8e-4):
self.max_iter = max_iter
self.kernels = np.zeros(shape = (8,3,3))
self.beta = beta
self.num_clusters = num_clusters
for i in range(9):
if i < 4:
self.kernels[i,i//3,i%3] = 1
elif i > 4:
self.kernels[i-1,i//3,i%3] = 1
self.img = img
if init_func is None:
self.labels = np.random.randint(low = 1,high = num_clusters + 1,size = img.shape,dtype = np.uint8)
def __call__(self):
img = self.img.reshape((-1,))
for iter in range(self.max_iter):
p1 = np.zeros(shape = (self.num_clusters,self.img.shape[0] * self.img.shape[1]))
for cluster_idx in range(self.num_clusters):
temp = np.zeros(shape = (self.img.shape))
for i in range(8):
res = cv.filter2D(self.labels,-1,self.kernels[i,:,:])
temp[(res == (cluster_idx + 1))] -= self.beta
temp[(res != (cluster_idx + 1))] += self.beta
temp = np.exp(-temp)
p1[cluster_idx,:] = temp.reshape((-1,))
p1 = p1 / np.sum(p1)
p1[p1 == 0] = 1e-3
mu = np.zeros(shape = (self.num_clusters,))
sigma = np.zeros(shape = (self.num_clusters,))
for i in range(self.num_clusters):
#mu[i] = np.mean(self.img[self.labels == (i+1)])
data = self.img[self.labels == (i+1)]
if np.sum(data) > 0:
mu[i] = np.mean(data)
sigma[i] = np.var(data)
else:
mu[i]= 0
sigma[i] = 1
#print(sigma[i])
#sigma[sigma == 0] = 1e-3
p2 = np.zeros(shape = (self.num_clusters,self.img.shape[0] * self.img.shape[1]))
for i in range(self.img.shape[0] * self.img.shape[1]):
for j in range(self.num_clusters):
#print(sigma[j])
p2[j,i] = -np.log(np.sqrt(2*np.pi)*sigma[j]) -(img[i]-mu[j])**2/2/sigma[j];
self.labels = np.argmax(np.log(p1) + p2,axis = 0) + 1
self.labels = np.reshape(self.labels,self.img.shape).astype(np.uint8)
print("-----------start-----------")
print(p1)
print("-" * 20)
print(p2)
print("----------end------------")
#print("iter {} over!".format(iter))
#self.show()
#print(self.labels)
def show(self):
h,w = self.img.shape
show_img = np.zeros(shape = (h,w,3),dtype = np.uint8)
show_img[self.labels == 1,:] = (0,255,255)
show_img[self.labels == 2,:] = (220,20,60)
show_img[self.labels == 3,:] = (65,105,225)
show_img[self.labels == 4,:] = (50,205,50)
#img = self.labels / (self.num_clusters) * 255
cv.imshow("res",show_img)
cv.waitKey(0)
if __name__ == "__main__":
img = cv.imread("/home/xueaoru/图片/0.jpg")
img = cv.cvtColor(img,cv.COLOR_BGR2GRAY)
img = img/255.
#img = np.random.rand(64,64)
#img = cv.resize(img,(256,256))
mrf = MRF(img = img,max_iter = 20,num_clusters = 2)
mrf()
mrf.show()
#print(mrf.kernels)
Input:

Output(num_clusters = 4):

Output(num_clusters = 2):



 浙公网安备 33010602011771号
浙公网安备 33010602011771号