DNA纳米孔测序技术
DNA测序技术迄今为止经历了三代的发展。第三代测序技术是纳米孔测序技术。
(文中内容出处作者:小灏纸 链接:https://zhuanlan.zhihu.com/p/432535702 来源:知乎)
一、Nanopore Sequencing技术重要发展阶段
- 1993年,科学家第一次实现了利用DNA分子通过α-溶血素纳米孔,这是整个纳米孔测序技术的基础和核心。
- 2008年,Jens Gundlach的团队使用MspA纳米孔成功鉴定出了单个DNA分子的序列信息。
- 2012年,利用phi29 DNA polymerase实现了自动地、较为缓慢稳定(使得过孔时产生的电信号稳定、可测量)的DNA单链过孔过程。
- 2015年,利用ONT公司提供的MinION测序仪,成功拼接出了E.coli的基因组信息,准确率高达99.4%。
- 2020年,开始出现利用纳米孔测序技术直接实现DNA序列甲基化检测的深度学习算法。
二、Nanopore Sequencing技术原理介绍
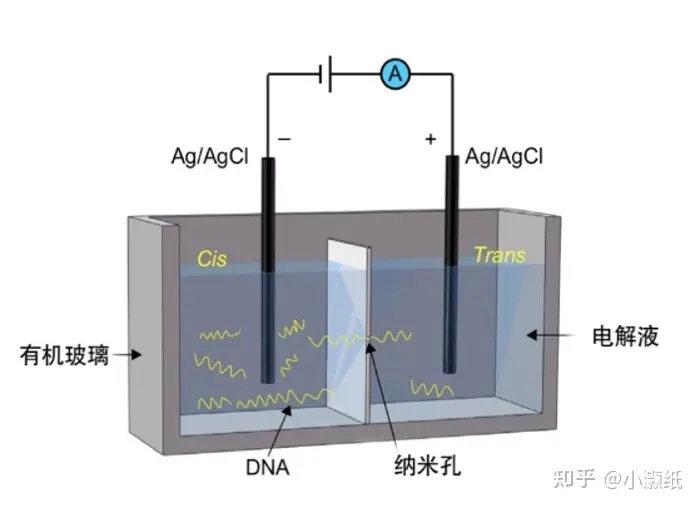
纳米孔测序技术原理如上图所示,在充斥着电解液的容器中,放置镶嵌有纳米孔蛋白的分子膜,膜上只有纳米孔可以使离子通过。首先利用外加电源在纳米孔两侧提供稳定的电势差,使得纳米孔中通过稳定的电流。然后在相关蛋白质和酶的辅助下,DNA分子以较为稳定的速度通过纳米孔,当纳米孔内被特定的核苷酸占据时会对过孔的电流产生扰动。较为直觉的解释是,较大的核苷酸占据纳米孔时,可供离子流过的空间变小,观测到的电流降低,实际过程中因为纳米孔为某种过膜蛋白,会和某些DNA分子发生偶联反应,对电流的影响机制更为复杂。最终,测序仪记录DNA分子过孔过程中产生的电流信号,再将这一特异性的电信号序列利用算法软件翻译为核苷酸序列,这一过程称为basecalling。
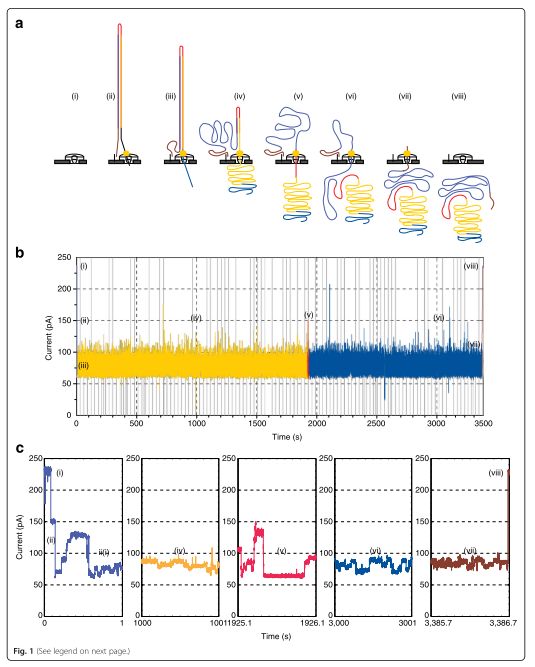
上图展示了ONT公司推出的MinION测序仪的详细工作原理。在测序之前,将接头小分子连接到基因组 DNA 或 cDNA 片段的两端。 这些接头有助于DNA链被纳米孔附近的锚点蛋白(图a中纳米孔左侧的小分子)捕获,并辅助在一条链的 5' 端加载过孔蛋白酶,即phi29 DNA polymerase等。 该酶可以在毫秒时间尺度内沿链进行单向单核苷酸位移,确保检测器可以检测到稳定、足够用于分辨核苷酸类别的电信号。
当DNA分子在过孔蛋白酶的帮助下通过纳米孔时,传感器检测到由占据孔的核苷酸序列的差异引起的电流信号的变化(如图b)。这些离子电流变化被分割为具有相关持续时间、平均振幅和方差的离散的events,然后使用basecalling算法将获取的events序列翻译为3-6个核苷酸长的 kmers(“词”)序列,这是因为纳米孔有一定厚度,电流信号的变化通常是由占据孔内的3-6个核苷酸长的序列共同决定的。
三、近年纳米孔测序技术的主要研究方向
1、研究方向:basecaller算法开发
因为电流信号的高噪声以及生物蛋白质纳米孔的差异性和纳米孔与核苷酸序列相互作用的复杂性,如何精确地将ONT生成的原始电信号翻译为序列信息(即basecalling)一直都是科学家们关注的重点,近年来也诞生了多种用于精确翻译电信号的basecaller工具。
- nanocall (MateiDavid et al. 2016)[1]:是一个开源的basecaller算法,利用了传统的机器学习算法HMM模型来刻画核苷酸序列信息和电信号之间的关系,在该算法中,观测的值是每个event的电信号,隐状态是当前event对应的k个核苷酸长度的DNA序列。其中k值为超参数,代表模型认为当前event的电信号由处于孔径内的多少个核苷酸共同决定。
- Deepnano (VladimirBozaet al. 2017)[2]:是一个利用深度学习算法来解决basecalling问题的工具,该工作借鉴了序列问题中的常用方法,利用适合于序列问题的RNN (recurrent neural network)模型来进行电信号的翻译,获得了不错的模型表现。
- Chiron (HaotianTenget al. 2018)[3]:是目前精度最高的开源basecaller算法,利用了CNN + RNN + CTC decoder的复杂深度学习架构提高了翻译精度,但计算代价比较高。
介绍的三种basecaller算法都是在ONT给出的events划分结果的基础上进行序列翻译,但在实际应用中,因为真实DNA序列上包含的大量修饰信息的存在,ONT给出的events划分结果并不一定准确,因此也有研究者开发了无需events划分信息,直接基于原始电信号进行序列翻译的basecaller,如2020年发表的工作Causalcall (Jingwen Zeng et al. 2020)[4][5]等。
- [1]David M, Dursi L J, Yao D, et al. Nanocall: an open source basecaller for Oxford Nanopore sequencing data[J]. Bioinformatics, 2017, 33(1): 49-55.
- [2]Boža V, Brejová B, Vinař T. DeepNano: deep recurrent neural networks for base calling in MinION nanopore reads[J]. PloS one, 2017, 12(6): e0178751.
- [3]Teng H, Cao M D, Hall M B, et al. Chiron: translating nanopore raw signal directly into nucleotide sequence using deep learning[J]. GigaScience, 2018, 7(5): giy037.
- [4]Zeng J, Cai H, Peng H, et al. Causalcall: Nanopore basecalling using a temporal convolutional network[J]. Frontiers in genetics, 2020, 10: 1332.
- [5]Ni P, Huang N, Zhang Z, et al. DeepSignal: detecting DNA methylation state from Nanopore sequencing reads using deep-learning[J]. Bioinformatics, 2019, 35(22): 4586-4595.
- [6]Liu Q, Fang L, Yu G, et al. Detection of DNA base modifications by deep recurrent neural network on Oxford Nanopore sequencing data[J]. Nature communications, 2019, 10(1): 1-11.
- [7]Brinkerhoff H, Kang A S W, Liu J, et al. Multiple rereads of single proteins at single–amino acid resolution using nanopores[J]. Science, 2021: eabl4381.
1. Nanocall:一款适用于牛津纳米孔测序数据的开源基因序列分析软件,由David M, Dursi L J, Yao D等人于2017年发表在"生物信息学"期刊上。
2. DeepNano:一种基于深度递归神经网络的基因序列分析软件,适用于MinION纳米孔测序数据,由Boža V, Brejová B, Vinař T等人于2017年发表在"PloS one"期刊上。
3. Chiron:一种基于深度学习技术,直接将纳米孔原始信号转换为核苷酸序列的软件,由Teng H, Cao M D, Hall M B等人于2018年发表在"GigaScience"上。
4. Causalcall:一种基于时域卷积神经网络的基因测序分析软件,适用于纳米孔测序数据的碱基识别,由Zeng J, Cai H, Peng H等人于2020年发表在"Frontiers in genetics"期刊上。
5. DeepSignal:一种检测DNA甲基化状态的软件,适用于纳米孔测序数据,采用深度学习技术实现,由Ni P, Huang N, Zhang Z等人于2019年发表在"Bioinformatics"期刊上。
6. 对牛津纳米孔测序数据中的DNA碱基修饰进行检测的软件,采用深度递归神经网络算法,由Liu Q, Fang L, Yu G等人于2019年发表在"Nature communications"期刊上。
7. Brinkerhoff:采用纳米孔测序技术,可以将单个蛋白质进行多次重读测序,并获得单个氨基酸的分辨率,由Brinkerhoff H, Kang A S W, Liu J等人于2021年发表在"Science"期刊上。
2、研究方向:甲基化检测
甲基化修饰是指甲基化酶作用于DNA分子上,形成带有甲基化功能基团或羟甲基化功能基团的核苷酸分子,比较常见的甲基化修饰有在胞嘧啶嘧啶环的五号位置上加入甲基化功能基团的5-甲基胞嘧啶(5-methylcytosine, 5mc)等。DNA分子上的甲基化修饰具有非常强的细胞特异性和细胞周期特异性,对表观遗传学研究有着重要的意义。
3、研究方向:蛋白质纳米孔测序技术
基因序列是翻译出的蛋白质序列的关键信息,但仅仅依靠基因的信息无法推测蛋白质的浓度、蛋白质的翻译后修饰(posttranslational modifications, PTMs)以及蛋白质剪切等信息,因此仅仅依靠DNA/RNA的基因组信息或者转录组学信息无法全面地描述蛋白质的表型。而纳米孔测序技术被认为是一种具有能够直接识别蛋白质表型和检测翻译后修饰的潜力的测序技术,能够为定量研究低丰度蛋白、翻译后修饰的分布和相关性等蛋白质组学的研究提供有力的支持。因此一批科学家致力于研究蛋白质序列纳米孔测序技术的可行性,并由Henry Brinkerhoff等人于2021年11月4日在Science杂志上发表了激动人心的研究成果。
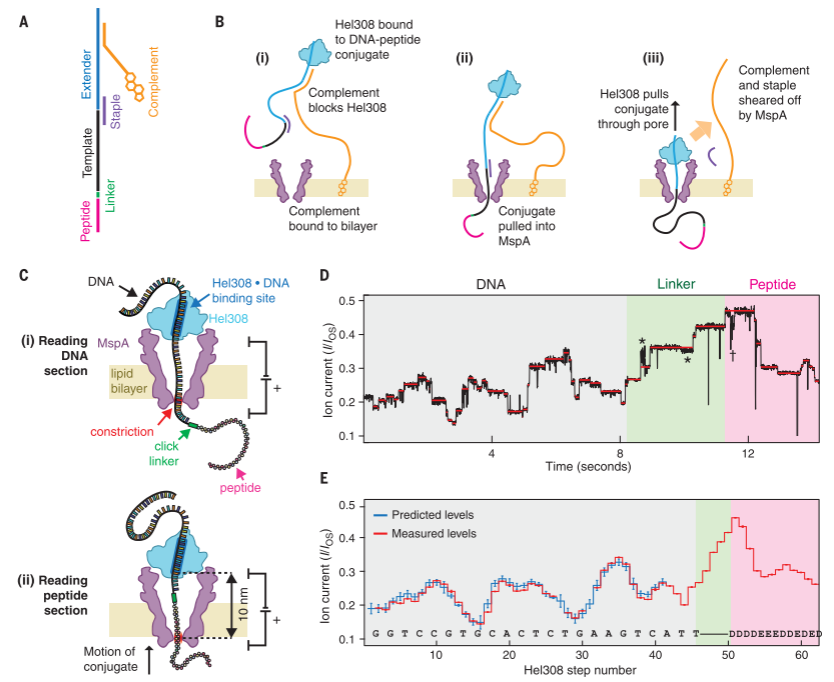
纳米孔测序技术用于蛋白质测序的关键在于找到一个合适的过孔动力蛋白,正如前面在纳米孔测序技术原理介绍部分所说,需要找到一个针对蛋白序列的具有较稳定的过孔速率的过孔动力蛋白。研究表明ClpX蛋白去折叠酶拉过纳米孔产生的信号可以区分不同的多肽分子,但这些电信号难以准确翻译为氨基酸序列,主要是ClpX产生的过孔信号速率不规则导致的。
在这篇文章中,作者仍然使用具有较为稳定过孔速率的DNA解旋酶作为过孔动力蛋白来辅助DNA-肽链聚合体通过纳米孔。在该实验方法中,待测肽链(粉红色)通过小分子的linker(绿色)和DNA分子(黑色)链接在一起,在extender(蓝色)和complement(橙色)分子的辅助作用下,该DNA-肽链聚合体可以锚定到纳米孔上,并在Hel308 DNA解旋酶(过孔动力蛋白)的作用下稳定地通过纳米孔,产生可以被检测和解读的特异性电信号序列。后续就可以利用生物信息学方法分割DNA序列和肽链的测序信息,并最终翻译得到待测肽链的氨基酸序列。
该文章为蛋白的纳米孔测序提供了一个成功的尝试,文章还有许多精细而有趣的工作:
- 利用分子动力学模拟了D、G、W氨基酸在过孔时纳米孔孔径的变化,一定程度上解释了相应氨基酸过孔时的电信号变化趋势。
- 利用re-reads操作,即对关键位点进行多次重复测量,来为关键位点提供更加准确可靠的测序结果,该方法可以一定程度缓解纳米孔测序技术本身具有的测序错误,然而会提供更长的测序信号,增加后续basecalling操作的计算负担。
该工作仍然具有一定的局限性:例如仅仅尝试了几种特定的氨基酸组成的序列,且必须依靠re-reads方法重复测序才能达到一定的翻译精度,距离真正多种氨基酸组成的复杂生物蛋白的准确测序还有一定的距离;另一方面,实验表明该方法对于正电性的肽链测序效果较差,且蛋白测序的读长还不能像DNA序列一样到达很长的水平。总体而言,本文工作为蛋白质纳米孔测序技术做出了非常重要的尝试,但距离实际的测序应用还有一段不小的距离。


