SVM算法及案例分析(SVM分类、SVM回归)
SVM算法的R语言实现
1.SVM分类
(1)标准分类模型
library(e1071) data(iris) attach(iris) model<-svm(Species~.,data=iris) #标准分类模型
(2)多分类模型
#步骤1 数据集准备
x<-subset(iris,select = Species) y<-Species
#步骤2 模型建立
model<-svm(x,y)
#步骤3 模型展示
summary(model) pred<-predict(model,x) table(pred,y)

#步骤4 多分类可视化
plot(cmdscale(dist(iris[,-5])),cex.axis=1.5,col.axis="blue",
col.lab="red",cex.lab=1.5,col=as.integer(iris[,5]),
pch=c("o","+")[1:150 %in% model$index+1],main="多分类可视化",
xlab="Species",ylab="Species")
# "+"表示支持向量,"o"表示普通样本点

2. SVM回归
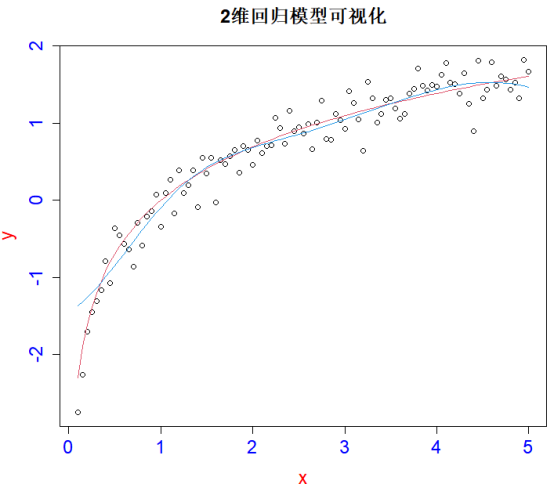
#步骤1 数据集准备
x<-seq(0.1,5,by=0.05) y<-log(x)+rnorm(x,sd=0.2)
#步骤2 模型建立
m<-svm(x,y) new<-predict(m,x) #支持向量机2维回归模型
#步骤3 模型可视化
plot(x,y,main="2维回归模型可视化",cex.axis=1.5,col.axis="blue",
col.lab="red",cex.lab=1.5,cex.main=1.5)+
points(x,log(x),col=2,type="l")+
points(x,new,col=4,type="l") #回归模型建立与可视化
3. SVM扩展包(kernlab包)
#步骤1 加载kernlab包
library(kernlab)
#步骤2 数据集准备
#svm<-ksvm(label~.,data,kernel,kpar,C,cross) x<-rbind(matrix(rnorm(120),,2),matrix(rnorm(120,mean = 3),,2)) y<-matrix(c(rep(1,60),rep(-1,60)))
#步骤3 模型建立与展示
svp<-ksvm(x,y,type="C-svc") svp

#步骤4 模型可视化
plot(svp,data=x) #得到数据的散点分类示意图

案例1:SVM算法应用与iris数据据(e1071包)
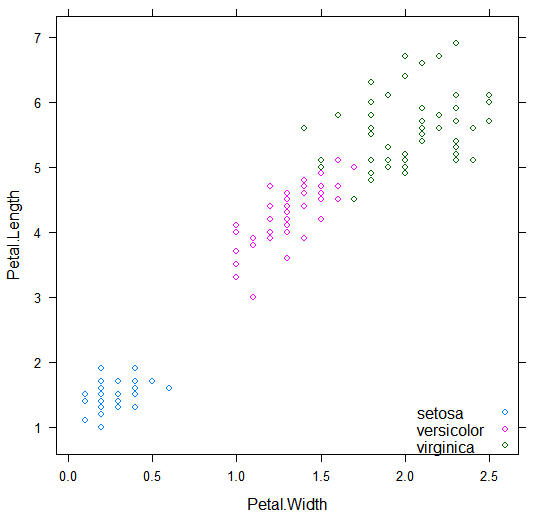
library(lattice) library(e1071) xyplot(Petal.Length~Petal.Width,data=iris,groups=Species,auto.key=list(corner=c(1,0))) #在正式建模之前,我们通过一个图形来初步判定数据的分布情况
data("iris")
attach(iris)
subdata<-iris[iris$Species!="virginica",]
subdata$Species<-factor(subdata$Species)
model<-svm(Species~Petal.Length+Petal.Width,data=subdata)
plot(model,subdata,Petal.Length~Petal.Width)

SVM算法案例分析iris数据集
#SVM算法案例
library(e1071)
data(iris)
summary(iris)
m<-svm(Species~.,data=iris) #以Species为标签,建立SVM模型
m #模型结果

x=iris[,-5] y=iris[,5] m<-svm(x,y,kernel="radial",gamma=if(is.vector(x))1 else 1/ncol(x)) summary(model) #查看m模型的相关结果

pred<-predict(m,x) #根据模型m对数据x进行预测 pred[sample(1:50,8)] #随机挑选8个预测结果进行展示

table(pred,y)

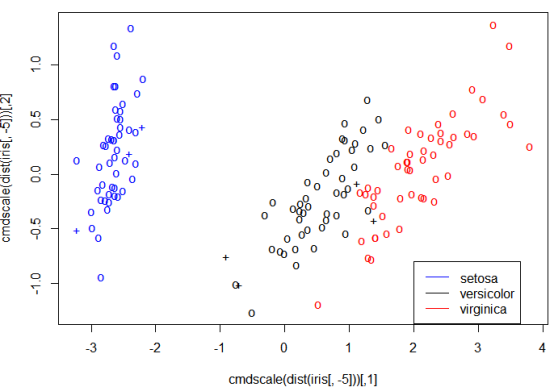
plot(cmdscale(dist(iris[,-5])),col=c("blue","black","red")
[as.integer(iris[,5])],pch=c("o","+")[1:150 %in% model$index+1]) #SVM分类作图,"+"表示支持向量,"o"表示普通样本点
legend(2,-0.8,c("setosa","versicolor","virginica"),col=c("blue","black","red"),lty=1)
#plot函数还可以从其他角度对SVM模型进行可视化分析
data(iris)
model<-svm(Species~.,data=iris)
plot(model,iris,Petal.Width~Petal.Length,fill=F,
symbolPalette=c("blue","black","red"),svSymbol="+") #绘制模型类别关于花瓣宽度核长度的分类情况
legend(1,2.5,c("setosa","versicolor","virginica"),col=c("blue","black","red"),lty=1)

案例2:SVM算法在基因表达数据的应用
library(ISLR) names(Khan)
dim(Khan$xtrain) #查看数据的维度

dim(Khan$xtest)

length(Khan$ytrain)

length(Khan$ytest)

结果分析:数据集由 2308 个基因的表达测定组成,训练集和测试集分别由 63和 20 个观测组成。
table(Khan$ytrain) #训练集中的类别及观测数目
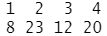
table(Khan$ytest) ##测试集中的类别及观测数目

#在这个数据集中,相对于观测的数目来说,特征的数目非常多,基于这一特点建议使用线性核函数,因为使用多项式核函数和径向核函数得到更高光滑性是没有必要的。
dat=data.frame(x=Khan$xtrain, y=as.factor(Khan$ytrain)) #训练数据集 out=svm(y~., data=dat, kernel="linear",cost=10) summary(out)

table(out$fitted, dat$y) #训练集混淆矩阵

结果分析:混淆矩阵结果显示,训练集的误装为0,事实上,这并不奇怪,因为相对于观测的数目来说,变量的数目较多意味着很容易找到把这些类别完全分开的超平面。
dat.te=data.frame(x=Khan$xtest, y=as.factor(Khan$ytest)) #测试集 pred.te=predict(out, newdata=dat.te) table(pred.te, dat.te$y) #测试集混淆矩阵

结果分析:混淆矩阵结果显示,测试集中有2个样本被错误分类了。
本文来自博客园,作者:zhang-X,转载请注明原文链接:https://www.cnblogs.com/YY-zhang/p/15990974.html




