使用传统方法(python)截断脑部MRI图像(二维)中线
使用传统方法(python)截断脑部MRI图像(二维)中线
任务描述
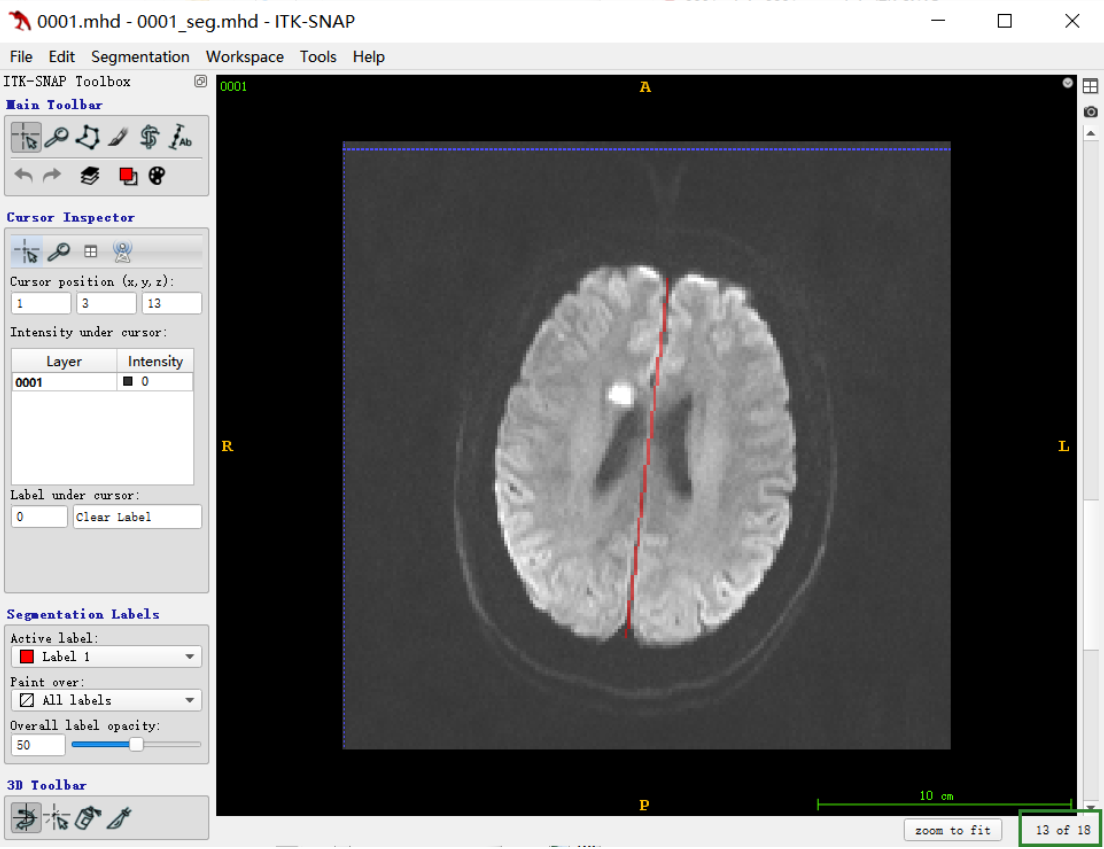
前提:根据对脑部MRI图像的每一层的中线手工标注的点,拟合出的中线,如下图:

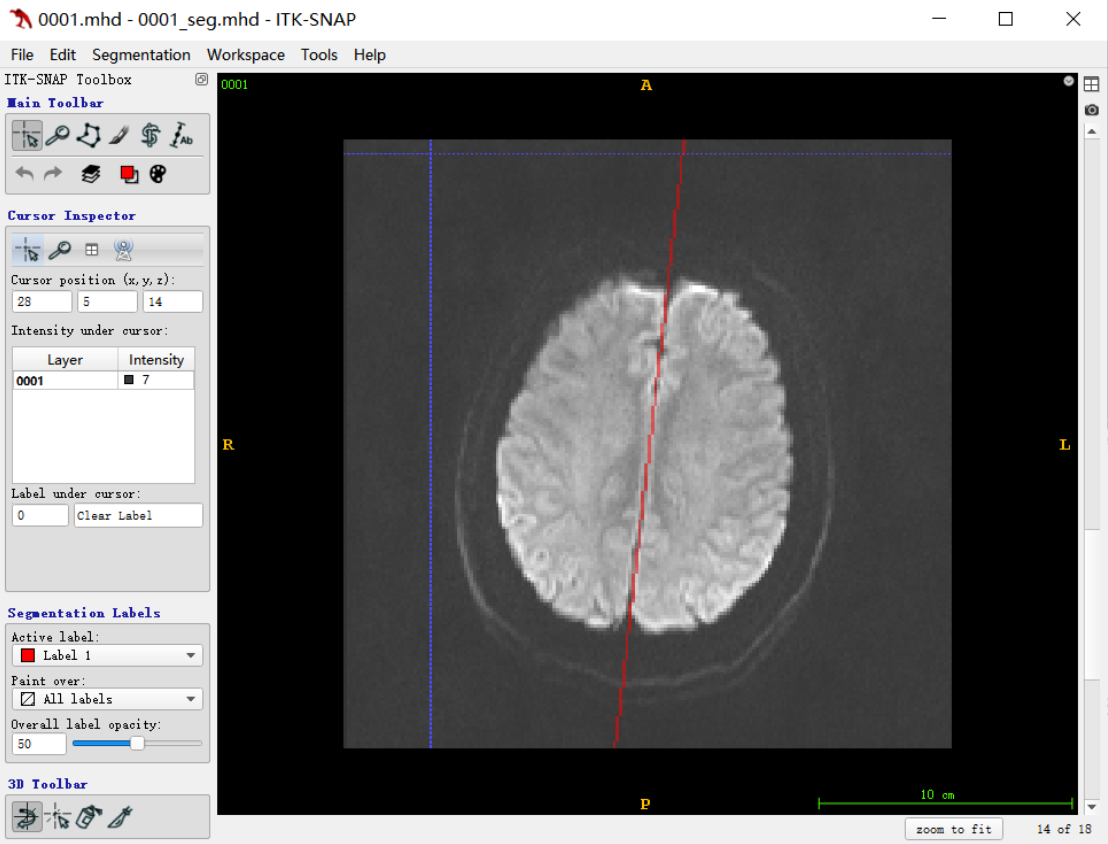
需求:截断拟合出来的中线超出脑部区域的部分,如下图中绿色框中的线段,(只留下高量区域的上端点和下端点连接的线段——中间部分),最后截断的效果需要和后面CT图像中求的线段对应起来,
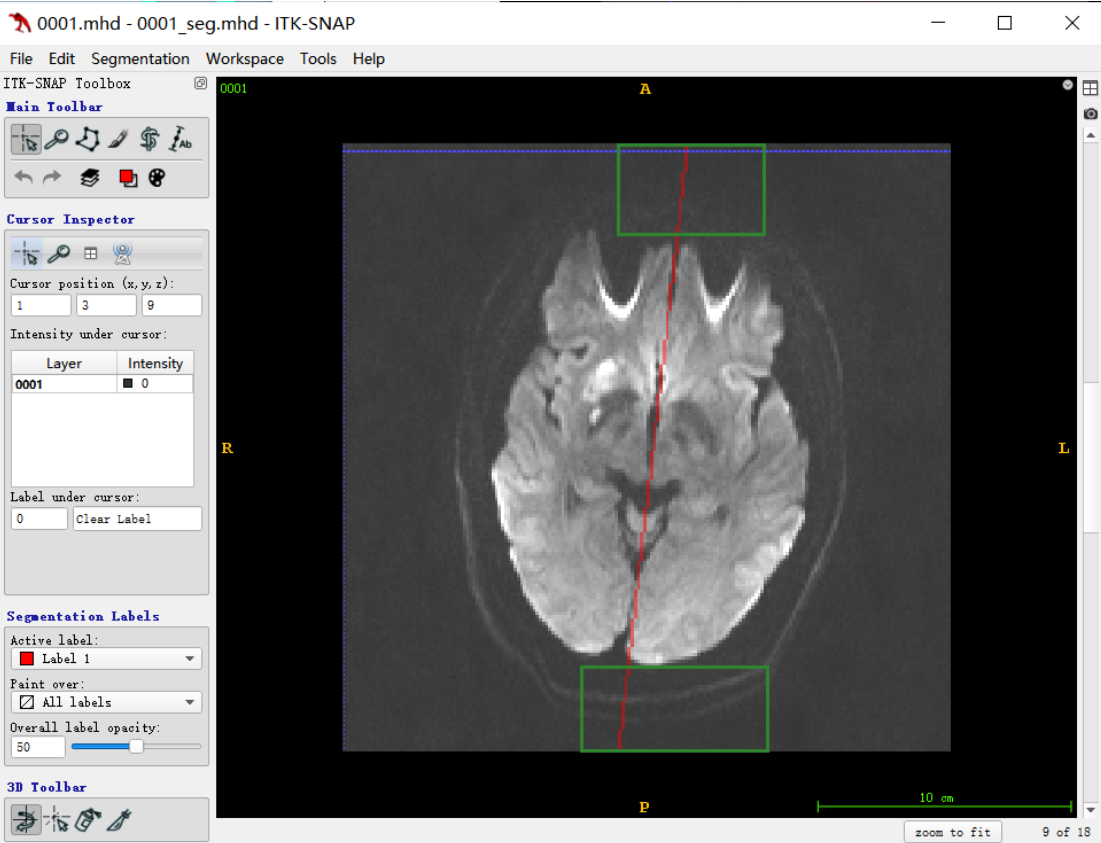
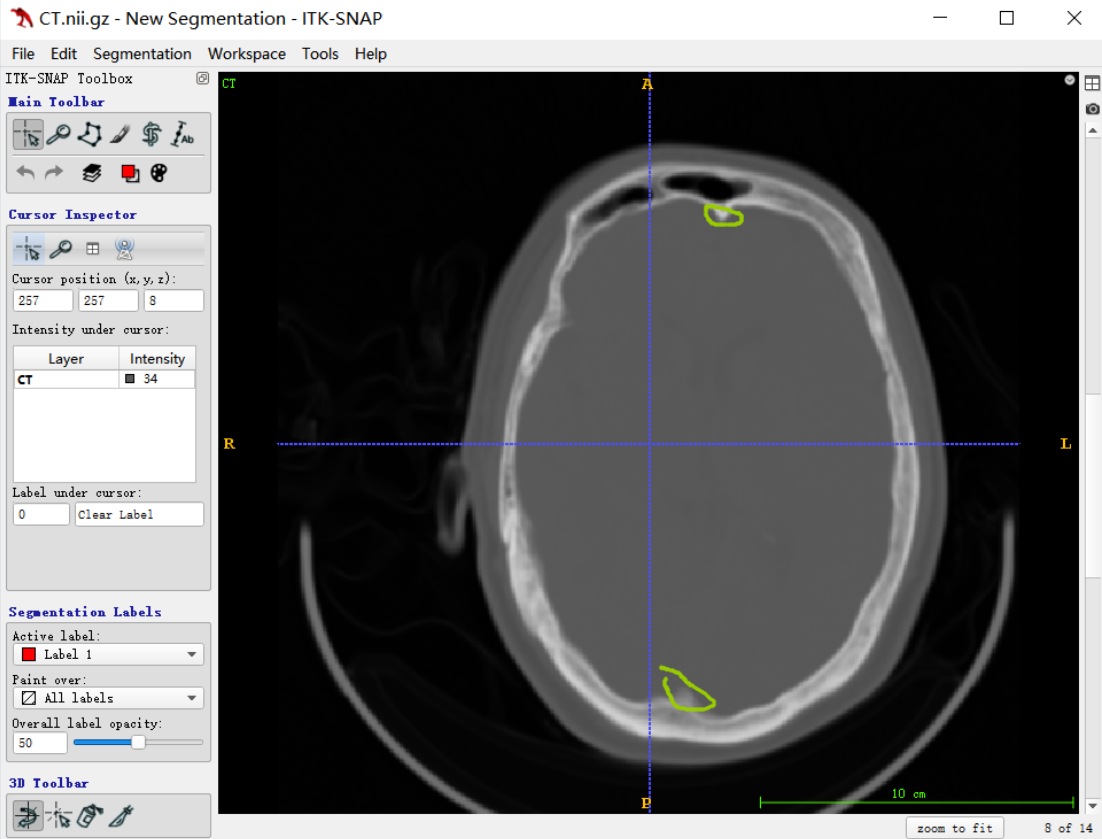
所以选要在MRI上至少截断的区域保证在颅骨的内圈,MRI的图像中我们需要在二值化时就去掉外围颅骨
效果
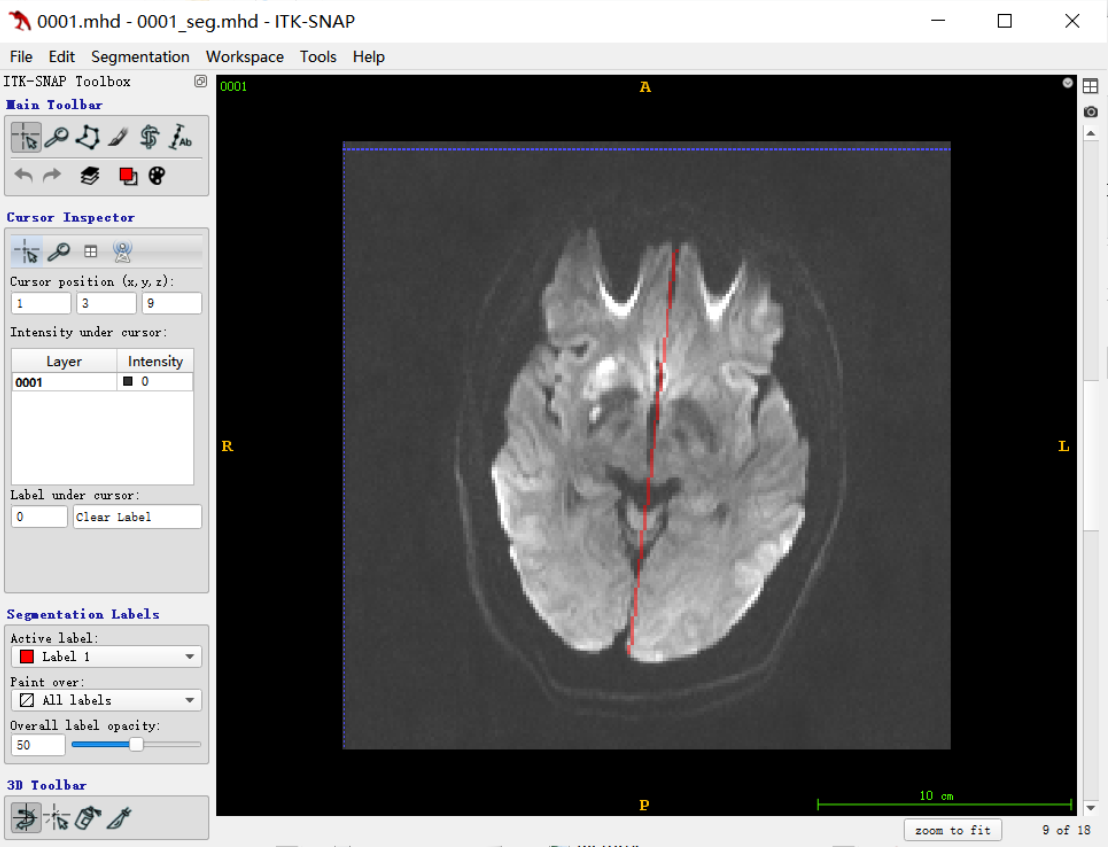
对中间层及以下的层使用传统方法截断有比较理想,中间层往上(因为鼻腔部位的像素值比较低,容易将其分割成背景)
算法流程图
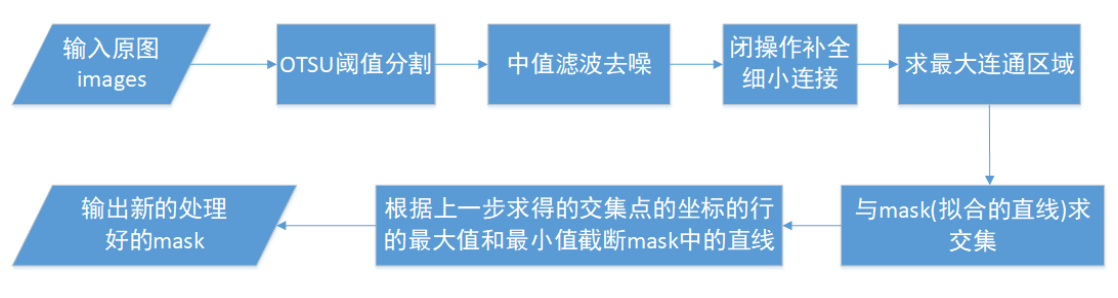
其中输入的images使用simpleITK读取相应的病例MRI的mhd文件,中值滤波器的大小设置为3,闭操作的结构元素设置为15(这些都是对数据测试时设置的经验值)
其中参考链接:
使用OTSU类间方差公式求二值化的阈值:https://blog.csdn.net/u012771236/article/details/44975831
求最大连通区域:https://blog.csdn.net/xuyangcao123/article/details/81023732
整体实现代码
import os
import glob
import cv2 as cv
import SimpleITK as sitk
import numpy as np
import matplotlib.pyplot as plt
import pytest
from numpy.core.tests.test_mem_overlap import xrange
from skimage.measure import label
def read_mhd(file_name):
'''
根据文件路径读取mhd文件,并返回其内容array、origin、spacing
'''
itkimage = sitk.ReadImage(file_name)
image_array = sitk.GetArrayFromImage(itkimage)
origin = itkimage.GetOrigin()
spacing = itkimage.GetSpacing()
return image_array, origin, spacing
def OTSU_enhance(img_gray, th_begin, th_end, th_step=1):
'''
根据类间方差最小求最适合的阈值
在th_begin、th_end中寻找合适的阈值
'''
assert img_gray.ndim == 2, "must input a gary_img"
max_g = 0
suitable_th = 0
for threshold in xrange(th_begin, th_end, th_step): # 前闭后开区间
bin_img = img_gray > threshold
bin_img_inv = img_gray <= threshold
fore_pix = np.sum(bin_img)
back_pix = np.sum(bin_img_inv)
if 0 == fore_pix:
break
if 0 == back_pix:
continue
w0 = float(fore_pix) / img_gray.size
u0 = float(np.sum(img_gray * bin_img)) / fore_pix
w1 = float(back_pix) / img_gray.size
u1 = float(np.sum(img_gray * bin_img_inv)) / back_pix
# 类间方差公式
g = w0 * w1 * (u0 - u1) * (u0 - u1)
if g > max_g:
max_g = g
suitable_th = threshold
return suitable_th
def close_demo(image, structure_size):
'''
闭操作:补全细小连接
'''
kenerl = cv.getStructuringElement(cv.MORPH_RECT, (structure_size, structure_size)) # 定义结构元素的形状和大小
# dst_1 = cv.dilate(image, kenerl) # 先膨胀
# dst = cv.erode(dst_1, kenerl) # 腐蚀操作
dst = cv.morphologyEx(image, cv.MORPH_CLOSE, kenerl) # 闭操作,与分开使用膨胀和腐蚀效果相同
return dst
def largestConnectComponent(bw_img):
'''
求bw_img中的最大联通区域
:param bw_img: 二值图
:return: 最大连通区域
'''
labeled_img, num = label(bw_img, connectivity=2, background=0, return_num=True) # 取连通区域,connectivity=2表示8领域
max_label = 0
max_num = 0
for i in range(1, num + 1): # lable的编号从1开始,防止将背景设置为最大连通域
if np.sum(labeled_img == i) > max_num:
max_num = np.sum(labeled_img == i)
max_label = i
lcc = (labeled_img == max_label) # 只留下最大连通区域,其他区域置0
y, x = np.array(np.where(lcc > 0)) # 连通区域取的是出去孔洞区域(不会存在取出来的连通区域内还包含孔洞的情况)
for index in range(len(y)):
if lcc[y[index]][x[index]] == 0:
lcc[y[index]][x[index]] = 255 # 将求得的连通区域中有孔洞的填充
return lcc
def seg_line(img, mask, threshold_min, threshold_max, structure_size):
'''
:param threshold_min: OTSU求合适阈值的低阈值
:param threshold_max: OTSU求合适阈值的高阈值
:param img: 原图
:param mask: mask
:param threshold: 二值化的阈值
:param structure_size: 闭操作的结构元素大小
:return: 截断mask中的直线
'''
# 只处理第9层以下的层
for i in range(img.shape[0]): # img和mask的层数对应
if i >= 8: # 索引从开始
binary_threshold = OTSU_enhance(img[i], threshold_min, threshold_max, 1) # 使用类间方差来计算合适的阈值
ret, binary = cv.threshold(img[i], binary_threshold, 255, cv.THRESH_BINARY) #二值化
blur = cv.medianBlur(binary, 3) # 中值滤波去噪,设置滤波器大小为3
# 在求最大连通区域前就对图像做一次闭操作,补全细小连接
img[i] = close_demo(blur, structure_size) # 结构元素的大小为15
img[i] = largestConnectComponent(img[i]) # 求img[i]的最大联通区域
# 将img和mask转成bool类型
bimg = img[i, :, :] > 0
bmask = mask[i, :, :] > 0
temp_data = bimg & bmask # 求img和mask交集
row, col = np.where(temp_data)
if len(row): # 如果交集不空
row_max = max(row)
row_min = min(row)
# 用这两个边界去截断原先的mask
mask[i, :row_min, :] = 0
mask[i, row_max:, :] = 0
else: # 若交集为空直接将mask清空
mask[i, :, :] = 0
# 取坐标索引,以下步骤暂时用不到
# row_list = row.tolist() # 将np.array转list,方便取索引
# row_max_index = row_list.index(row_max) # 取得行的最大值的索引
# row_min_index = row_list.index(row_min) # 取得行的最小值的索引
# # print('row_max_index: ', row_max_index)
# # print('row_min_index: ', row_min_index)
# # print('row_max: ', row_max)
# # print('col: ', col[row_max_index])
# top_point = (row_min, col[row_min_index]) # 上端点
# bottom_point = (row_max, col[row_max_index]) # 下端点
# # 用位运算取均值避免溢出
# middle_row = (row_min & row_max) + (row_min ^ row_max) >> 1
# middle_col = (col[row_min_index] & col[row_max_index]) + (col[row_min_index] ^ col[row_max_index]) >> 1
# middle_point = (middle_row, middle_col) # 中点坐标
# end_points = [top_point, bottom_point] # 两个端点坐标
return mask
if __name__ == "__main__":
images_dir = 'C:\\Users\\XXXX\\Desktop\\XXX\\images\\'
masks_dir = 'C:\\Users\\XXXX\\Desktop\\XXXX\\masks\\'
new_masks_save_dir = 'C:\\Users\\XXXX\\Desktop\\XXXX\\MRI_masks_seg_line\\'
images_paths = glob.glob(images_dir + '\\*.mhd')
masks_paths = images_paths.copy()
for i in range(len(images_paths)):
masks_paths[i] = masks_paths[i].replace(images_dir, masks_dir)
masks_paths[i] = masks_paths[i].replace('.mhd', '_seg.mhd')
# print(images_paths[i])
# print(masks_paths[i])
img, _, _ = read_mhd(images_paths[i]) # Z,Y,X,int16
mask, _, _ = read_mhd(masks_paths[i]) # Z,Y,X,uint16
# 此次MRI数据的像素值0-500左右
new_mask = seg_line(img, mask, 50, 100, 15) # OTSU在50到100中求合适阈值(为了去掉MRI图像中颅骨的外围(50左右)),闭操作的结构元素设置为15(测试所得经验值),
mask = sitk.GetImageFromArray(new_mask)
image_file_id = os.path.relpath(masks_paths[i], masks_dir).split('.')[0] # 取出mask文件的序号和_seg
maskSavePath = os.path.join(new_masks_save_dir, '{}.mhd'.format(image_file_id)) # 拼接mask的绝对路径
sitk.WriteImage(mask, maskSavePath) # 将新的mask保存成mhd文件
算法并不泛化
传统方法,在设置阈值(二值化阈值、滤波器大小、闭操作结构元素大小等参数)时都会导致不同数据的效果偏差,对于中间层往上这些层的数据来说,就算单独处理(中间层以上设置以上参数不同,中间层以下设置另一套参数),效果依然不尽人意(如以下的典型例子),考虑到我们这些数据用于后期对模型的训练,如果训练集处理的就是有问题的,后期模型用来预测的效果也会变差,所以我们最后在中间层往的数据直接使用ITK-SNAP手工擦除
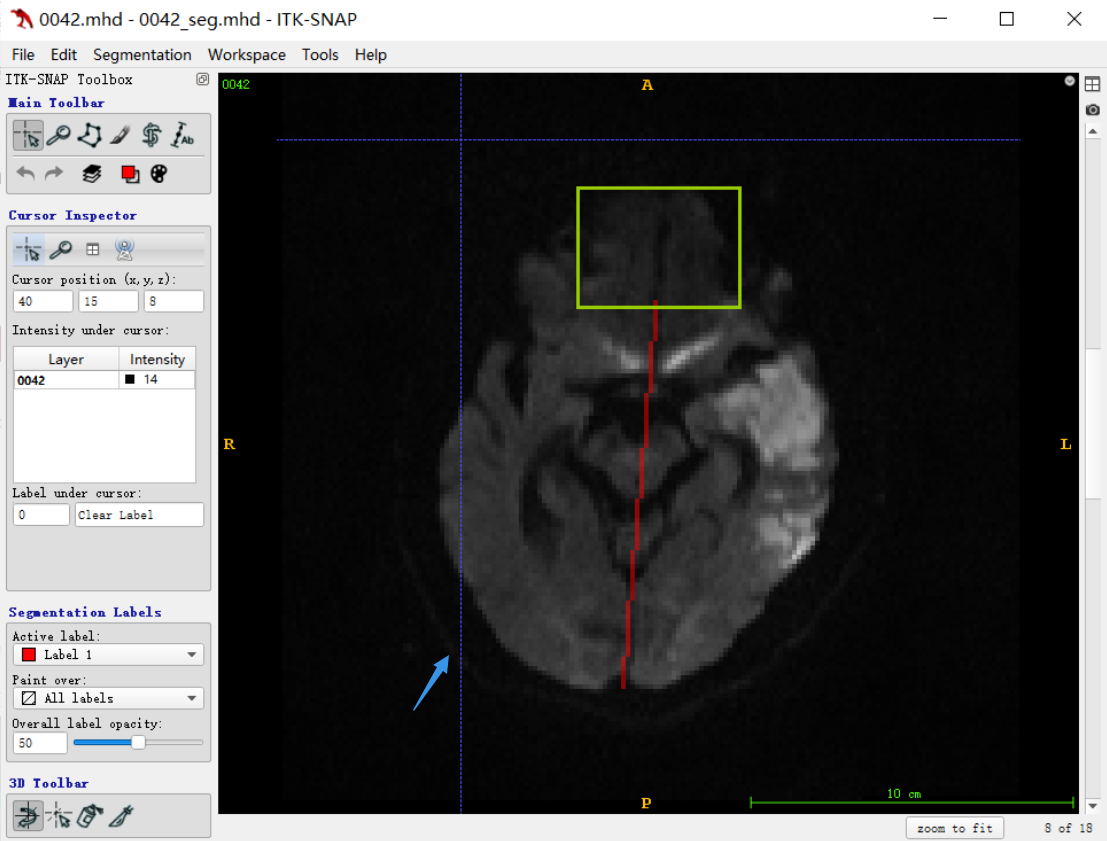
鼻腔部位像素值比颅骨外围的像素值更低,二值化以后去掉颅骨外围也将鼻腔部位去掉了
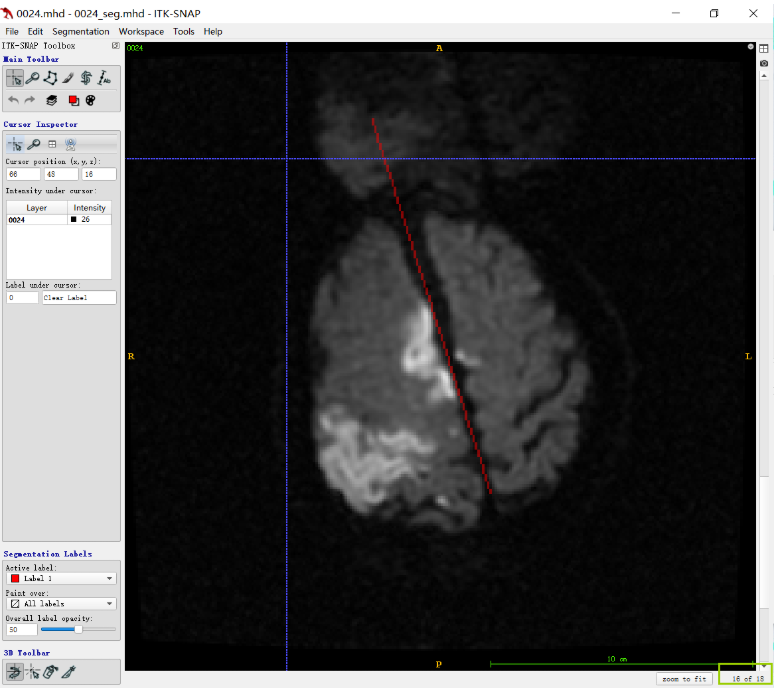
MRI原图像包含重影,导致求连通区域时,将重影一起计算到一个label,截断的线超出理想范围


 浙公网安备 33010602011771号
浙公网安备 33010602011771号