《Construction of a 3D whole organism spatial atlas by joint modelling of multiple slices with deep neural networks》 论文精读
《Construction of a 3D whole organism spatial atlas by joint modelling of multiple slices with deep neural networks》
《通过深度神经网络联合建模多个切片构建三维整体生物体空间图谱》
nature machine intelligence / 11 - September - 2023
2023.11.07
![[lab.magiconch.com][福音戰士標題生成器]-1695021721029](https://s2.loli.net/2023/09/18/zXu5EpoCmKH8FiJ.jpg)
关键创新点总结
-
基于GAT的模型构建
-
独特的切片配准和3D立体的图结构设计
简单的总结
STitch3D,巧妙地利用多个二维组织切片的数据,重构了三维的细胞结构,使我们能更全面地分析和理解复杂的生物系统。这种从二维到三维的飞跃,克服了目前主流仅依赖单个切片进行空间转录组分析的限制,无法观察到完整的三维生物过程。STitch3D通过整合多个切片的空间转录组数据,并借助单细胞RNA测序结果,实现了精确的三维细胞区域识别和细胞类型分布预测。关键的是,该方法能有效建模并消除不同切片之间的批效应,提取有意义的生物学变异,从而构建非常强大和可靠的三维转录组模型。全面的实验结果证明,STitch3D使组织乃至整个生物体尺度的三维空间转录组分析成为可能。输出结果还可支撑各类后续分析,提高对复杂生物系统的整体理解。
主要流程概述
本文主要解决两个任务:
第一个任务是识别具有生物学意义的三维空间区域,其中的点具有相似的基因表达,用来揭示组织结构。然后支持下游分析,如检测与三维空间模式相关的区域相关基因(region-related genes with 3D spatial patterns)。
第二个任务是通过整合多个空间转录组切片和单细胞RNA测序图谱(a matched scRNA-seq reference)来推断三维细胞类型分布。
它在一个统一的框架中解决了上述两个三维分析任务,以提供三维组织体系结构的互补视图。通过建模来自多个切片的基因表达和空间位置,STitch3D可以区分切片之间的生物学变异和批效应,并整合跨切片的信息来生成强大的三维组织模型。
STitch3D的主要流程:
使用多个二维ST切片来重建三维组织结构:
STitch3D的输入是多个ST切片和一个匹配的单细胞RNA测序参考库。示意如下图:
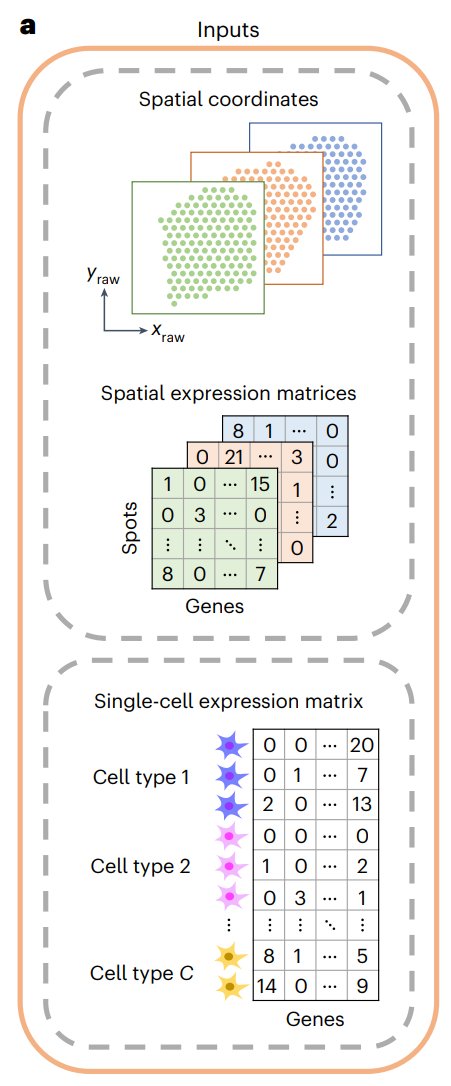
预处理步骤包括通过切片对齐、构建三维空间点坐标,以及构建全局三维邻域图:
STitch3D的主要模型结合这些结构来进行表示学习,以进行三维空间域识别和三维细胞类型解混(deconvolution)。
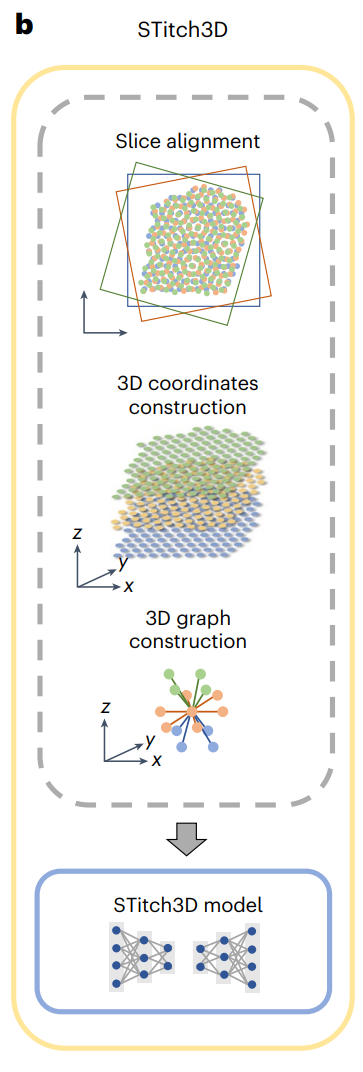
在这些步骤之后,STitch3D被训练来整合所有切片中的信息。引入一个共享的潜在空间(latent space)来提取有意义的生物学变异并促进批效应的去除。在隐空间中,每个点都有其表示,用于同时执行空间域识别和细胞类型解混任务。
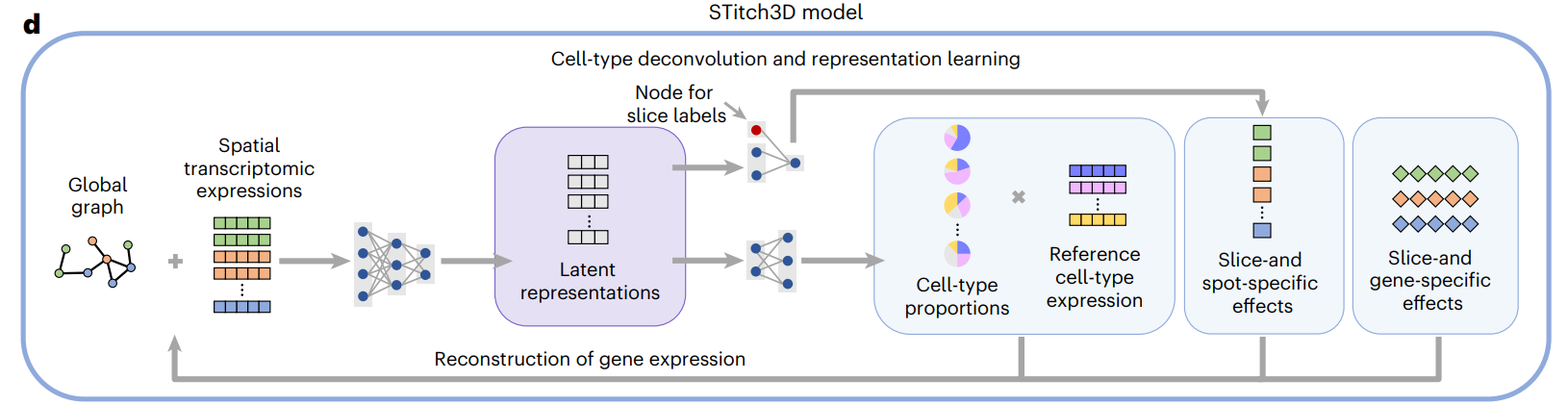
具体来说,STitch3D通过一个基于三维邻域图的图注意力网络,将多个切片中点的基因表达和空间信息映射到共享的潜在空间中。另一个神经网络\(fx\)(·)被引入来从潜在表示中推断细胞类型比例。
通过对整合多个切片的批效应建模,STitch3D被训练来重建ST基因表达,方法是将估计的细胞类型占比结果与特异性基因表达谱相结合。在训练之后,STitch3D学习到空间化的点表示和细胞类型比例。这些表示用于利用聚类算法进行三维空间域识别,而细胞类型比例等信息有助于恢复三维细胞类型分布。具体印证见result部分,这里暂且不表。
Methods
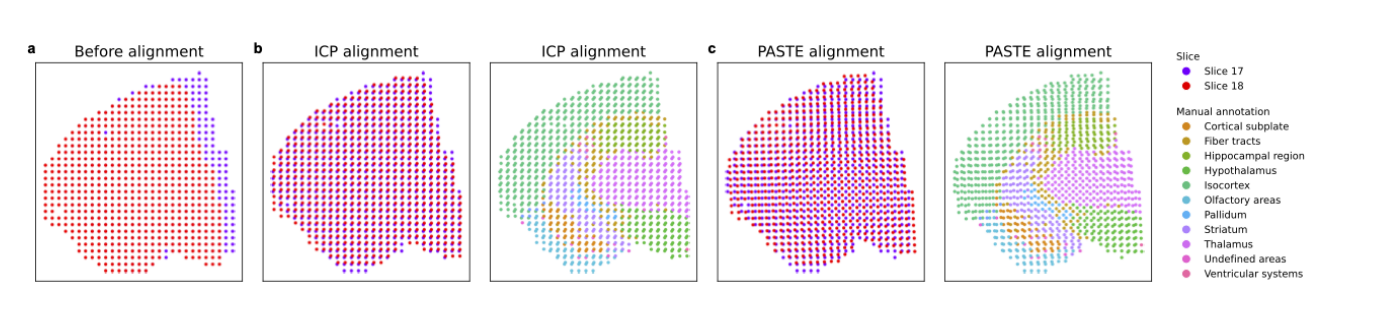
STitch3D的空间点的配准
采用迭代最近点(ICP)算法或PASTE算法将多个切片对齐。
STitch3D通过空间点的配准来对齐多个切片。默认情况下,STitch3D采用迭代最近点(ICP)算法来成对地对齐切片。在对齐之前,STitch3D根据邻域数量检测每个切片中的边缘点。例如,对于使用Visium平台获得的数据集,一个非边缘点应该有六个邻域点。为了鲁棒性,我们将邻域点数量少于五个且大于一个的点定义为边缘点(1< spot_n <5)。在使用ST平台获取的数据集中,一个非边缘点应该有八个邻域点。相应地,如果一个点有少于七个且大于一个的邻域点,则将其识别为边缘点(1< spot_n <7)。
在数据预处理完成之后,使用ICP算法成对地对齐来自两个切片的边缘点。假设边缘点通常位于组织边界上。设 \(P = {p1,p2,...pm}\)为源切片中的边缘点位置集合,\(Q = {q1,q2,...qk}\)为目标切片中的边缘点位置集合,其中\(pi,qi∈R^2\)。
ICP算法通过迭代执行以下步骤直到收敛来对齐点:
- 对源点云P中的每个点,在目标点云Q中找到其最近点(二维坐标),形成一个新的集合Q′。然后,获得包含目标点云中的点的\(Q′ = {q′1,q′2,...q′m}\)。
- 通过求解最小二乘问题找到具有最佳旋转和转换的变换:
- 将变换\(\hat{\mathrm{R}}\text{p}_i+\hat{\mathrm{v}}\)应用到源点云P。
对于多个切片,STitch3D采用ICP算法顺序进行成对对齐。 除了ICP算法,STitch3D还集成了一个最近提出的称为PASTE的方法,作为成对对齐任务的可选选择。
在多个切片对齐之后,STitch3D沿着新引入的z轴 来将点的二维位置组装成3维的( assembles the 2D locations of spots along a newly introduced z axis),该轴上的数据描绘了切片对之间的距离(此处有问题,没说明白是三位空间距离是不是物理距离),以构建点的三维空间位置。根据点的三维空间位置,STitch3D为所有切片中的点构建一个全局三维邻域图。默认情况下,如果两个点之间的距离小于一个切片中两个最近点之间距离的1.1倍,则它们在全局三维图中用一条边连接。
基因选择和构建细胞类型特异性基因表达谱矩阵
STitch3D使用T检验找到该细胞类型的前K个标记基因,默认设置K=500。
所有细胞类型的顶级标记基因(前500)然后被拼组在一起,由此获得STitch3D使用的基因完整列表。
对于来自C个细胞类型的选择出的G个基因,STitch3D构建细胞类型特异性基因表达谱矩阵\(V ∈ R^{C×G}\)详细过程见下图:
如果参考单细胞RNA测序数据集包含不同批次,STitch3D为每个批次计算一个细胞类型特异性基因表达谱矩阵,然后取平均以获得整体的细胞类型特异性基因表达谱矩阵V。
STitch3D模型进行三维空间区域检测和三维细胞类型解混
接下来,我们使用STitch3D模型进行三维空间区域检测和三维细胞类型解混。
STitch3D通过整合空间转录组数据集的多个切片和单细胞RNA测序参考数据集的信息进行三维分析。在ST数据中,我们使用\(s = 1,2,...,S\)作为组织切片的索引。对于切片s,我们将其点数表示为\(N_s\),观测到的基因表达计数矩阵为\(Y^s\) = [\(Y^s_{n,g}\)]∈\(R^{N_s×G}\),其中\(n = 1,2,...,N_s\)表示点的索引,\(g = 1,2,...,G\)表示基因的索引。在经注释的单细胞RNA测序参考数据集中,我们将获得的特异性基因表达谱表示为矩阵\(V = [V_c,g]∈R^{C×G}\),其中\(c = 1,2,...,C\)是细胞类型的索引。
在这个矩阵中,行向量\(V_c,∈R^G\),满足\(∑_gV_{c,g} = 1\),表征细胞类型c的平均表达特征。为进行三维分析,STitch3D首先将所有组织切片的点的基因表达信息编码到一个共享的潜在空间,通过神经网络映射现在的3D空转坐标。连接所有切片\(s = 1,2,...,S\)的点索引以引入新的全局点索引\(i = 1,2,...,N\),其中\(N = N_1 + N_2 + ⋯ + N_S\)表示所有切片中的点数。将\(A = [Ai,j]∈R^{N×N}\)表示为指定三维全局图的全局邻接矩阵,其中如果点i和点j之间存在边,即点i和点j是三维邻域点,则\(Ai,j = 1\),否则\(Ai,j = 0\)。设\(Y = [Yi,g]∈R^{N×G}\)是所有切片连接后的基因表达计数矩阵。
点在隐空间的坐标表示通过如下式子:
上式中我们对计数矩阵Y进行归一化和对数转换,\(X = [Xi,g]∈R^{N×G}\)通过基于图的注意力网络\(f_Z(·)\)编码到\(Z ∈ R^{N×p},p\)是隐空间的维度。显然,潜在表示Z对基因表达X和图A中给出的空间邻居的信息进行了编码。
上文中包含了太多的集合和图的信息,这里简单总结一下上文中对齐、引入隐空间的过程。
在得到隐空间信息的\(Z ∈ R^{N×p}\),在这里需要重复一下:潜在表示Z,对基因表达X和图A中给出的空间邻居的信息进行了编码,之后STitch3D基于点的潜在表示 Z 生成细胞类型比例。我们将点i中的细胞类型c的比例表示为\(β(i,c)\)。设\(βi = [(βi,1), (βi,2), ⋯, (βi,C)]^ T ∈ R^C\)其中\(βi,c ≥ 0,∑^C_{c=1} βi,c = 1\)表示点i的细胞类型比例,\(Zi ∈ R^P\)是点i获得的潜在表示信息,其中\(i = 1,2,⋯,N\),我们假设细胞类型比例\(βi\)可以通过神经网络\(fβ(·)\)与潜在表示\(Zi\)相关联,即\(βi = fβ(Zi)\)。值得注意的是,通过基于图的注意力网络\(fZ(·)\)获得的隐编码Z包含了三维全局空间信息。因此,由于\(βi\)是从\(Zi\)生成的,空间位置信息也被并入估计的细胞类型比例\(βi\)中。
总结一下:即通过这个过程,模型可以获得集成了基因表达和位置内容的细胞类型分布预测,为下一步进行细胞类型的空间解混做铺垫。
为了考虑ST数据与scRNA-seq数据之间的技术效应造成的影响以及多个ST切片之间的批效应,STitch3D在其模型中进一步引入了两个效应\(α^s_i\)和\(γ^s_g\),其中\(α^s_i ∈ R\)代表切片和点特异性效应,\(γ^s_g ∈ R\)代表切片和基因特异性效应。结合细胞类型特异性基因表达谱矩阵\(V = [Vc,g]\),细胞类型比例\(βi = [βi,c]\),以及两个效应\(α^s_i\)和\(γ^s_g\),STitch3D能够用以下模型重构ST数据中的观测计数:
其中\(li\)是点i中的观测到的总转录物计数。STitch3D将设置为默认使用泊松分布,并在全文中使用此版本。我们使用切片特异性神经网络从共享潜在空间生成切片和点特异性效应\(α^s_i\):
其中s是切片标签。此处\(f_{\alpha}\)代表一个NN,之所以这样建模\(α^s_i\),是因为假设切片和点特异性效应\(α^s_i\)与基因表达信息相关,后者在\(Zi\)中提炼,也与切片标签\(s\)相关。我们将切片和基因特异性效应\(γ^s_g ∈ R\)建模为可训练参数。通过考虑\(α^s_i\)和\(γ^s_g ∈ R\)中的不想要变异,\(βi\)和\(Zi\)可以更好地捕获有意义的生物学变异。基于此模型,损失函数为:
考虑了一个正则项以增强保留多个切片之间的生物学变异。具体地,我们将正则项设计为:
其中,切片特异性网络\(f_X(·,s)\)用于根据\(Zi\)和切片标签s重构\(Xi\)。这两个网络\(fZ(·)\)和\(fX(·,s)\)共同形成了X和Z之间的自动编码器结构。该正则项鼓励切片特异网络fX预测出符合每个切片真实观测的表达谱。而潜在表示Zi是网络\(fZ(·)\)的输出,不包含切片信息。所以为了预测出切片特异的表达,Zi必须编码所有切片共享的生物学信息。这推动了\(fZ(·)\)提取共享的生物变异,从而起到了增强模型对生物学变异保留在正则项\(R_{AE}\)中,切片特异性网络\(fX(·,s)\)旨在考虑切片之间的批效应,从而鼓励切片共享网络\(fZ(·)\)在Z中提炼生物学信息。STitch3D中的最终目标函数为
其中\(k_{AE}\)是\(R_{AE}\)的系数,固定为0.1。
根据以上设计,STitch3D应用随机梯度下降来更新其模型中的可训练参数。在训练后,STitch3D输出潜在表示\(Zi\)和细胞类型比例\(βi\)。潜在表示用于利用社区检测算法(如Louvain算法和基于高斯混合模型的聚类算法)进行三维空间域识别任务。
结合获得的三维空间位置,这样识别出的簇显现出所有切片中的三维空间区域。同时,细胞类型比例\(βi\)显示不同细胞类型的三维空间分布,为ST研究提供了更高空间分辨率的全面视图。
具体的模型结构
对于网络F(z),其本质上是GAT网络,包含一个图注意力层和一个密集层
针对Fx模型
GAT层的详细信息:
σ(⋅)是激活函数,W是图注意力层中的网络参数,v1和v2是与图注意力机制相关的参数。参数v1和v2用于在训练过程中学习3D邻域spot之间的边权重\(ai, j\)
class DeconvNet(nn.Module):
def __init__(self,
hidden_dims, # dimensionality of hidden layers
n_celltypes, # number of cell types
n_slices, # number of slices
n_heads, # number of attention heads
slice_emb_dim, # dimensionality of slice id embedding
coef_fe,
):
super().__init__()
# define layers
# encoder layers
self.encoder_layer1 = GATMultiHead(hidden_dims[0], hidden_dims[1], n_heads=n_heads, concat_heads=True)
self.encoder_layer2 = DenseLayer(hidden_dims[1], hidden_dims[2])
# decoder layers
self.decoder_layer1 = GATMultiHead(hidden_dims[2] + slice_emb_dim, hidden_dims[1], n_heads=n_heads, concat_heads=True)
self.decoder_layer2 = DenseLayer(hidden_dims[1], hidden_dims[0])
# deconvolution layers
self.deconv_alpha_layer = DenseLayer(hidden_dims[2] + slice_emb_dim, 1, zero_init=True)
self.deconv_beta_layer = DenseLayer(hidden_dims[2], n_celltypes, zero_init=True)
self.gamma = nn.Parameter(torch.Tensor(n_slices, hidden_dims[0]).zero_())
self.slice_emb = nn.Embedding(n_slices, slice_emb_dim)
self.coef_fe = coef_fe
def forward(self,
adj_matrix, # adjacency matrix including self-connections
node_feats, # input node features
count_matrix, # gene expression counts
library_size, # library size (based on Y)
slice_label, # slice label
basis, # basis matrix
):
# encoder
Z = self.encoder(adj_matrix, node_feats)
# deconvolutioner
slice_label_emb = self.slice_emb(slice_label)
beta, alpha = self.deconvolutioner(Z, slice_label_emb)
# decoder
node_feats_recon = self.decoder(adj_matrix, Z, slice_label_emb)
# reconstruction loss of node features
self.features_loss = torch.mean(torch.sqrt(torch.sum(torch.pow(node_feats-node_feats_recon, 2), axis=1)))
# deconvolution loss
log_lam = torch.log(torch.matmul(beta, basis) + 1e-6) + alpha + self.gamma[slice_label]
lam = torch.exp(log_lam)
self.decon_loss = - torch.mean(torch.sum(count_matrix *
(torch.log(library_size + 1e-6) + log_lam) - library_size * lam, axis=1))
# Total loss
loss = self.decon_loss + self.coef_fe * self.features_loss
return loss
def deconvolutioner(self, Z, slice_label_emb):
beta = self.deconv_beta_layer(F.elu(Z))
beta = F.softmax(beta, dim=1)
H = F.elu(torch.cat((Z, slice_label_emb), axis=1))
alpha = self.deconv_alpha_layer(H)
return beta, alpha
完整的数据流向梳理:
基因表达水平的去噪和填充
STitch3D的细胞类型解卷积结果还能够去噪低质量基因的表达,并填充ST数据集中未测量到的的基因表达。对于需要去噪或填充的感兴趣基因,STitch3D首先基于单细胞RNA测序参考,计算出一个细胞类型特定的基因表达概要向量\(V′ = [V′ c]∈ℝ^{C×1}\),方式与高度可变基因的细胞类型特定基因表达概要矩阵V获取相同。然后,使用STitch3D估计的每个spot中的细胞类型比例$βi \(,感兴趣基因的去噪或填充表达水平由\)βi,c ≥ 0,∑^C_{c=1} βi,c = 1$给出。
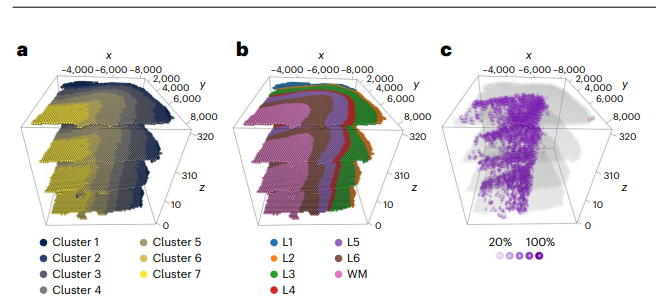
人类背外侧前额叶皮质(DLPFC)数据集评估空间检测性能
标注了六个层次(L1-L6)和白质(WM),当应用于每个单独的切片时,STitch3D稳定地恢复了层次结构
在多切片分析中应用时,STitch3D产生了一致的结果:

并使用调整后的Rand指数(ARI)来评估准确性。与单切片结果相比,STitch3D的多切片结果获得了更高的ARI分数,STitch3D的3D空间区域识别优势在于它在共享的潜在空间中有效地整合了切片(图2d)
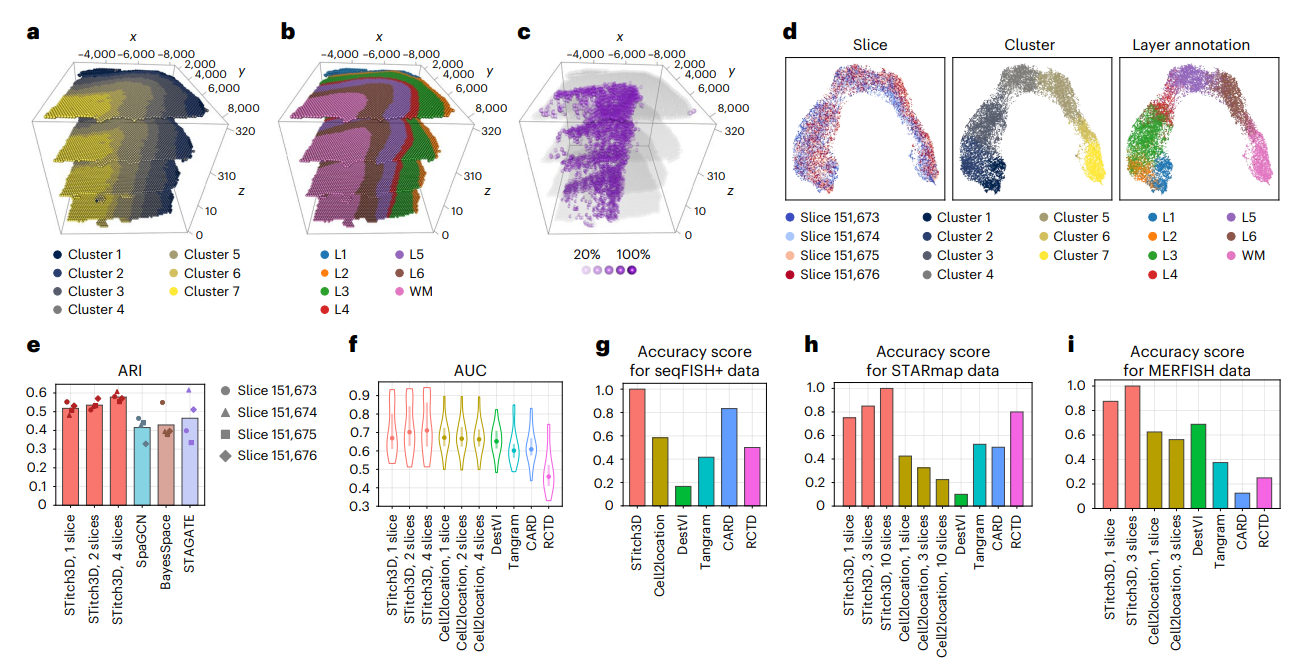
图2D详细部分:

STitch3D的细胞类型解卷积性能
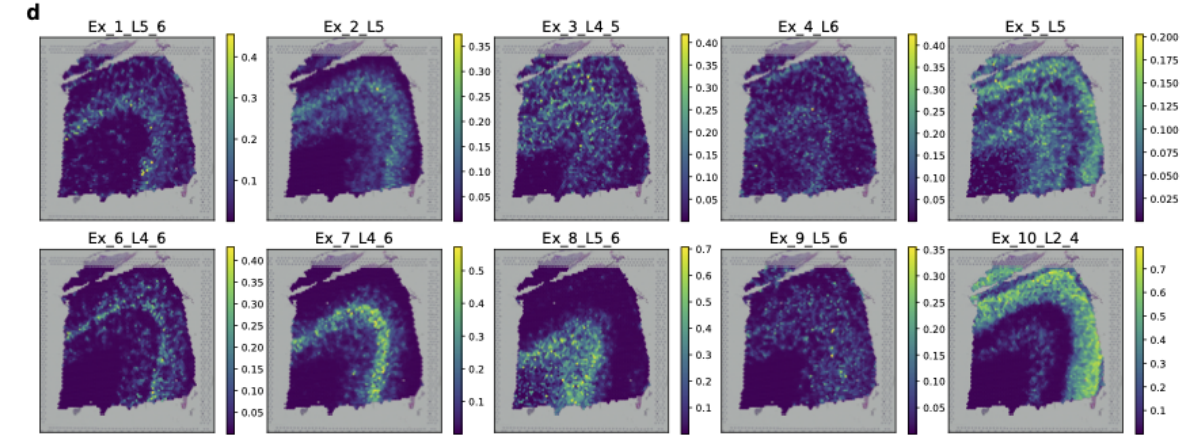
参考另一篇文章的针对十个兴奋性神经元亚型的细胞类型标注,每个亚型的层特异性是基于层标记基因确定的,基于ROC分析来评估细胞类型解卷积的可靠性,其中使用估计的神经元亚型比例来恢复层特异性。
ROC下面积(AUC)的较高值表示更可靠的性能:
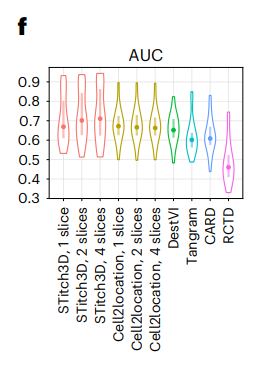
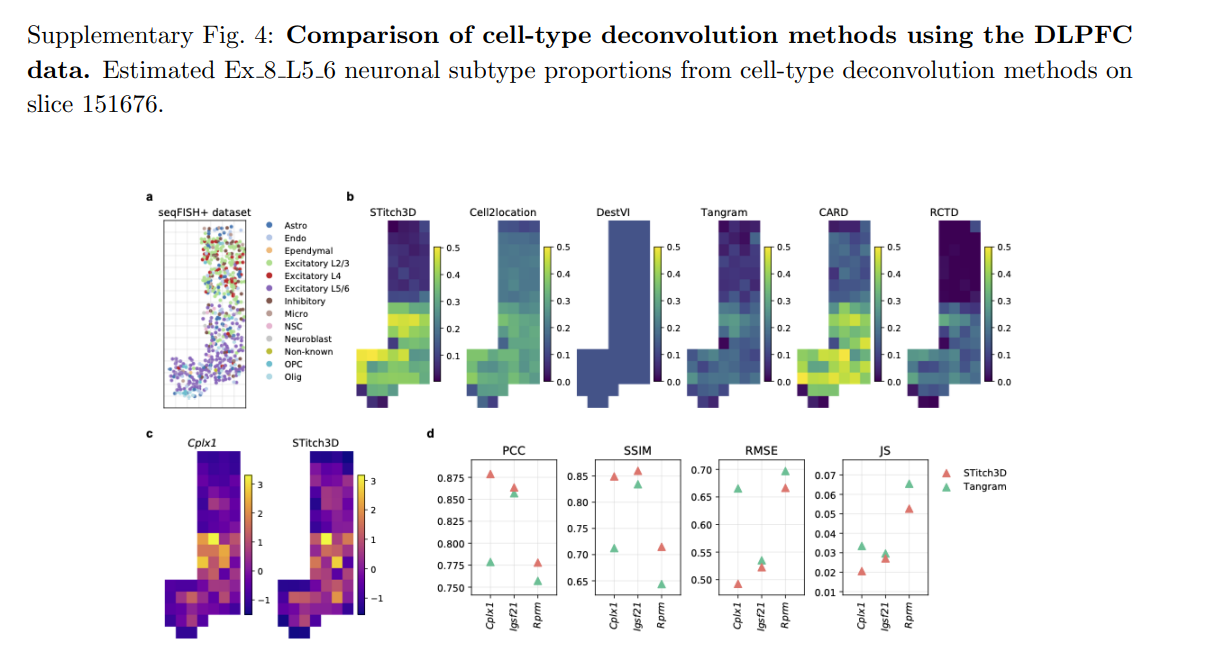
以Ex_8_L5_6这一命名的神经亚型为例,它具有层次5-6标记基因的高表达,STitch3D在层次5-6中明显丰富:
附图3:
附图4:
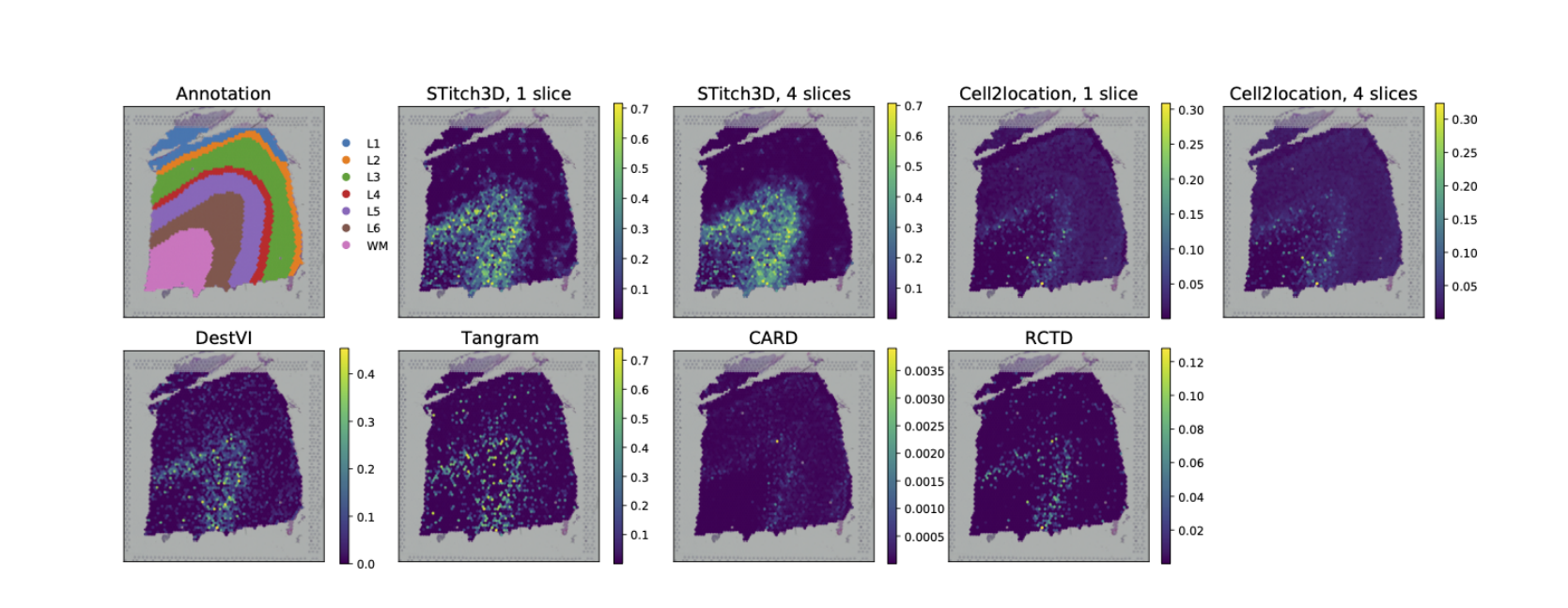
与定量评估一致,STitch3D的多切片结果中,层次1-4中Ex_8_L5_6比例较低且较少噪音
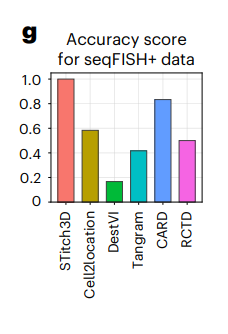
同时还进行了模拟测试,由于在模拟数据集中已知真实的细胞类型比例,使用总体准确度评分进行定量测量:
使用小鼠皮层seqFISH+数据集,将细胞划分为不同的点,创建了一个模拟的ST切片,使用相应的单细胞参考【26】,STitch3D在所有方法中获得了最高的总体准确度分数:
成熟小鼠脑重建
STitch3D可以准确重建复杂的3D成年小鼠脑。我们使用了横断面的35个切片,并包含了59种细胞类型的细胞类型(参考【16】),先计算了配准的准确度:
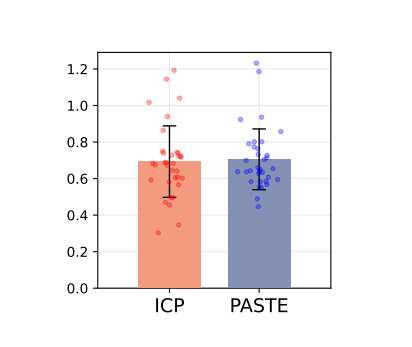
对于切片A中的每个spot,我们计算了所选spot与切片B中具有与所选spot相同区域标签的spot之间的最小距离。然后,通过对两个切片中所有spot的这些最小距离求平均得到得分。较小的值表示性能更好。
将大脑划分为有组织的3D领域:
被标记为簇1、2和5的三个层次结构领域形成了同一皮层区域
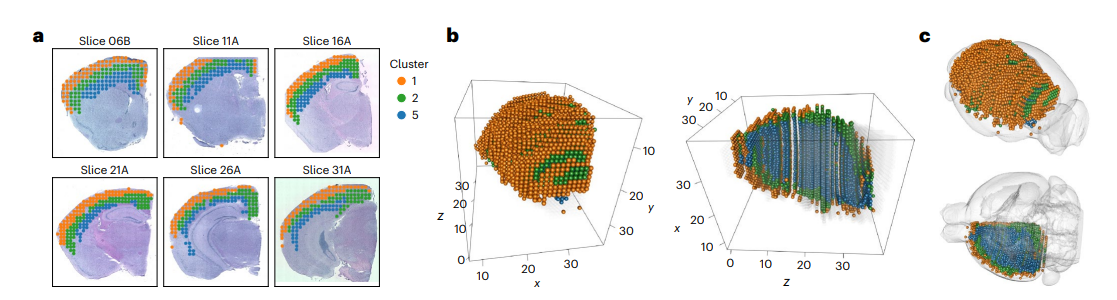
在这些簇内的伪时间分析显示出与所有切片上的皮层发生相一致的模式

簇3和9对应于海马和丘脑区域,沿AP轴发生变化,表明STitch3D能够在切片之间保留生物变异性:
附图17d,e和18
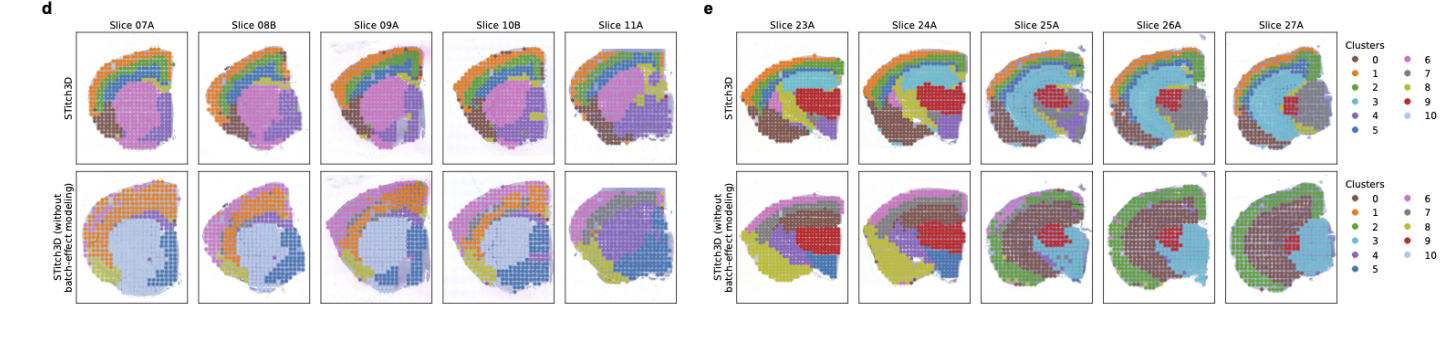
通过这个示例,还验证了STitch3D批次效应建模的有效性。当我们从STitch3D中删除它时,切片的整合性较差,导致切片之间的空间领域检测不一致

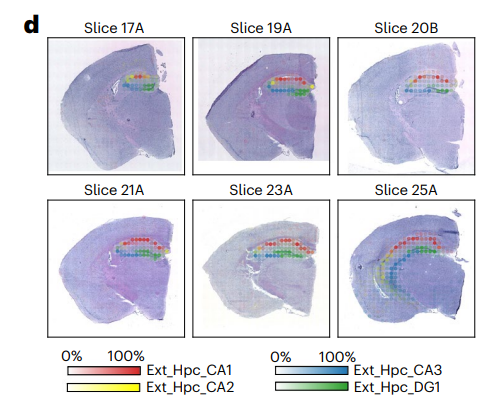
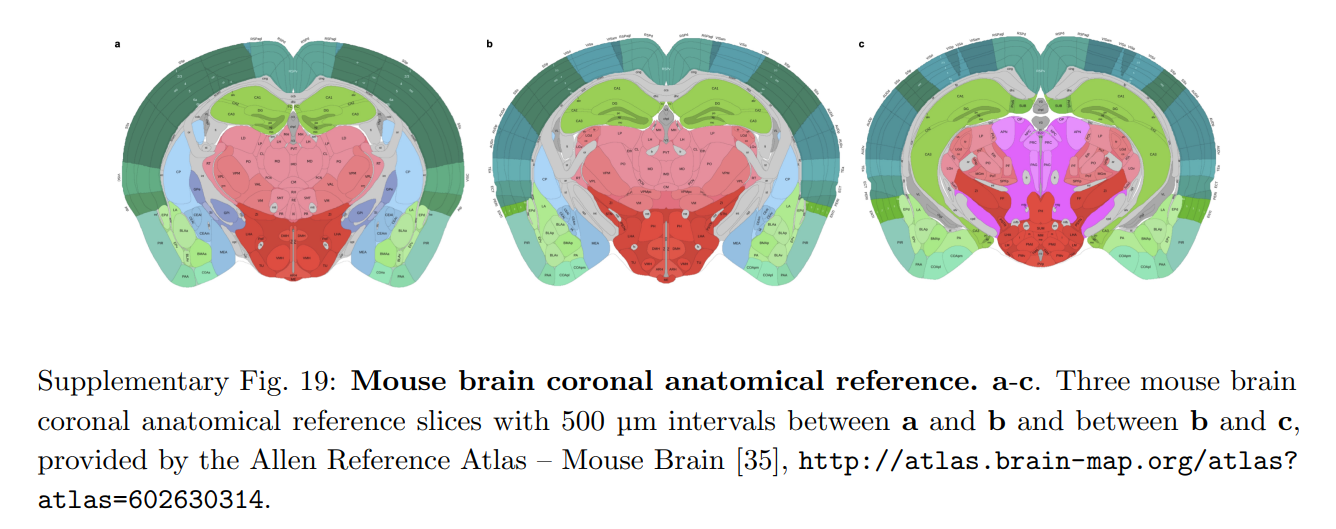
利用来自参考文献的细胞类型的精细标志,STitch3D展示了3D细胞类型分布。例如,它准确重建了海马区角结节(CA)和齿状回(DG)中的四种海马神经元类型的分布(图3e,f)。
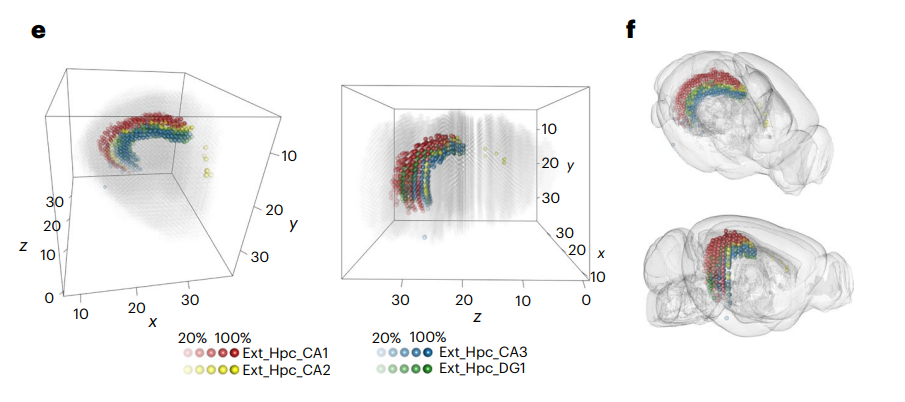
这些分布正确匹配了Allen参考图谱 - 小鼠大脑29中标注的CA1、CA2、CA3和DG海马区域【参考29】(图3d和附图19)


 浙公网安备 33010602011771号
浙公网安备 33010602011771号