PAM50怎么计算
看来还得和临床结合才知道为什么提出问题
问题的背景
为什么提出PAM50分型,因为乳腺癌病人的异质性特别大,所以需要将这些病人进行一个分型,以便更好的治疗。
发展的历史
最初是根据免疫组化的三个指标进行分型的
- HR+ HER2-
- HR+ HER2+
- HR- HER2+
- TNBC
随着RNA-seq的发展,得到的信息更多,于是提出了PAM50分型,也就是根据这50个分子去确定乳腺癌的表型。50个分子怎么确定的呢?又分为哪几类呢?
50个分子的确定 :根据层次聚类的结果将病人分为5类,选择每一类里面top10的基因,一共50个基因。
划分的五类[1]:
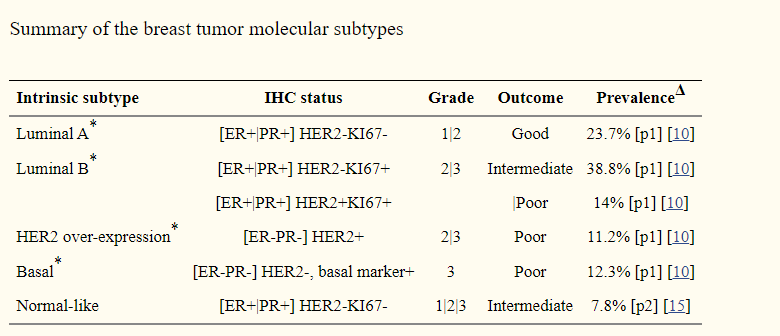
这两个分型之间存在什么关系呢?下面是一篇文章计算出来的结果[2]
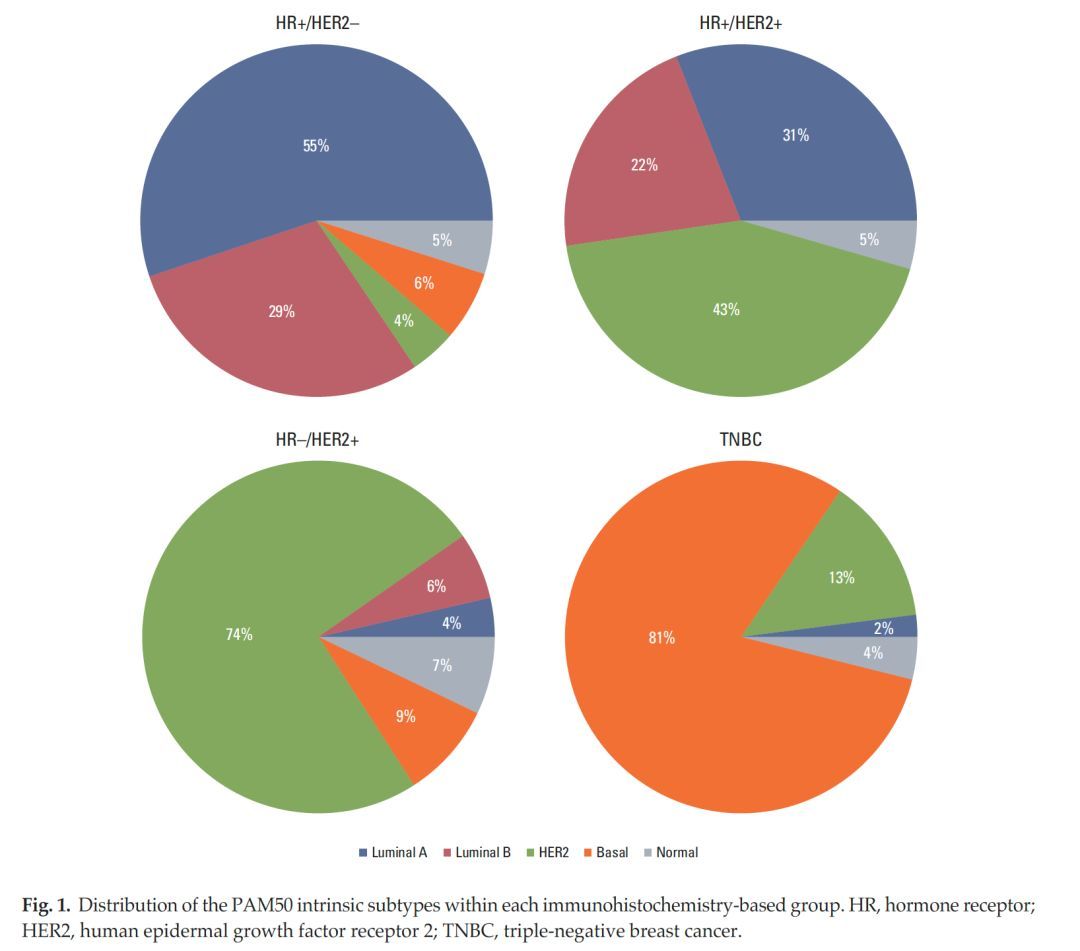
怎么计算
不讲原理只放代码感觉有点耍流氓,那还是先讲原理把
首先根据找到的病人层次聚类,找到5组病人50个基因的中心(centroids),这个不用咱们去训练,已经有公认的了,接着很简单,来了一个新的病人,直接去看和哪个中心比较近就定为哪一类的病人。
简单粗暴但是好用。至于这个距离怎么计算,欧氏距离,pearson,spearman系数均可以。
现在还有些论文在研究RNA-seq和digital multiplexed gene expression technologies计算出来的结果是否一致,发现大部分差不多[3]。
代码
这里直接放上作者的代码吧,还找了挺久的,里面最关键的是pam50_centroids.txt这个文件,记得把自己的基因表达数据scale一下就可以了。
参考文献
- Am J Cancer Res. 2015; 5(10): 2929–2943.
- Cancer Res Treat. 2019;51(2):737-747
- A. C. Picornell et al/BMC Genomics
- https://zhuanlan.zhihu.com/p/137070362
- J Clin Oncol. 2009 Mar 10; 27(8): 1160–1167.

 浙公网安备 33010602011771号
浙公网安备 33010602011771号