1. KEGG富集分析简介
1. KEGG富集分析的作用
在生物信息学分析过程中,KEGG通路富集分析常常应用于差异表达基因的功能注释,了解差异表达基因的相关功能与作用通路。
2. KEGG气泡图的解读

例如上图显示的是KEGG点状图,横轴GeneRatio表示对应pathway差异基因数/能够对应到pathway数据库中的差异基因数,即代表该pathway下的差异基因个数占差异基因总数的比例,纵轴表示富集到的pathway的描述信息。
qvalue代表富集分析得到的qvalue,Count代表富集基因数目。showCategory指定展示的pathway的个数,比如展示显著富集的top10个,即p.adjust(矫正后的pvalue)最小的10个。不同的颜色代表不同adjusted p-value,从蓝色到红色,表示adjusted p-value从大到小,富集程度越来越显著。
3. KEGG柱形图的解读
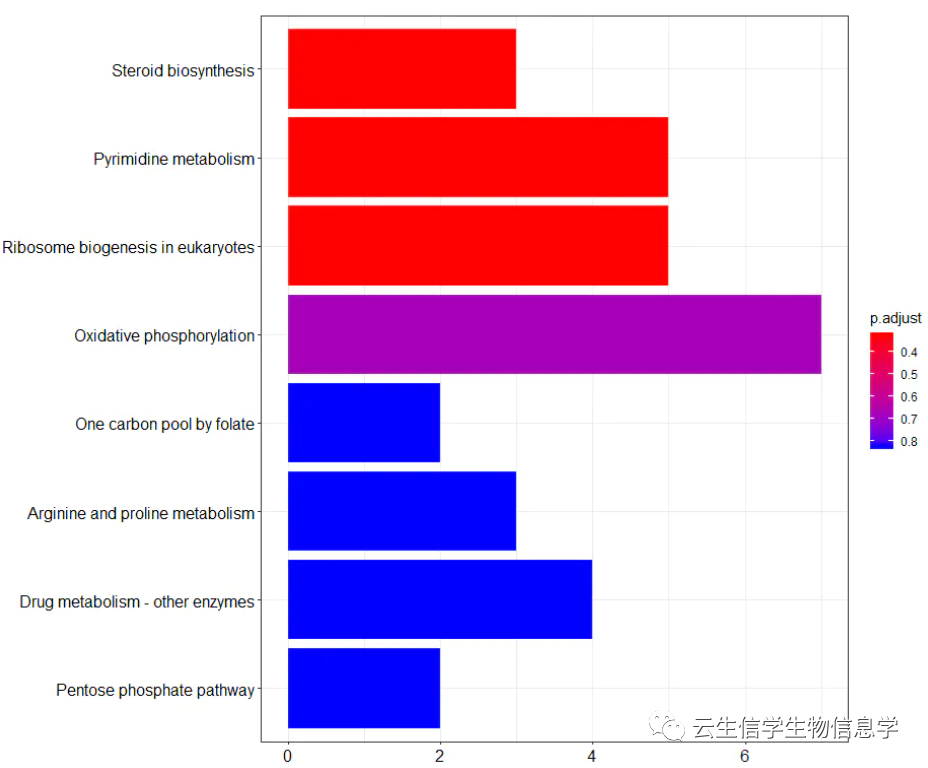
上图中的横轴为该pathway的差异基因个数,纵轴为富集到的pathway的描述信息。
同样showCategory指定展示的pathway的个数,比如可以展示显著富集的top10个,即p.adjust最小的10个。条形的颜色对应p.adjust值,从小到大,对应蓝色到红色。
4. KEGG通路图的解读

KEGG官网样式的通路图是一幅线框图,即由点和线构成的,其中红色基因上调,绿色基因下调。点和线构成的分子间的关系类型,比如蛋白-蛋白互作包含大量磷酸化(+p)和去磷酸化(-p)的过程等等。
2. 条形图代码

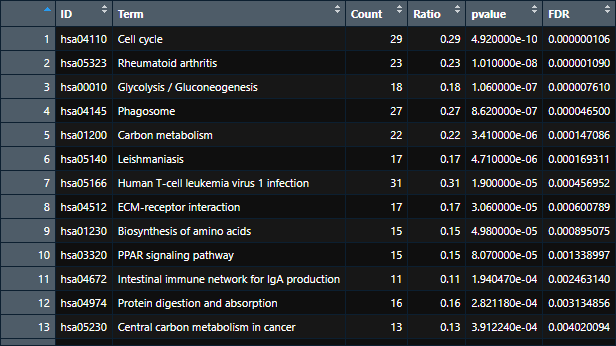
该条形图展示的是富集在每个Term的基因数目。Term可以是GO或者通路名称等等。FDR是矫正后的pֵ值。
输入数据
该输入数据的每一行显示的是一个Term(GO或通路)中富集到的基因数目、比例、P值。

ID Term Count Ratio pvalue FDR hsa04110 Cell cycle 29 0.29 4.92E-10 1.06E-07 hsa05323 Rheumatoid arthritis 23 0.23 1.01E-08 1.09E-06 hsa00010 Glycolysis / Gluconeogenesis 18 0.18 1.06E-07 7.61E-06 hsa04145 Phagosome 27 0.27 8.62E-07 4.65E-05 hsa01200 Carbon metabolism 22 0.22 3.41E-06 0.000147086 hsa05140 Leishmaniasis 17 0.17 4.71E-06 0.000169311 hsa05166 Human T-cell leukemia virus 1 infection 31 0.31 1.90E-05 0.000456952 hsa04512 ECM-receptor interaction 17 0.17 3.06E-05 0.000600789 hsa01230 Biosynthesis of amino acids 15 0.15 4.98E-05 0.000895075 hsa03320 PPAR signaling pathway 15 0.15 8.07E-05 0.001338997 hsa04672 Intestinal immune network for IgA production 11 0.11 0.000194047 0.00246314 hsa04974 Protein digestion and absorption 16 0.16 0.000282118 0.003134856 hsa05230 Central carbon metabolism in cancer 13 0.13 0.000391224 0.004020094 hsa05152 Tuberculosis 24 0.24 0.000413079 0.004051728 hsa04066 HIF-1 signaling pathway 17 0.17 0.000470448 0.004413814 hsa00052 Galactose metabolism 8 0.08 0.000536204 0.004821133 hsa04973 Carbohydrate digestion and absorption 10 0.1 0.0005968 0.005151327 hsa04151 PI3K-Akt signaling pathway 38 0.38 0.000923352 0.007663446 hsa04659 Th17 cell differentiation 16 0.16 0.001093815 0.008741993 hsa05205 Proteoglycans in cancer 25 0.25 0.001196583 0.009221785 hsa00250 Alanine, aspartate and glutamate metabolism 8 0.08 0.001553291 0.011172793 hsa04514 Cell adhesion molecules 19 0.19 0.002505451 0.015447142 hsa04922 Glucagon signaling pathway 15 0.15 0.002704018 0.015920412 hsa04668 TNF signaling pathway 15 0.15 0.004613543 0.023703667 hsa04918 Thyroid hormone synthesis 11 0.11 0.007325236 0.031375446 hsa05321 Inflammatory bowel disease 10 0.1 0.007401499 0.031375446 hsa04972 Pancreatic secretion 13 0.13 0.012082574 0.041385593 hsa04910 Insulin signaling pathway 16 0.16 0.012928402 0.043510049 hsa05332 Graft-versus-host disease 7 0.07 0.015483236 0.047730277 hsa04115 p53 signaling pathway 10 0.1 0.016357389 0.049714821
代码
# 需要三列信息
# Term:GO或者通路名称
# Count:富集在每个term的基因数目
# FDR:矫正后的p
library(ggplot2) # 引用包
setwd("") # 设置工作目录
rt <- read.table("input.txt", header = T, sep = "\t", check.names = F) # 读取文件
# 按FDR排序
labels <- rt[order(rt$FDR, decreasing = T), "Term"] # 将Term按FDR排序
rt$Term = factor(rt$Term, levels = labels) # 将Term设为因子
# 绘制
p <- ggplot(data = rt) +
geom_bar(aes(x = Term, y = Count, fill = FDR), stat = "identity") + # 用原始数据绘制初始条形图
coord_flip() + # 翻转坐标系
scale_fill_gradient(low = "red", high = "blue") + # 添加双色渐变色阶
xlab("Term") + # 设置x轴标签
ylab("Gene count") + # 设置y轴标签
theme(axis.text.x = element_text(color = "black", size = 10), # 设置x轴文本颜色及字体大小
axis.text.y = element_text(color = "black", size = 10)) + # 设置y轴文本颜色及字体大小
scale_y_continuous(expand = c(0, 0)) + # y轴(Gene count)连续数据位置标度
scale_x_discrete(expand = c(0,0)) + # x轴(Term)离散数据的位置尺度
theme_bw() # 设置全局主题
ggsave("barplot.pdf", width = 7, height = 5) #保存图片



