16S测序和宏基因组测序的区别
https://mp.weixin.qq.com/s/xsL9GuLs7b3nRF8VeRtinQ
http://www.sohu.com/a/146620511_769248
宏基因组测序(MetagenomicsSequencing)是对环境样品中全部微生物的总DNA(也称宏基因组:Metagenomic)进行高通量测序,主要研究微生物种群结构、基因功能活性、微生物之间的相互协作关系以及微生物与环境之间的关系。宏基因组测序研究摆脱了微生物分离纯培养的限制,扩展了微生物资源的利用空间,为环境微生物群落的研究提供了有效工具。
微生物测序研究常用手段包括16S等扩增子测序和宏基因组测序,这两者技术手段的主要区别如下:
1测序原理不同
16S rDNA基因存在于所有细菌的基因组中,具有高度的保守性。该序列包含9个高变区和10个保守区(如下图),通过对某一段高变区序列(V4区或V3-V4区)进行PCR扩增后进行测序,得到1500bp左右的序列。宏基因组测序则是将微生物基因组DNA随机打断成500bp的小片段,然后在片段两端加入通用引物进行PCR扩增测序,再通过组装的方式,将小片段拼接成较长的序列。
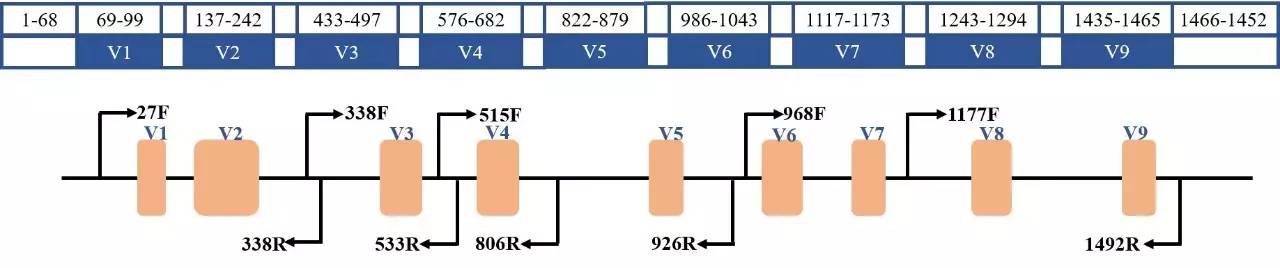
图 细菌16S区域
2研究目的不同
16S测序主要研究群落的物种组成、物种间的进化关系以及群落的多样性。宏基因组测序在16S测序分析的基础上还可以进行基因和功能层面的深入研究(GO、Pathway等)。
3物种鉴定深度不同
16S测序得到的序列很多注释不到种水平,而宏基因组测序则能鉴定微生物到种水平甚至菌株水平。对于16S测序而言,任何一个高变区或几个高变区,尽管具有很高的特异性,但是某些物种(尤其是分类水平较低的种水平)在这些高变区可能非常相近,能够区分它们的特异性片段可能不在扩增区域内。宏基因组测序通过对微生物基因组随机打断,并通过组装将小片段拼接成较长的序列。因此,在物种鉴定过程中,宏基因组测序具有较高的优势。
Tips:通常情况下,在微生态研究中,建议同时结合宏基因组测序和16S测序两种技术手段,可以更高效、更准确地研究微生物群落组成结构、多样性以及功能情况。




【推荐】国内首个AI IDE,深度理解中文开发场景,立即下载体验Trae
【推荐】编程新体验,更懂你的AI,立即体验豆包MarsCode编程助手
【推荐】抖音旗下AI助手豆包,你的智能百科全书,全免费不限次数
【推荐】轻量又高性能的 SSH 工具 IShell:AI 加持,快人一步