(un)Weighted UniFrac 分析
(un)Weighted UniFrac 分析
UniFrac分析利用各样品序列间的进化信息来比较环境样品在特定的进化谱系中是否有显著的微生物群落差异。 UniFrac 可用于beta 多样性的评估分析,即对样品两两之间进行比较分析,得到样品间的unifrac距离矩阵。其计算方法为:首先利用来自不同环境样品的OTU 代表序列构建一个进化树,Unifrac 度量标准根据构建的进化树枝的长度计量两个不同环境样品之间的差异,差异通过0-1 距离值表示,进化树上最早分化的树枝之间的距离为1,即差异最大,来自相同环境的样品在进化树中会较大几率集中在相同的节点下,即它们之间的树枝长度较短,相似性高。若两个群落完全相同,那么它们没有各自独立的进化过程,UniFrac值为0;若两个群落在进化树中完全分开,即它们是完全独立的两个进化过程,那么UniFrac值为1。
从UniFrac的定义中,可以看出它只考虑序列是否在群落中出现,而不考虑序列的丰度。若两个群落包含的物种完全相同,那么不管每个物种的丰度是否有差别或者差别的大小,UniFrac值为0。weighted unifrac方法,就是在UniFrac的基础上,将序列的丰度纳入考虑,它能够区分物种丰度的差别。在计算中, weighted unifrac按照每条枝指向的叶节点中来自两个群落的比例,给每条枝加权重。因此unweighted unifrac 可以检测样品间变化的存在,而weighted unifrac 可以更进一步定量的检测样品间不同谱系上发生的变异。
软件及算法:使用FastTree(version 2.1.3 http://www.microbesonline.org/fasttree/)根据最大似然法( approximately-maximum-likelihood phylogenetic trees ) 构建进化树,然后利用Fastunifrac[2] (http://unifrac.colorado.edu/)分析得到样品间距离矩阵。 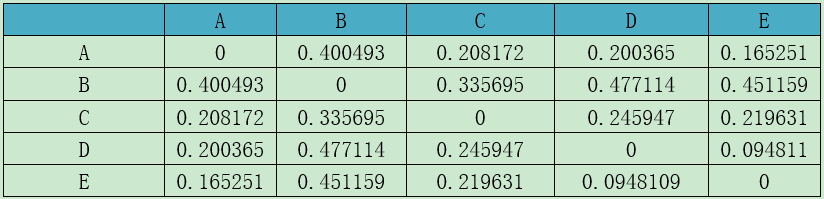
Table(un)weighted unifrac distance matrix
注:第一行和第一列均为样品。
参考文献:
[1] Tanya Yatsunenko, Federico, et al. Human gut microbiome viewed across age and geography. Nature486, 222–227 (14 June 2012) doi:10.1038.nature11053.
[2] Micah Hamady, Catherine Lozupone and Rob Knight. Fast UniFrac:facilitatinghigh-throughput phylogenetic analyses of microbial communities including analysis of pyrosequencing and PhyloChip data.The ISME Journal (2010) 4, 17–27; doi:10.1038/ismej.2009.97
基于UniFrac 的Pcoa 分析
Unifrac 分析得到的距离矩阵可用于多种分析方法,可通过多变量统计学方法PCoA 分析,直观显示不同环境样品中微生物进化上的相似性及差异性。 PCoA(principal co-ordinates analysis)是一种研究数据相似性或差异性的可视化方法,通过一系列的特征值和特征向量进行排序后,选择主要排在前几位的特征值,PCoA 可以找到距离矩阵中最主要的坐标,结果是数据矩阵的一个旋转,它没有改变样品点之间的相互位置关系,只是改变了坐标系统。通过PCoA 可以观察个体或群体间的差异。
分析软件:R 语言PCoA 分析和作PCoA 图。
unifrac.pcoa.tiff :样品PCoA 分析图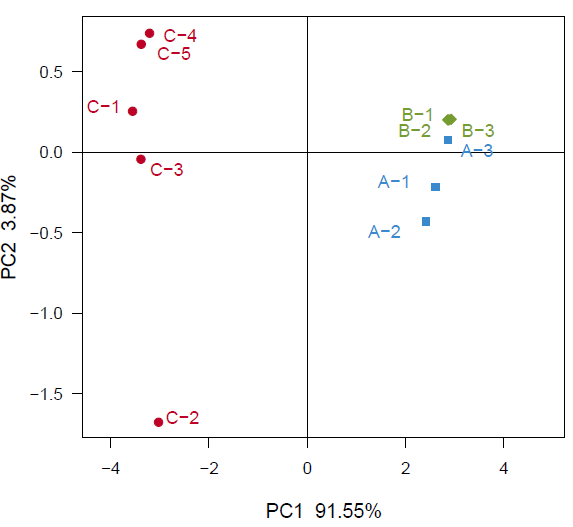
Fig (un)weighted unifrac PCoA analysis
参考文献:
Xiao-Tao Jiang ,Xin Peng, et al.Illumina Sequencing of 16S rRNA Tag Revealed Spatial Variations of Bacterial Communities in a Mangrove Wetland. Microb Ecol (2013) 66:96–104.DOI10.1007/s00248-013-0238-8.
注:PC1 和PC2 是两个主坐标成分,PC1 表示尽可能最大解释数据变化的主坐标成分,PC2 为解释余下的变化度中占比例最大的主坐标成分,PC3 等依次类推。
基于UniFrac 的多样品相似度树分析
Unifrac 分析得到的距离矩阵可用于多种分析方法,通过层次聚类(Hierarchical cluatering)[1]中的非加权组平均法UPGMA 构建进化树等图形可视化处理,可以直观显示不同环境样品中微生物进化上的相似性及差异性。
UPGMA(Unweighted pair group method with arithmetic mean)假设在进化过程中所有核苷酸/氨基酸都有相同的变异率,即存在着一个分子钟。通过树枝的距离和聚类的远近可以观察样品间的进化距离。
分析软件: R 语言vegan 包UPGMA 分析和作进化树。
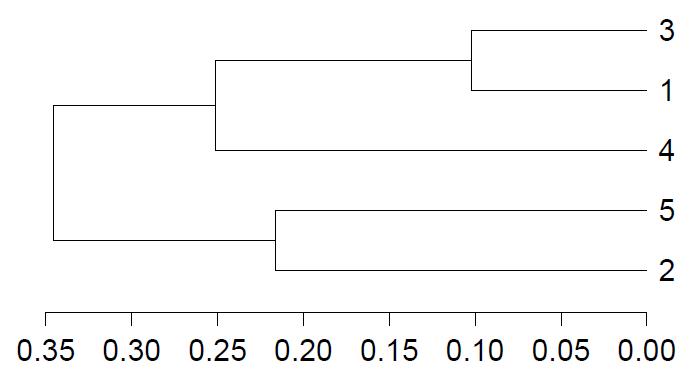 (un) weighted unifrac tree analysis
(un) weighted unifrac tree analysis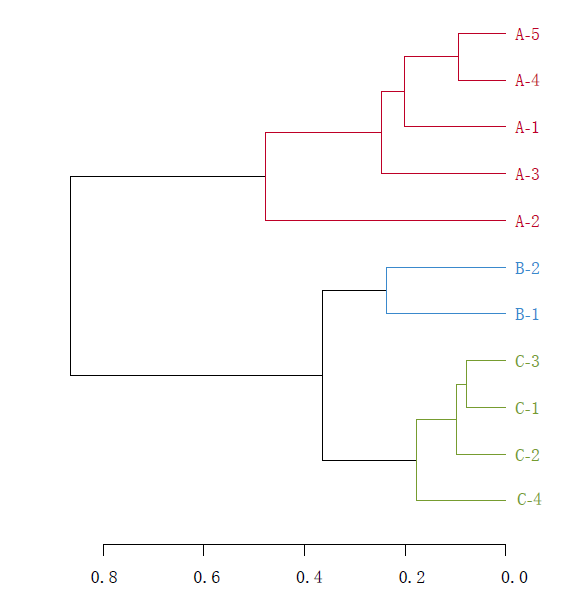
(un) weighted unifrac tree analysis
注:树枝颜色为预先定义的不同分组标注。
参考文献:
[1] Magali Noval Rivas, PhD, Oliver T. Burton, et al. A microbita signature associated with experimental food allergy promotes allergic senitization and anaphylaxis. The Journal of Allergy and Clinical Immunology.Volume 131, Issue 1 , Pages 201-212, January 2013.


 浙公网安备 33010602011771号
浙公网安备 33010602011771号