GET代码学习
这段代码展示了如何将数据从 DataFrame 形式转化为 “blocks” 的过程。具体来说,它通过处理 DataFrame 的每一行(每一行代表一个原子),将它们按残基(residue)分组,每个组被称为一个 "block"。以下是对代码的详细分析,解释如何将数据转化为 "block"。
主要步骤:
-
循环遍历每个原子(DataFrame 的每一行):
df.itertuples()用于按行遍历 DataFrame,每一行都代表一个原子。- 从每一行中提取原子所属的残基 ID、原子名称、元素类型及其空间坐标等信息。
-
按残基(residue)分组:
res_id是一个字符串,它将残基 ID 和插入代码(insertion code)拼接起来形成独特的标识符。如果没有插入代码,只使用残基 ID。- 如果
res_id发生变化(即遇到新的残基),意味着当前残基的原子分组已经结束,形成了一个完整的 "block"。 - 新的 "block" 使用
Block类来表示,包含残基的符号(res_symbol)和对应的原子列表(units)。
-
跳过氢原子:
- 如果元素为氢(H),则跳过这一行(
continue),因为氢原子在许多情况下被忽略。
- 如果元素为氢(H),则跳过这一行(
-
创建
Atom实例:- 每个原子被创建为一个
Atom对象,包含原子的名称、三维坐标(x, y, z),以及元素类型。 - 这些
Atom对象会被添加到当前残基的 "block" 中。
- 每个原子被创建为一个
-
生成
Block对象:- 当遍历到新的残基时,前一个残基的 "block" 完成,创建并存储到
blocks列表中。 - 遍历结束后,最后一个残基的 "block" 被加入到
blocks中。
- 当遍历到新的残基时,前一个残基的 "block" 完成,创建并存储到
代码分析:
1. df_to_blocks 函数:
这是将 DataFrame 中的原子数据分块(block)的核心函数。
def df_to_blocks(df, key_residue='residue', key_insertion_code='insertion_code', key_resname='resname',
key_atom_name='atom_name', key_element='element', key_x='x', key_y='y', key_z='z') -> List[Block]:
last_res_id, last_res_symbol = None, None # 保存上一个残基的ID和符号
blocks, units = [], [] # blocks存放每个block,units存放每个block中的原子
for row in df.itertuples(): # 遍历每一行(原子)
residue = getattr(row, key_residue) # 提取残基ID
if key_insertion_code is None:
res_id = str(residue)
else:
insert_code = getattr(row, key_insertion_code) # 提取插入代码
res_id = f'{residue}{insert_code}'.rstrip() # 拼接残基ID和插入代码
# 如果残基ID发生变化,意味着当前block结束
if res_id != last_res_id:
block = Block(last_res_symbol, units) # 创建一个新的Block对象
blocks.append(block) # 将Block添加到blocks列表
units = [] # 清空units以开始新的Block
last_res_id = res_id # 更新为新的残基ID
last_res_symbol = VOCAB.abrv_to_symbol(getattr(row, key_resname)) # 通过残基名称获取符号
# 处理当前原子
atom = getattr(row, key_atom_name) # 获取原子名称
element = getattr(row, key_element) # 获取元素类型
if element == 'H': # 跳过氢原子
continue
units.append(Atom(atom, [getattr(row, axis) for axis in [key_x, key_y, key_z]], element)) # 添加原子
blocks = blocks[1:] # 忽略第一个空block
blocks.append(Block(last_res_symbol, units)) # 添加最后一个Block
return blocks
详细步骤解释:
-
初始化:
last_res_id和last_res_symbol用于追踪前一个残基的信息,blocks用于存放最终的 Block 列表,units用于暂存当前 Block 的原子。
-
按行遍历 DataFrame:
df.itertuples()会将 DataFrame 中每一行作为一个元组返回,通过getattr()可以访问每个属性(例如residue、atom_name等)。
-
检查残基 ID 变化:
- 当
res_id发生变化时,说明已经处理完当前残基,需要创建一个新的 Block。 - 使用
Block(last_res_symbol, units)创建一个 Block 对象,units则是该 Block 中的所有原子。
- 当
-
处理原子信息:
- 每一行代表一个原子,创建
Atom对象时,会提取该原子的名称、三维坐标和元素信息。
- 每一行代表一个原子,创建
-
返回结果:
- 最后返回
blocks列表,包含所有的 Block 对象。
- 最后返回
总结:
- 代码中的
df_to_blocks函数从 DataFrame 中提取原子,并将它们按残基分组,创建了一个个 Block。每个 Block 包含一个残基的信息(符号和原子列表)。 - 如果 DataFrame 中的数据已经按照这种结构组织好,那么这段代码可以将数据高效地转换为模型可以处理的块状数据结构。
这个分组和转换过程的核心是识别残基 ID 的变化,然后将每个残基的原子聚合成一个 Block。
按残基分组是什么意思。也就是说一个block里面会有多个相同的残基么?
“按残基分组”是指将同一个残基(residue)中所有的原子归类到一个 block 中。在蛋白质结构中,残基通常指氨基酸,而每个氨基酸残基包含若干原子,例如碳、氢、氧、氮等。
一个 block 代表的是一个残基及其包含的所有原子。因此,一个 block 里面不会有多个相同的残基,而是每个残基单独作为一个 block,包含该残基的所有原子。每个残基只对应一个 block,多个相同的残基(例如多个“甘氨酸”残基)会分别出现在不同的 block 中。
详细解释:
- 在蛋白质结构中,残基通常指的是氨基酸。每个氨基酸残基包含多个原子(例如,氮原子、碳原子、氧原子等),这些原子组成了氨基酸的骨架和侧链。
- 当你“按残基分组”时,代码会遍历每个原子,并根据原子所属的残基(通过残基 ID
res_id)将同一残基的所有原子放在一起。 - 一个 block 就是代表了一个残基的原子集群。举例来说,一个“甘氨酸”残基,它包含 C、N、O 等原子,这些原子会被归为同一个 block。
举例:
假设我们有一个蛋白质片段,包含两个氨基酸残基:甘氨酸(Gly)和丙氨酸(Ala)。每个氨基酸残基包含若干原子:
-
甘氨酸残基(Gly):
- 原子:N、C、O、H
- 这些原子被分组到同一个 block 中,表示为 Block 1。
-
丙氨酸残基(Ala):
- 原子:N、C、O、H、Cβ(侧链上的原子)
- 这些原子被分组到另一个 block 中,表示为 Block 2。
代码的具体过程:
- 当代码遍历 DataFrame 时,它会检查每一行原子所属的残基 ID(
res_id)。 - 当它遇到一个新的残基 ID 时,意味着前一个残基的所有原子已经处理完毕,生成一个 block。
- 一个 block 包含当前残基的所有原子。然后,开始处理下一个残基,创建新的 block。
总结:
- 一个 block 只包含一个残基的所有原子。
- 不同的残基会分别生成不同的 block。
- 多个相同的残基(例如多个“甘氨酸”残基)会出现在不同的 block 中,而不是合并在一起。
希望这能帮助你理解按残基分组和 block 之间的关系!如果还有其他问题,请告诉我。
if isinstance(items, list):
data = process_map(self._preprocess, items, max_workers=n_cpu, chunksize=10) 所以一个items包含了两个蛋白质,将蛋白质1的所有残基转换为block1,而将蛋白质2的所有残基转换为了block2,是这样么?
是的,理解是正确的。在这段代码中,items 代表的是输入数据,其中包含了两个蛋白质的原子信息。当 items 是一个列表时,process_map(self._preprocess, items) 会将列表中的每一项传递给 self._preprocess 函数进行处理。
具体流程:
-
items包含了两个蛋白质的结构数据:items['atoms_interface1']:包含了蛋白质 1 的所有原子信息。items['atoms_interface2']:包含了蛋白质 2 的所有原子信息。
-
在
_preprocess函数中:blocks1 = df_to_blocks(item['atoms_interface1'])会将 蛋白质 1 的所有残基转换为blocks1,即一个包含每个残基的“block”的列表。blocks2 = df_to_blocks(item['atoms_interface2'])会将 蛋白质 2 的所有残基转换为blocks2,同样也是一个包含每个残基的“block”的列表。
-
最后,
blocks1和blocks2分别表示两个蛋白质的残基结构,这两个列表会被传递到blocks_to_data(blocks1, blocks2)中进行进一步处理,可能是用于对接或相互作用分析。
结论:
是的,_preprocess 函数的作用就是将蛋白质 1 的残基转换为 blocks1,将蛋白质 2 的残基转换为 blocks2,每个 block 都代表一个残基及其包含的所有原子信息。
这里的blocks1和blocks2是指什么
在这段代码中,blocks1 和 blocks2 是从 item['atoms_interface1'] 和 item['atoms_interface2'] 中提取出来的数据。它们代表了两个不同的分子界面(interface1 和 interface2)中的残基和原子的分组信息,经过 df_to_blocks() 函数处理后被表示为“block”的形式。
blocks1 和 blocks2 具体指什么?
-
blocks1:是从item['atoms_interface1']中提取出来的“block”,表示分子界面 1(interface1)中按残基分组的原子列表。这个分子界面包含了某个蛋白质或分子片段的一部分(通常是参与对接或相互作用的表面区域)。 -
blocks2:是从item['atoms_interface2']中提取出来的“block”,表示分子界面 2(interface2)中按残基分组的原子列表。这个界面通常是与blocks1进行相互作用的另一部分(例如另一个蛋白质或分子)。
每个 block 是由一个残基的所有原子组成的对象,可能包含残基的符号和它的原子坐标、原子类型等信息。
df_to_blocks() 函数的作用:
df_to_blocks()函数将 DataFrame 中的原子信息按残基分组,生成多个Block对象,每个Block包含一个残基的所有原子信息。blocks1和blocks2代表两个不同分子界面中的所有残基,每个界面都被分成若干Block。
示例场景:
假设 item['atoms_interface1'] 和 item['atoms_interface2'] 分别包含两个不同蛋白质(或分子)的界面原子信息,那么:
blocks1会包含interface1中所有残基及其原子的分组;blocks2会包含interface2中所有残基及其原子的分组。
这些分子界面信息可能被用于计算两个蛋白质之间的对接相互作用,或者分析它们的几何结构。
blocks_to_data(blocks1, blocks2) 的作用:
- 这个函数(虽然代码中没有定义)看起来是将
blocks1和blocks2结合在一起,可能用于计算两个界面的相互作用或距离矩阵,或者其他结构相关的指标。它会将两个界面的“block”数据转化为模型需要的输入形式。
data['label'] = item['affinity']['neglog_aff']:
- 最后一步将
item['affinity']['neglog_aff']赋值给data['label'],表示这个样本的目标值(label),通常是一个关于亲和力的数值(例如结合自由能的负对数),用作监督学习中的标签。
总结:
blocks1和blocks2是分别从item['atoms_interface1']和item['atoms_interface2']中提取出来的“block”对象,代表了按残基分组的原子信息。- 它们代表了两个分子界面的结构,这些结构可能用于进一步的相互作用分析或模型输入。
blocks_to_data的函数
这段代码实现了 blocks_to_data 函数,它的作用是将多个 Block 对象的列表转化为统一的数据格式,通常用于神经网络模型的输入。具体来说,它将 Block 中的各类数据(如原子类型、原子坐标、残基类型等)整合成模型需要的输入格式。以下是对每个步骤的详细分析:
函数功能:
- 输入:函数接受若干个
blocks_list参数,每个参数是一个Block对象的列表(即多个残基的集合)。 - 输出:函数返回一个字典,字典中包含了处理后的所有
Block数据,组织成统一格式,方便后续处理。
主要步骤:
-
初始化变量:
B: 存储每个 block(残基)的类型,来自VOCAB.symbol_to_idx。A: 存储原子的类型,来自VOCAB.get_atom_global_idx()和block.to_data()。X: 存储原子的坐标信息。atom_positions: 存储原子位置的索引,用于进一步计算原子的几何信息。block_lengths: 记录每个 block(残基)包含的原子数量。segment_ids: 记录这些 block 属于哪个分子或片段(即哪一个blocks_list)。
-
处理每个
blocks_list:- 外层循环
for i, blocks in enumerate(blocks_list)遍历输入的多个blocks_list(每个界面或分子),并逐一处理其中的blocks(代表每个残基)。
- 外层循环
-
处理每个 block:
- 对于每个
blocks_list,首先创建一个 "global node"(全局节点),即代表整个分子的虚拟中心点,用于保存整个分子的全局信息。 - 每个
Block对象调用block.to_data()方法,该方法返回b(残基类型)、a(原子类型)、x(原子坐标)、positions(原子位置索引)和block_len(该残基包含的原子数)。
- 对于每个
-
计算全局节点的中心点:
cur_X[0] = np.mean(cur_X[1:], axis=0):该操作将当前Block的所有原子坐标(去掉全局节点本身)取平均值,并将结果存储为全局节点的坐标。也就是说,"全局节点" 的坐标是所有原子坐标的质心。
-
生成段 ID:
cur_segment_ids = [i for _ in cur_B]:创建一个segment_ids列表,用于标记这些 block 来自于哪个blocks_list(可以理解为它们属于哪个分子或片段)。
-
将当前 block 的信息存入全局列表:
B.extend(cur_B)、A.extend(cur_A)等操作将处理好的 block 数据添加到全局变量中,最终将所有 block 的信息合并在一起。
-
返回数据:
- 最终返回的数据是一个字典
data,其中包含了处理好的所有 block 和原子信息。这些信息包括:X: 原子的坐标数组。B: 残基类型的索引。A: 原子类型的索引。atom_positions: 原子的几何位置信息。block_lengths: 每个残基包含的原子数量。segment_ids: 每个残基所属的分子片段。
- 最终返回的数据是一个字典
详细字段解释:
-
X:- 形状为
[Natom, 2, 3],表示每个原子的三维坐标。X[0]是全局节点的坐标(残基的中心),其余的是残基中每个原子的坐标。
- 形状为
-
B:B表示每个残基的类型。它是一个列表,每个元素对应一个残基,表示残基的种类(由VOCAB.symbol_to_idx()转换)。
-
A:A是所有原子的类型列表,每个原子通过VOCAB.get_atom_global_idx()或block.to_data()得到其类型。
-
atom_positions:atom_positions记录每个原子的位置信息,它可能是用于更复杂的几何计算或进一步分析。
-
block_lengths:block_lengths记录每个残基包含的原子数量。每个残基有不同数量的原子,这个列表提供了每个残基的长度信息。
-
segment_ids:segment_ids用于标记这些 block 属于哪个片段(如不同的分子或不同的界面),使得不同blocks_list可以区分开。
总结:
blocks_to_data 函数的核心目的是将 Block 对象的列表转化为一个统一的数据格式,包括残基类型、原子类型、坐标等信息。这些数据会被用于后续的神经网络模型或其他分析任务。
形状为 [Natom, 2, 3] 这里的维度2是什么意思
在 X 的形状 [Natom, 2, 3] 中,维度 2 对应的是每个原子的两种坐标表示。具体解释如下:
-
Natom: 表示所有原子的数量。这个维度的大小取决于输入数据中总共包含的原子数(包括所有 block 中的原子)。 -
2: 这个维度表示两种不同的坐标表示方式:- 第一种表示(
X[:, 0, :])通常是指全局节点的坐标,即代表整个残基或整个分子片段的质心坐标(block 中所有原子坐标的平均值)。 - 第二种表示(
X[:, 1, :])是原子的实际坐标,即每个原子相对于残基的三维空间位置。
- 第一种表示(
-
3: 表示每个原子的三维空间坐标(即x, y, z三个轴的坐标)。这个维度的大小始终为 3,表示三维坐标系中的位置。
详细解释:
-
X[:, 0, :]: 对应的是每个 block 的全局坐标,即质心(或残基的中心位置)。这种坐标表示是通过np.mean(cur_X[1:], axis=0)计算得出的,表示该 block 中所有原子的平均坐标。 -
X[:, 1, :]: 对应的是原子的实际坐标,表示该原子在三维空间中的x, y, z位置。
举个例子:
假设有 2 个原子,X 的形状为 [2, 2, 3]:
X = [
[[x_global1, y_global1, z_global1], [x_atom1, y_atom1, z_atom1]],
[[x_global2, y_global2, z_global2], [x_atom2, y_atom2, z_atom2]]
]
-
对于第一个原子:
X[0, 0, :]是全局节点的坐标(残基的中心或质心坐标)。X[0, 1, :]是该原子的具体坐标。
-
对于第二个原子:
X[1, 0, :]是另一个 block 的全局坐标。X[1, 1, :]是第二个原子的具体坐标。
总结:
- 维度
2中的第一个位置存储全局节点的坐标(质心),而第二个位置存储实际的原子坐标。通过这种方式,每个原子都有两种坐标表示,既有它本身的坐标,也有全局坐标。
index里面这些变量是什么
从你提供的截图来看,self.indexes 是一个包含字典的列表,每个字典对应一个蛋白质-配体对的结合数据。这些变量存储的是蛋白质和配体的亲和力及其相关的物理化学数据。下面是这些变量的具体含义:
每个字典中的键值解释:
-
affinity:- 包含结合亲和力的相关信息,通常用于评估蛋白质与配体之间的结合强度。
Kd:解离常数(dissociation constant),表示蛋白质和配体在平衡时的结合强度。Kd 值越小,表示结合强度越高。dG:自由能变化(Gibbs free energy change),通常是由解离常数计算得出,代表了结合反应的能量变化。通常,dG 值越低,结合越稳定。neglog_aff:亲和力的负对数值,可能用于处理 Kd 值较小时的数值稳定性。
-
id:- 这是蛋白质-配体对的唯一标识符(通常是 PDB ID),每个 PDB ID 对应一种特定的蛋白质结构或蛋白质-配体复合体。
具体示例:
-
affinity:
'Kd': 5.59e-07:表示蛋白质-配体的解离常数为5.59 × 10^-7 M,表明蛋白质和配体结合较紧密。'dG': -8.53:自由能变化为-8.53 kcal/mol,结合的稳定性较高。'neglog_aff': 6.25:可能是亲和力的负对数值,通常用于评估结合强度。
-
id:
- 例如,
'id': '3bpl':这是蛋白质-配体对的唯一标识符,在 PDB 数据库中可能查找到这个复合物的结构。
- 例如,
总结:
self.indexes 是一个包含多个蛋白质-配体对的数据列表,每个字典包含了:
- 亲和力相关的数值(
Kd,dG,neglog_aff)。 - 蛋白质-配体对的唯一标识符(
id)。
这些信息可能用于分析蛋白质与配体的相互作用,以及模型的输入或标签(label)。
是的,data['label'] = item['affinity']['neglog_aff'] 这行代码的作用是将结合强度(以负对数亲和力 neglog_aff 的形式)提取出来,并将其作为 data['label'],用于模型的标签(label)。
具体解释:
-
item['affinity']['neglog_aff']:这里的neglog_aff是亲和力的负对数(通常是-log(Kd),其中 Kd 是解离常数),用于衡量蛋白质与配体的结合强度。- Kd (解离常数):Kd 值越小,表明蛋白质和配体结合越紧密。为了便于数值计算和处理,Kd 常常以负对数形式表示,因此
neglog_aff越大,表示结合强度越强。
- Kd (解离常数):Kd 值越小,表明蛋白质和配体结合越紧密。为了便于数值计算和处理,Kd 常常以负对数形式表示,因此
-
data['label']:将提取的neglog_aff值赋值给data['label'],这个label将作为模型的目标值,用于监督学习或预测蛋白质-配体之间的结合强度。
为什么使用 neglog_aff?
使用亲和力的负对数(neglog_aff)而不是 Kd 值的原因是:
- 数值稳定性:Kd 值的范围可能非常广,从 10^-9 到 10^-3 甚至更大。直接使用 Kd 值进行计算可能导致数值不稳定。通过对 Kd 取负对数,可以缩小数值范围,使其更适合用于机器学习模型。
- 线性关系:在某些情况下,
-log(Kd)与结合强度的物理化学过程有更好的线性关系,因此更适合用于回归模型。
总结:
这行代码是提取蛋白质-配体结合强度的 neglog_aff 值,并将其作为标签(label),用于模型训练或评估结合强度的任务。
以batch为单位的数据
根据你提供的截图内容,下面是对各个变量的解释以及它们可能对应的数据含义:
-
A:
- 内容:
tensor([2, 9, 8, ..., 8, 10, 8], device='cuda:0') - 解释: 这是一个表示原子索引的 Tensor,通常对应的是某种类型的原子属性或索引。它可能是每个原子所对应的类型(例如不同元素的类别)。
- 内容:
-
B:
- 内容:
tensor([3, 2, 7, ..., 12, 14, 5], device='cuda:0') - 解释: 这个变量通常表示块(block)或残基(residue)的索引或类型。它可能是表示不同残基或块的分类信息。
- 内容:
-
Z:
- 内容:
tensor([[[0.2207, 17.2229, -14.8194], [-11.4810, 22.9620, -30.7930]]], device='cuda:0') - 解释:
Z表示原子的三维坐标信息。这个 Tensor 的形状是(8548, 1, 3),表示 8548 个原子的三维坐标。这通常用于计算分子之间的距离或作为模型输入。
- 内容:
-
atom_positions:
- 内容:
tensor([2, 3, 4, ..., 3, 3, 5], device='cuda:0') - 解释: 这是每个原子的位置信息,可能是原子在结构中的顺序或与三维坐标有关的索引。结合
Z使用时,它可以帮助定位每个原子的具体位置。
- 内容:
-
block_lengths:
- 内容:
tensor([1, 8, 8, ..., 8, 8, 5], device='cuda:0') - 解释: 这是一个记录每个块(block)或残基(residue)包含多少原子的 Tensor。它可以帮助定义每个块的大小和结构。
- 内容:
-
label:
- 内容:
tensor([10.3841, 5.7959, 5.0969, ..., 9.7696], device='cuda:0') - 解释: 这是目标变量的标签信息,通常用于表示结合亲和力(affinity)或其他分子间属性。这些标签是模型训练时的目标值。
- 内容:
-
lengths:
- 内容:
tensor([92, 111, 82, ..., 47, 175, 39], device='cuda:0') - 解释: 这是每个样本(分子或块)的长度信息,表示每个分子或残基块的长度,用于动态批处理或其他模型输入处理。
- 内容:
-
return_loss:
- 内容:
False - 解释: 这是一个布尔值,表示是否返回损失值(loss)。它通常用于控制在训练过程中是否需要计算并返回损失值。
- 内容:
-
return_noise:
- 内容:
False - 解释: 这是一个布尔值,表示是否返回噪声信息。它可能用于控制在一些模型中是否引入噪声以进行数据增强或模拟不同情况。
- 内容:
-
segment_ids:
- 内容:
tensor([0, 0, 0, ..., 1, 1, 1], device='cuda:0') - 解释: 这是一个表示分段信息的 Tensor,用于标记数据中不同片段的边界。它可能用于区分来自不同分子或残基的部分。
- 内容:
-
self (AffinityPredictor):
- 解释: 这是模型的实例对象(
AffinityPredictor),它可能是你正在使用的模型类,专门用于预测分子亲和力或其他相关任务。这个模型可能包含了一个块嵌入层(BlockEmbedding),用于将分子块嵌入为向量表示。
- 解释: 这是模型的实例对象(
总结:
这些变量是模型输入数据的不同部分。大致可以归类为以下几类:
- 几何和结构信息:如
Z(三维坐标)、atom_positions、block_lengths、lengths。 - 标签和目标值:如
label(亲和力标签)。 - 模型的控制和处理标志:如
return_loss和return_noise。 - 其他辅助信息:如
segment_ids和A、B(分别表示原子和块的索引或类型)。
这些信息结合起来构成了用于分子建模和预测的输入数据,以及用于控制训练过程的标志。
边序号的构建
在这段代码中,construct_edges 函数的主要作用是根据输入的块(block)、批次(batch)、段(segment)等信息,利用 edge_constructor 来构建 边集(edges),这些边表示分子结构或其他复杂结构中各节点(如原子、块)之间的连接关系。
边的构建步骤
-
不进行切片的边构建:
当complexity == -1时,不进行任何切片操作,直接调用edge_constructor来构建所有边:intra_edges, inter_edges, global_global_edges, global_normal_edges, _ = edge_constructor(B, batch_id, segment_ids, X=X, block_id=block_id) return intra_edges, inter_edges, global_global_edges, global_normal_edges在这里,
edge_constructor会直接生成以下几种边:intra_edges: 在块(block)内部的边。inter_edges: 块与块之间的边。global_global_edges: 全局节点之间的边。global_normal_edges: 全局节点与普通节点之间的边。
-
进行切片的边构建:
当complexity != -1时,会根据指定的复杂度complexity对数据进行切片。每次处理数据的一个小批次(mini-batch),并为每个小批次构建边,最后再合并这些边。具体过程如下:
-
初始化部分变量:
offset, bs_id_start, bs_id_end = 0, 0, 0 mini_intra_edges, mini_inter_edges, mini_global_global_edges, mini_global_normal_edges = [], [], [], [] batch_size = batch_id.max() + 1 unit_batch_id = batch_id[block_id] lengths = scatter_sum(torch.ones_like(batch_id), batch_id, dim=0)这里
batch_size是指总的批次数量,unit_batch_id表示每个原子的批次ID,lengths计算每个批次中的块的数量。 -
逐个批次处理:
while bs_id_end < batch_size: bs_id_start = bs_id_end bs_id_end += 1 while bs_id_end + 1 <= batch_size and \ (lengths[bs_id_start:bs_id_end + 1] * lengths[bs_id_start:bs_id_end + 1].max()).sum() < complexity: bs_id_end += 1这部分代码用于迭代处理每个批次的数据,同时根据
complexity控制每个小批次的复杂度。lengths[bs_id_start:bs_id_end + 1]计算当前批次中块的数量,用于决定是否要继续向下一个批次扩展。 -
选择当前批次中的块和原子:
block_is_in = (batch_id >= bs_id_start) & (batch_id < bs_id_end) unit_is_in = (unit_batch_id >= bs_id_start) & (unit_batch_id < bs_id_end) B_mini, batch_id_mini, segment_ids_mini = B[block_is_in], batch_id[block_is_in], segment_ids[block_is_in] X_mini, block_id_mini = X[unit_is_in], block_id[unit_is_in]这里
block_is_in和unit_is_in用于选择当前批次中的块和原子。B_mini、batch_id_mini等变量存储的是当前小批次中的块、批次ID、段ID、位置等信息。 -
为当前批次构建边:
intra_edges, inter_edges, global_global_edges, global_normal_edges, _ = edge_constructor( B_mini, batch_id_mini - bs_id_start, segment_ids_mini, X=X_mini, block_id=block_id_mini - offset)对于当前小批次,通过
edge_constructor构建边集,包括块内边、块间边、全局边等。 -
累加偏移量:
offset += B_mini.shape[0]由于在每个小批次之间的索引会重新计算,因此需要通过
offset来保持原始的全局索引。 -
将小批次的边合并:
通过检查edge_constructor是否有提供given_intra_edges和given_inter_edges,如果没有的话,将小批次中的边添加到合并后的列表中,最终将这些小批次的边合并成全局的边:if hasattr(edge_constructor, 'given_intra_edges'): intra_edges = edge_constructor.given_intra_edges else: intra_edges = torch.cat(mini_intra_edges, dim=1) if hasattr(edge_constructor, 'given_inter_edges'): inter_edges = edge_constructor.given_inter_edges else: inter_edges = torch.cat(mini_inter_edges, dim=1) if global_global_edges is not None: global_global_edges = torch.cat(mini_global_global_edges, dim=1) if global_normal_edges is not None: global_normal_edges = torch.cat(mini_global_normal_edges, dim=1)
-
边的类型总结
最终返回的边包括以下几种类型:
- intra_edges: 块内部的边,即块中原子之间的边。
- inter_edges: 块之间的边,即不同块中的原子之间的边。
- global_global_edges: 全局节点之间的边,用于捕捉全局信息。
- global_normal_edges: 全局节点与普通节点之间的边,用于连接全局和局部信息。
这些边将被用于进一步的图神经网络中,用来建模分子结构的几何和拓扑信息。
如果你有关于这个过程的具体问题,欢迎进一步提问!
也就是说它这里的中心节点和残基类型有关么
是的,self.global_block_id = VOCAB.symbol_to_idx(VOCAB.GLB) 这部分代码表明,中心节点(global node)的构建与 残基类型(block type) 有关。
具体原因如下:
-
残基类型映射(Residue Type Mapping):
VOCAB.symbol_to_idx是一个将符号(例如残基类型)映射为索引的函数。- 在这种情况下,
VOCAB.GLB是一个特殊的符号,代表中心节点或全局节点。 symbol_to_idx(VOCAB.GLB)将这个符号映射为一个整数索引,用作模型中的全局节点 ID(即global_block_id)。
-
残基类型的作用:
- 不同的残基类型(例如
ALA、GLY等)通常具有不同的特性,因此这些类型可能会影响中心节点的构建。 - 全局节点(中心节点)会代表整个残基块的全局信息,因此残基类型的索引决定了全局节点的具体 ID。
- 不同的残基类型(例如
-
中心节点与残基的关系:
- 每个残基或块都会有一个对应的中心节点,这个节点的作用是汇总残基内部的所有信息,或与其他残基的中心节点通信。
- 因此,中心节点的构建依赖于残基的类型,具体表现在残基的类型决定了该残基的中心节点的 ID(
global_block_id)。
-
全局节点的特性:
- 由于全局节点是根据残基类型生成的,因此在模型中,中心节点可以作为残基在高层次上的表示(如同残基的一个超级节点),连接其他残基的全局节点或者其他结构。
- 全局节点还可以用于跨越不同层次的图结构,帮助实现全局信息的传播。
结论:
是的,中心节点(global node)和残基类型有关。残基的类型决定了中心节点的 ID(通过 VOCAB.symbol_to_idx 映射得到),从而在构建图结构时,中心节点能够反映该残基类型的全局信息。这种机制允许模型在残基的局部和全局层次上处理和传递信息。
self.global_block_id = VOCAB.symbol_to_idx(VOCAB.GLB) 的意思是:
-
VOCAB.GLB: 这是一个特殊的全局标识符(GLB),用于表示全局节点的符号。例如,在残基或块的表示中,GLB可能代表这个块的全局节点符号,用于图中的全局节点信息传递。 -
VOCAB.symbol_to_idx(): 这是一个方法,接受一个符号(symbol)作为输入,并返回该符号对应的 索引(ID)。在这个例子中,VOCAB.symbol_to_idx(VOCAB.GLB)将VOCAB.GLB这个全局节点的符号转换为相应的索引值(global_block_id)。 -
symbol2idx: 在VOCAB类中,symbol2idx是一个 字典,它将符号映射为索引。比如,symbol2idx中可能包含类似的映射:symbol2idx = { 'ALA': 0, 'GLY': 1, 'VAL': 2, 'GLB': 999, # Example mapping for global node # ... }symbol_to_idx函数使用symbol2idx字典来查找符号对应的索引。 -
UNK: 如果符号在symbol2idx字典中不存在,symbol_to_idx方法会使用abrv2idx['UNK'],即一个 默认的未知符号索引。UNK是 "unknown" 的缩写,表示未知或未定义的符号。
具体过程:
- 输入符号:
VOCAB.GLB是全局节点的符号。 - 查找索引: 调用
VOCAB.symbol_to_idx(VOCAB.GLB),从symbol2idx字典中查找GLB符号对应的索引。 - 返回索引: 如果
GLB符号存在于symbol2idx中,返回相应的索引值,赋值给self.global_block_id。如果不存在,则返回UNK的索引。
例子:
假设 symbol2idx 字典中有如下映射:
symbol2idx = {
'ALA': 0,
'GLY': 1,
'GLB': 999, # Global node
# ...
}
abrv2idx = {
'UNK': -1 # Unknown token
}
那么 VOCAB.symbol_to_idx(VOCAB.GLB) 将返回 999,并赋值给 self.global_block_id。
总结:
self.global_block_id是从VOCAB.GLB这个符号通过symbol_to_idx方法得到的索引,用于标识全局节点。symbol_to_idx方法通过查找symbol2idx字典,找到对应的符号索引。如果符号不存在,返回默认的未知符号索引UNK。
k_neighbors是9
当 k_neighbors 设置为 9 时,这意味着每个残基或原子将最多与其最近的 9 个邻居 构建边。这些邻居是基于它们的 空间距离 计算出来的。具体的构建过程如下:
1. 块内边构建 (_construct_intra_edges)
- 在每个块内部,残基或原子之间会通过 K 最近邻算法构建边,最多连接到最近的 9 个邻居。这确保了每个残基或原子与周围的邻近结构形成紧密连接。
- 例如,假设有一个残基包含多个原子,KNN 会选择距离最近的 9 个原子,并在它们之间构建边。
2. 块间边构建 (_construct_inter_edges)
- 对于不同块(或残基)之间,也会通过 K 最近邻算法构建边,确保每个块与最近的 9 个其他块相连。
- 这意味着,每个残基不仅会与自己内部的原子构建边,还会与最近的 9 个其他残基建立连接。这个连接可以跨越分子结构中的不同部分。
3. KNN 的作用
- KNN 限制了每个节点(残基或原子)最多只与 9 个邻居相连,避免了过多的边,减少计算复杂度。同时,这种构建方式可以确保在复杂的结构中,每个节点仍然与其最近的邻居保持紧密联系。
- 在复杂的分子或蛋白质结构中,残基之间的距离可能会变化很大。KNN 的策略可以确保只选择最相关的连接,忽略与距离较远的残基之间的连接,从而减少冗余信息。
4. 计算边的数量
- 每个残基或原子都会通过距离最近的 9 个邻居进行连接,因此每个节点的边数量上限是 9 条(除非它的邻居少于 9 个)。
- 对于不同块(或残基)之间的连接,同样会构建最多 9 条边。
总结:
当 k_neighbors=9 时,意味着每个残基(或块)会与它最接近的 9 个邻居 构建边。这些边可以是块内部的原子之间的边,也可以是块之间的连接边。通过这种方式,模型能在残基之间保留合理的连接数量,避免产生过多的无关连接,同时保持足够的局部信息传递。
在 construct_edges 函数中,边的构建分为四类:块内部的边(intra_edges)、块之间的边(inter_edges)、全局节点之间的边(global_global_edges)、全局节点和普通节点之间的边(global_normal_edges)。每一类边代表不同的结构和连接类型,下面详细解释每类边的构建逻辑:
1. 块内部的边(intra_edges)
-
定义:块内部的边是指同一残基(或块)内部的原子之间的连接。
-
构建方式:
intra_edges通过_construct_intra_edges函数构建,它会调用 K 最近邻(KNN)算法,为块内部的原子构建边。这意味着每个原子会与其距离最近的 9 个邻居(由k_neighbors=9控制)相连。- 在实际构建过程中,首先会从父类的
super()._construct_intra_edges函数中得到所有候选内部边,然后基于它们的空间位置(由X决定)计算距离,选择最接近的 9 个原子构建 KNN 边。
def _construct_intra_edges(self, S, batch_id, segment_ids, **kwargs): all_intra_edges = super()._construct_intra_edges(S, batch_id, segment_ids) X, block_id = kwargs['X'], kwargs['block_id'] src_dst = all_intra_edges.T dist = _block_edge_dist(X, block_id, src_dst) intra_edges = _knn_edges(dist, src_dst, self.k_neighbors, (self.offsets, batch_id, self.max_n, self.gni2lni)) return intra_edges该方法的具体流程是:
- 使用
super()._construct_intra_edges获取候选内部边的起点和终点。 - 调用
_block_edge_dist计算这些候选边的空间距离。 - 使用
_knn_edges函数,通过 KNN 算法,基于距离选择最近的k_neighbors个邻居,构建intra_edges。
2. 块之间的边(inter_edges)
-
定义:块之间的边是指不同残基(或块)之间的连接,连接的是不同块内的原子。
-
构建方式:
inter_edges通过_construct_inter_edges函数构建,方法与intra_edges类似,使用 KNN 算法来选择不同块(或残基)之间的最近邻原子连接。- 构建步骤也包括先调用父类方法获取候选的块间边,然后通过计算这些边的空间距离,选择最接近的 9 个邻居构建 KNN 边。
def _construct_inter_edges(self, S, batch_id, segment_ids, **kwargs): all_inter_edges = super()._construct_inter_edges(S, batch_id, segment_ids) X, block_id = kwargs['X'], kwargs['block_id'] src_dst = all_inter_edges.T dist = _block_edge_dist(X, block_id, src_dst) inter_edges = _knn_edges(dist, src_dst, self.k_neighbors, (self.offsets, batch_id, self.max_n, self.gni2lni)) return inter_edges流程:
- 通过
super()._construct_inter_edges获取候选块间边。 - 计算这些候选块间边的空间距离(基于
X提供的原子坐标)。 - 使用 KNN 筛选出最近的 9 个邻居,并构建
inter_edges。
3. 全局节点之间的边(global_global_edges)
-
定义:全局节点之间的边是指全局节点(通常是残基或块的中心节点)之间的连接。这些节点用于在全局范围内传递信息。
-
构建方式:
- 如果启用了全局消息传递(即
global_message_passing=True),则通过_construct_global_edges来构建全局节点之间的边。 - 这些边的构建通常是通过检查残基或块的中心位置或全局信息来决定的。
def _construct_global_edges(self, S, batch_id, segment_ids, **kwargs): if self.global_message_passing: return super()._construct_global_edges(S, batch_id, segment_ids, **kwargs) else: return None, None流程:
- 如果
global_message_passing=True,则调用父类方法super()._construct_global_edges构建全局节点之间的边。 - 如果
global_message_passing=False,则不构建全局边,返回None。
- 如果启用了全局消息传递(即
4. 全局节点和普通节点之间的边(global_normal_edges)
- 定义:全局节点和普通节点之间的边是指连接全局节点(如残基的中心节点)与普通原子节点的边。这些边用于在全局节点与局部节点之间传递信息。
- 构建方式:
global_normal_edges通常是在全局节点和普通节点之间进行连接,具体构建方式与global_global_edges类似。- 如果全局消息传递是启用的,这类边也会被构建,连接全局节点与普通节点,帮助信息在局部和全局之间传播。
5. 边的汇总与拼接
在整个边构建的过程中,construct_edges 函数会逐批处理每个块和单位(单位是残基中的原子),根据每个批次内的复杂度(例如边的数量)来控制每次构建的边数。最后,将构建的边分别拼接成完整的边集合:
if hasattr(edge_constructor, 'given_intra_edges'):
intra_edges = edge_constructor.given_intra_edges
else:
intra_edges = torch.cat(mini_intra_edges, dim=1)
对于每一类边,最终将所有的小批次边拼接成一个完整的边集合。包括:
intra_edges: 块内部的边。inter_edges: 块之间的边。global_global_edges: 全局节点之间的边。global_normal_edges: 全局节点和普通节点之间的边。
总结:
- 块内部的边 (
intra_edges): 连接同一块内部的原子,通过 KNN 算法选择距离最近的k_neighbors=9个原子。 - 块之间的边 (
inter_edges): 连接不同块(残基)之间的原子,同样通过 KNN 算法选择最近的 9 个邻居。 - 全局节点之间的边 (
global_global_edges): 如果启用全局消息传递,连接全局节点(通常是块的中心节点)之间的边。 - 全局节点和普通节点之间的边 (
global_normal_edges): 如果启用全局消息传递,连接全局节点和普通节点的边。
通过这些边的构建,模型能够在局部和全局层面上捕捉到残基和原子之间的关系,帮助信息在图结构中有效传播。
批次化的处理边
这个代码的逻辑用于 构建批次内 的边连接,包括残基内部的边(intra_edges)、残基之间的边(inter_edges)、全局节点到全局节点的边(global_global_edges)、全局节点到普通节点的边(global_normal_edges)。这段代码通过分批次处理构建边,避免在一次处理中处理太多的边和节点,确保模型在处理复杂度较高的图时,仍然能够合理地进行计算。
下面逐步解释整个代码的逻辑:
1. 变量初始化
offset, bs_id_start, bs_id_end = 0, 0, 0
mini_intra_edges, mini_inter_edges, mini_global_global_edges, mini_global_normal_edges = [], [], [], []
offset:偏移量,用于在小批次中调整边的索引,以保证多个小批次的边索引能够正确连接到全局。bs_id_start和bs_id_end:用于记录当前处理的小批次的起始和结束位置,按照batch_id来划分小批次。mini_intra_edges、mini_inter_edges等:用于暂存当前小批次中的边,稍后将它们拼接成完整的边集合。
2. 获取批次大小和单位(unit)的批次 ID
batch_size = batch_id.max() + 1 # 获取批次大小
unit_batch_id = batch_id[block_id] # 根据 block_id 提取单位的 batch_id
lengths = scatter_sum(torch.ones_like(batch_id), batch_id, dim=0) # 计算每个 batch_id 对应的长度
batch_size:当前整个数据的批次大小,即有多少个不同的batch_id。unit_batch_id:提取与block_id对应的单位(atom)的batch_id,用于将这些单位划分到不同的小批次中。lengths:记录每个batch_id(即每个小批次)中包含的元素个数。
3. 划分小批次并构建边
while bs_id_end < batch_size:
bs_id_start = bs_id_end
bs_id_end += 1
while bs_id_end + 1 <= batch_size and \
(lengths[bs_id_start:bs_id_end + 1] * lengths[bs_id_start:bs_id_end + 1].max()).sum() < complexity:
bs_id_end += 1
- 外部的
while循环控制小批次的起始位置和结束位置。 - 内部的
while循环用来动态地增加bs_id_end,直到该小批次的复杂度(即节点数量和边的复杂度的乘积)达到给定的complexity限制。
3.1 划分批次中包含的残基和单位
block_is_in = (batch_id >= bs_id_start) & (batch_id < bs_id_end)
unit_is_in = (unit_batch_id >= bs_id_start) & (unit_batch_id < bs_id_end)
B_mini, batch_id_mini, segment_ids_mini = B[block_is_in], batch_id[block_is_in], segment_ids[block_is_in]
X_mini, block_id_mini = X[unit_is_in], block_id[unit_is_in]
block_is_in和unit_is_in:通过条件判断,确定哪些残基和单位属于当前小批次范围。B_mini、batch_id_mini等:获取当前小批次中的残基和单位信息,用于构建边。
3.2 构建小批次中的边
intra_edges, inter_edges, global_global_edges, global_normal_edges, _ = edge_constructor(
B_mini, batch_id_mini - bs_id_start, segment_ids_mini, X=X_mini, block_id=block_id_mini - offset)
- 调用
edge_constructor函数,针对当前的小批次数据,构建内部边(intra_edges)、外部边(inter_edges)、全局到全局的边(global_global_edges)以及全局到普通节点的边(global_normal_edges)。 - 注意这里对
batch_id_mini和block_id_mini进行了调整,以确保它们在当前小批次范围内使用正确的索引(通过减去bs_id_start和offset)。
3.3 拼接小批次的边
if not hasattr(edge_constructor, 'given_intra_edges'):
mini_intra_edges.append(intra_edges + offset)
if not hasattr(edge_constructor, 'given_inter_edges'):
mini_inter_edges.append(inter_edges + offset)
if global_global_edges is not None:
mini_global_global_edges.append(global_global_edges + offset)
if global_normal_edges is not None:
mini_global_normal_edges.append(global_normal_edges + offset)
- 对每个构建好的小批次边集合,检查是否存在全局的边约束。如果没有全局边约束,则将小批次的边加上偏移量
offset后,拼接到mini_intra_edges等列表中。 - 每个小批次的边构建完后,将其加入到完整的边集合中,确保多个小批次的边能够在全局图中正确连接。
4. 拼接所有小批次的边
if hasattr(edge_constructor, 'given_intra_edges'):
intra_edges = edge_constructor.given_intra_edges
else:
intra_edges = torch.cat(mini_intra_edges, dim=1)
if hasattr(edge_constructor, 'given_inter_edges'):
inter_edges = edge_constructor.given_inter_edges
else:
inter_edges = torch.cat(mini_inter_edges, dim=1)
if global_global_edges is not None:
global_global_edges = torch.cat(mini_global_global_edges, dim=1)
if global_normal_edges is not None:
global_normal_edges = torch.cat(mini_global_normal_edges, dim=1)
- 经过循环处理所有的小批次后,最终将所有的
mini_intra_edges、mini_inter_edges、mini_global_global_edges和mini_global_normal_edges进行拼接,形成完整的边集合。 - 如果
edge_constructor已经提供了全局的边约束,则使用全局的边集合,而不需要重新构建。
5. 返回构建好的边
return intra_edges, inter_edges, global_global_edges, global_normal_edges
- 最终返回所有构建好的边,包括内部边、外部边和全局边。
总结:
- 批次划分:代码通过循环将批次划分成多个小批次,每个小批次中的残基和单位都会被分配到合适的组。
- 边的构建:在每个小批次中,调用
edge_constructor函数来构建边,包括内部边、外部边和全局边。 - 边的拼接:每个小批次的边都会加上偏移量后拼接,保证多个小批次的边连接到全局图中不会出现错误。
- 顺序不会影响边构建:即使批次划分是顺序进行的,所有小批次的残基都会被处理,且最终的边集合会覆盖整个批次中的所有残基。
不会出现残基因为先后顺序而没有边的情况,代码已经处理了这些情况。
每种边会有不同的embedding
是的,在这个代码片段中,如果 self.global_message_passing 为 True,则 四种不同类型的边 会有不同的嵌入(embedding)处理。具体分析如下:
1. 四种边类型的嵌入(embedding)区别
当 self.global_message_passing=True 时,edges 包含四种不同类型的边:
intra_edges: 块内部的边,代表同一块(残基)内原子之间的连接。inter_edges: 块之间的边,代表不同块(残基)之间的连接。global_global_edges: 全局节点之间的边,连接全局节点(每个残基的中心节点)之间的边。global_normal_edges: 全局节点和普通节点之间的边,连接全局节点和残基中的普通节点。
每种边的嵌入:
edge_attr = torch.cat([
torch.zeros_like(intra_edges[0]), # 对应 intra_edges
torch.ones_like(inter_edges[0]), # 对应 inter_edges
torch.ones_like(global_global_edges[0]) * 2, # 对应 global_global_edges
torch.ones_like(global_normal_edges[0]) * 3 # 对应 global_normal_edges
])
torch.zeros_like(intra_edges[0]):对于 块内部的边(intra_edges),使用 0 作为边属性。torch.ones_like(inter_edges[0]):对于 块之间的边(inter_edges),使用 1 作为边属性。torch.ones_like(global_global_edges[0]) * 2:对于 全局节点之间的边(global_global_edges),使用 2 作为边属性。torch.ones_like(global_normal_edges[0]) * 3:对于 全局节点和普通节点之间的边(global_normal_edges),使用 3 作为边属性。
2. 嵌入操作
edge_attr = self.edge_embedding(edge_attr)
在设置好每种边的标签之后,edge_attr 会通过 self.edge_embedding 进行嵌入(embedding)。self.edge_embedding 是一个 nn.Embedding,它根据 edge_attr 的值(0, 1, 2, 3)为不同类型的边分配不同的嵌入向量。
edge_embedding(0):为块内部的边分配一个嵌入向量。edge_embedding(1):为块之间的边分配一个不同的嵌入向量。edge_embedding(2):为全局节点之间的边分配一个不同的嵌入向量。edge_embedding(3):为全局节点和普通节点之间的边分配一个不同的嵌入向量。
3. 区别在哪?
- 块内部的边(intra_edges) 和 块之间的边(inter_edges) 有不同的边属性标记(分别为0和1),因此它们经过
edge_embedding时会得到不同的嵌入。 - 全局节点之间的边(global_global_edges) 和 全局节点与普通节点之间的边(global_normal_edges) 也有不同的边属性标记(分别为2和3),因此它们会得到各自的嵌入。
4. self.global_message_passing 为 False 的情况
如果 self.global_message_passing=False,则只构建 块内部的边 和 块之间的边,没有全局节点的边:
edges = torch.cat([intra_edges, inter_edges], dim=1)
edge_attr = torch.cat([torch.zeros_like(intra_edges[0]), torch.ones_like(inter_edges[0])])
在这种情况下:
- 块内部的边 的嵌入标记为
0,块之间的边标记为1。 - 不再涉及全局节点相关的边和嵌入。
总结:
当 self.global_message_passing=True 时,不同类型的边(内部边、块间边、全局节点间边、全局节点与普通节点间边)被分别标记为 0, 1, 2, 3,并通过 self.edge_embedding 赋予它们不同的嵌入向量。这种差异化的嵌入帮助模型区分不同类型的边,使得在图卷积过程中可以对不同的边类型进行独立的处理。
边的关系
从你的描述来看,似乎想明确一下这些边在残基和原子层次上的定义。根据你假设 block 是残基的情况下,以下是每种边的具体解释:
-
intra_edges(原子与原子的边):
- 这些边是残基内部原子之间的边,表示在同一个残基(即氨基酸残基)内部,原子与原子之间的连接。比如,氨基酸的侧链、主链上的碳原子(C)、氮原子(N)和氧原子(O)之间的化学键连接。
-
inter_edges(残基与残基的边):
- 这些边表示不同残基之间的连接。由于是残基级别的边,表示相邻的残基之间的相互作用,通常是通过肽键连接的氨基酸残基。
inter_edges反映了局部残基之间的相互作用。
- 这些边表示不同残基之间的连接。由于是残基级别的边,表示相邻的残基之间的相互作用,通常是通过肽键连接的氨基酸残基。
-
global_global_edges(残基与残基的全局边):
- 这些边是残基之间的全局连接,表示在整个蛋白质分子结构中,非局部的残基之间的相互作用。全局边可以捕捉较远距离残基之间的联系,如远距离残基间通过氢键或疏水相互作用发生的作用。
-
global_normal_edges(残基与残基的全局与局部边):
- 这些边表示全局节点(通常是特定的、在整体结构中具有重要功能的残基)与普通残基之间的连接。例如,蛋白质的活性位点残基与周围其他残基的相互作用属于这种连接。这些边可以反映功能性残基与非功能性残基之间的相互作用。
总的来说,这些边都是在不同层次上对蛋白质分子进行建模的方式,intra_edges 建模了局部残基内部的原子结构,inter_edges 和 global_edges 建模了残基之间的局部和全局联系,而 global_normal_edges 则建模了功能性和非功能性残基之间的交互。
那对于残基与残基的边,是怎么计算距离的
对于残基与残基的边,距离的计算涉及到如何定义残基之间的距离。在你的代码中,_block_edge_dist 函数正是用于计算残基(block)之间的距离,以下是该函数的详细计算步骤:
1. 残基和原子的对应关系
- 每个残基由多个原子组成,因此在计算残基之间的距离时,需要先计算这些残基中原子之间的距离。代码中的
block_id表示每个原子所属的残基,X则是每个原子的坐标。
2. 提取原子对
_unit_edges_from_block_edges函数负责从给定的block_src_dst(残基对)中提取出所有可能的原子对(unit pair),即确定哪些原子与哪些原子之间会形成边。
(unit_src, unit_dst), (edge_id, _, _) = _unit_edges_from_block_edges(block_id, src_dst)
unit_src和unit_dst是原子对之间的索引,分别表示每个残基对中具体的原子对。block_src_dst是给定的残基对,用src_dst表示残基的对(例如 [残基1, 残基2])。
3. 计算原子对之间的距离
- 提取了原子对之后,开始计算原子之间的距离。首先根据
unit_src和unit_dst提取出相应的原子坐标:
src_x, dst_x = X[unit_src], X[unit_dst] # [Eu, k, 3]
X[unit_src]和X[unit_dst]提取了每对原子的三维坐标。接下来,计算它们的欧氏距离:
dist = torch.norm(src_x - dst_x, dim=-1) # [Eu, k]
- 这里
torch.norm用于计算两个原子之间的欧氏距离,dist是所有原子对之间的距离。
4. 残基对之间的最小距离
- 由于每个残基对包含多个原子对,因此需要选取这些原子对中距离最小的一对作为这两个残基之间的距离:
dist = torch.min(dist, dim=-1).values # [Eu]
torch.min用于选择每对残基中的原子对距离的最小值。此时dist就代表了每个残基对之间的最小距离。
5. 汇总残基对的距离
- 最后,使用
scatter_min汇总每个block_src_dst对应的残基之间的最小原子对距离,得到最终的残基对距离:
dist = scatter_min(dist, edge_id)[0] # [Eb]
scatter_min根据残基对的索引edge_id汇总所有原子对的最小距离,最终得到每对残基之间的距离。
总结
对于残基-残基的边,距离的计算如下:
- 先找到所有残基对中的原子对(即每个残基内部的所有原子与其他残基中原子的组合)。
- 计算每对原子的欧氏距离。
- 对每对残基,选取最小的原子对距离,作为这对残基的距离。
这种方法确保了残基之间的距离是基于它们的最靠近的原子对,从而反映了残基之间的最近接触点。
是的,正如你所理解的,对于两个残基,代码中是选取它们之间距离最小的原子对来作为这两个残基的距离。这是一个常见的策略,用于简化复杂分子结构中多个原子之间的距离度量。
具体步骤总结如下:
-
残基内部的原子:每个残基包含多个原子,残基之间的距离实际上是由这些原子之间的距离决定的。
-
原子对的距离计算:对于两个残基,计算它们内部每个原子与对方残基中所有原子的距离。这将产生一个原子对距离矩阵。
-
最小距离选取:从这个原子对距离矩阵中,选取距离最小的原子对,并将其作为两个残基之间的距离。这可以理解为这两个残基在空间上最靠近的部分。
这种方法的优点是,它能够反映残基之间最紧密的相互作用,而不需要计算所有原子对的平均距离或其他复杂的度量。这种最小距离的度量在很多分子动力学和蛋白质结构分析中都非常常见。
_unit_edges_from_block_edges 和 _construct_inter_edges的边
你提到的 _unit_edges_from_block_edges 和 _construct_inter_edges 之间的功能确实有一些重叠,但它们的目的和实现层次是不同的,主要区别在于以下几点:
1. 功能层次不同
-
_unit_edges_from_block_edges:这个函数的核心任务是将 残基级别的边(block-level edges)细化为 原子级别的边(unit-level edges)。换句话说,它将残基与残基之间的连接细化到原子与原子之间的具体连接。其作用是在残基之间定义的边的基础上,生成所有可能的原子对之间的边,或者选择最近的k个原子对。 -
_construct_inter_edges:这是一个更高层次的函数,负责在残基级别生成 不同残基之间的边(inter-residue edges)。它的输出是残基与残基之间的连接,通常是在蛋白质的结构层次上,用于构建残基之间的交互模型。
2. 功能目的不同
-
_unit_edges_from_block_edges:这个函数主要用于从更高层次的残基-残基边推导出原子-原子边,用来细化残基间的相互作用。这通常是为了在残基间连接的基础上,进一步考虑原子间的交互作用。 -
_construct_inter_edges:这个函数的目的是在残基之间生成交互连接,代表不同残基间的相互作用。这些残基可以通过多种方式生成边(如基于距离、K近邻等),用于在更高抽象层次上捕捉蛋白质结构中的残基间相互作用。
3. 实现方式的不同
-
_unit_edges_from_block_edges:该函数接收残基-残基的连接,并根据每个残基内部的原子生成更细化的原子-原子连接。这使得它可以在模型中引入原子级别的相互作用。 -
_construct_inter_edges:这个函数负责根据残基级别的输入(如残基坐标和类别),构造残基之间的边。这些边直接在残基级别上建模,不涉及残基内部的原子结构。
4. 是否涉及K近邻(KNN)
-
_unit_edges_from_block_edges:它可以选取残基间的最小原子对距离,并且可以通过k参数限制每个原子只连接最近的k个原子。 -
_construct_inter_edges:这个函数可能也会使用 KNN 来构造残基之间的连接,但它的连接是在残基层次上进行的,而不是细化到原子层次。
为什么不会完全重合?
- 不同的粒度:
_unit_edges_from_block_edges工作在 原子级别,它细化了已经存在的残基级别的连接。而_construct_inter_edges工作在 残基级别,直接生成残基之间的边,不涉及到原子之间的细节。 - 使用场景不同:如果你的模型需要考虑原子级别的交互,可能会先通过
_construct_inter_edges生成残基间的边,然后用_unit_edges_from_block_edges来细化成原子间的边。但如果你的模型只在残基级别工作,那么_construct_inter_edges就足够了。
总结
虽然这两个函数的功能看起来类似,但它们作用在不同的层次。_unit_edges_from_block_edges 是为了细化残基之间的边,生成原子级别的交互,而 _construct_inter_edges 直接工作在残基层次上,负责生成残基之间的交互连接。因此它们在不同的层次上各自发挥作用,并不会完全重合。、
我是说序号。因为残基和原子都是通过序号来表示。 序号的重合问题
明白了,你的意思是残基和原子的序号是否会重合。这涉及到在模型中如何标记和区分残基和原子的序号。通常,残基和原子的序号是不同的,模型在生成它们的边时也会用不同的方式处理它们的序号,以避免重合和混淆。
原子和残基序号的关系
-
残基序号 (
block_id):- 每个残基有一个唯一的序号,这个序号用于标识蛋白质链中的每个氨基酸残基。残基序号是全局的,即它在整个蛋白质链中是唯一的,表示的是蛋白质中的第几个氨基酸。
-
原子序号 (
unit_id):- 每个原子也有自己的序号,这个序号用于标识属于某个残基的具体原子。原子序号是基于残基的局部编号,比如第一个残基的所有原子有自己的序号,第二个残基的原子也有自己的序号。这些原子序号通常是连续的,因为残基之间有很多原子组成。
区分残基和原子序号
为了避免残基序号和原子序号重合,通常会采用两种方式来处理:
-
独立的序号体系:
- 残基和原子有各自独立的序号范围。残基序号 (
block_id) 是在残基级别定义的,表示蛋白质链中每个残基的位置;原子序号 (unit_id) 是在原子级别定义的,表示属于特定残基的原子。比如:block_id是每个残基的序号,可能是从 0 开始,直到蛋白质链的最后一个残基。unit_id是每个原子的序号,可能是针对所有原子的一个全局编号或者针对每个残基内部编号。
- 残基和原子有各自独立的序号范围。残基序号 (
-
通过
block_id和unit_id映射:- 为了避免重合和混淆,模型通常会通过
block_id和unit_id的映射关系来表示不同粒度的信息。也就是说:- 每个原子都有其对应的残基号
block_id,表示该原子属于哪个残基。 - 通过
unit_block_id可以将原子和它们所属的残基关联起来,从而清晰地区分残基和原子。
- 每个原子都有其对应的残基号
- 为了避免重合和混淆,模型通常会通过
具体处理方式
在你的代码中,block_id 表示残基的编号,unit_block_id 表示每个原子对应的残基编号。以下是代码中如何处理残基和原子序号的具体逻辑:
- 残基序号:
block_id是为每个残基分配的编号。 - 原子序号:通过
unit_block_id,可以知道每个原子属于哪个残基。这意味着,原子序号不会与残基序号直接重合,因为它们表示的层次不同。
例如,函数 _unit_edges_from_block_edges 通过 unit_block_id 确定每个原子与其所属残基之间的关系,同时通过 block_src_dst 定义了残基之间的边,进而细化成原子之间的边。原子之间的边依赖于这些残基之间的连接。
总结
原子和残基的序号是不会重合的。模型中通过 block_id(残基序号)和 unit_block_id(原子对应的残基序号)来区分它们。残基和原子各自有独立的序号,且原子序号通常是基于残基局部生成的,通过这种方式避免了在表示和计算过程中混淆原子和残基。
return (unit_src, unit_dst), (edge_id, unit_edge_src_start, unit_edge_src_id) 这是处理的函数,请帮我确认,是包含同一个残基中原子的边么,还是只包含跨block的边
从函数 _unit_edges_from_block_edges 的逻辑来看,它主要是基于 残基级别的边(block_src_dst)来生成 原子级别的边。因此,这个函数处理的边仅限于跨残基(block)的边,不包含同一个残基内部的原子之间的边。以下是详细的分析过程:
函数逻辑分解
-
输入参数:
unit_block_id: 每个原子所属残基的 ID。block_src_dst: 这是一个形状为[Eb, 2]的张量,表示残基级别的边。每对src, dst表示两个残基(block)之间的连接。
函数的核心任务是根据
block_src_dst(残基间的边)生成具体的原子级别的边。 -
残基到原子的映射:
block_n_units = scatter_sum(torch.ones_like(unit_block_id), unit_block_id) # [Nb], 每个残基中的原子数量 block_offsets = F.pad(torch.cumsum(block_n_units[:-1], dim=0), (1, 0), value=0) # [Nb]
计算范数
stable_norm 函数的作用不是求和,而是 计算向量的范数(norm),通常是 欧氏范数(即L2范数)。在这个特定的实现中,它调用了 PyTorch 的 torch.norm 函数。
torch.norm 的作用
torch.norm 用于计算输入张量的范数,通常是对张量的各个维度进行平方和开平方,即 L2范数。你可以通过传递不同的参数来计算不同类型的范数,比如 L1范数(绝对值和)或者 L∞范数(最大值范数)。
在没有传递其他参数时,torch.norm(input) 默认计算 L2范数,其计算公式为:
[
\text{L2 norm} = \sqrt{\sum_i x_i^2}
]
其中 ( x_i ) 是 input 张量中的每个元素。
代码解释
def stable_norm(input, *args, **kwargs):
return torch.norm(input, *args, **kwargs)
这段代码是调用 torch.norm 来计算输入 input 的范数。*args 和 **kwargs 允许你向 torch.norm 传递额外的参数,以控制如何计算范数。
后面的代码被定义但未实际执行,因为在执行 return torch.norm(input, *args, **kwargs) 后,函数就返回了结果,不会执行接下来的部分。如果将后续代码激活,它的目的是避免数值不稳定性(如在接近 0 的值上操作),但当前这个实现中,这些额外的步骤没有生效。
总结
这个 stable_norm 函数(在它当前的形式下)并不是求和,而是计算张量的范数(通常是L2范数)。
我可以理解成它是将映射的16拆分成4份么
是的,你可以理解成 将映射的 16 维向量拆分成 4 份,每份 4 维,并分别送入 4 个注意力头中处理。
具体来说:
- RBF 的输出维度是 16(
n_rbf = 16),这表示原子对的距离经过 RBF 函数映射到一个 16 维的特征空间。 - 由于模型使用了 多头注意力机制,有 4 个注意力头(
n_head = 4)。因此,将这 16 维的 RBF 特征 平均分配 到每个注意力头中,每个注意力头负责处理 4 维 的特征。
你可以将这个过程理解为:
- 16 维的向量 被均匀分割为 4 份,每份 4 维。
- 每个注意力头(4 个)分别处理这 4 维特征向量。
最终,每条边的 RBF 特征会被传递到 4 个注意力头中,每个头处理一个 4 维的特征。这就是为什么 D 的维度从 (471988, 16) 最终变成 (471988, 4, 4)。
总结:
是的,这个过程就是将原本的 16 维向量分成 4 份,每个注意力头处理一份 4 维的特征。
关于 D 经过 RBF 处理后维度变为 (471988, 4, 4) 中两个 4 的具体来源,主要涉及两个核心概念:
- 多头注意力机制中的头数量 (
n_head)。 - 径向基函数(Radial Basis Function, RBF)输出的维度 (
n_rbf)。
让我详细解释这两个 4 是如何产生的。
1. 第一个 4:注意力头的数量(n_head)
你代码中的注意力机制使用了 多头注意力,即 n_head 表示有多少个注意力头。根据你的代码,n_head 的值为 4:
H = H.view(H.shape[0], self.n_head, -1) # 多头注意力机制
这意味着你在计算注意力时,将特征划分为 4 个独立的部分(每个部分即为一个注意力头),每个头单独处理自己的特征信息。
因此,第一个 4 是由 n_head = 4 得来的,表示你有 4 个注意力头,每个头都有独立的计算分支。
2. 第二个 4:每个头上的 RBF 输出维度
你的代码使用了 径向基函数(RBF) 来处理原子对的距离信息。RBF 的作用是将一个标量(例如距离 D)映射到一个更高维的空间,捕捉更复杂的距离依赖关系。
- 在代码中,
n_rbf是 16,这意味着 RBF 将距离D映射到一个 16 维的向量空间。 - 然而,你的注意力机制是多头的,每个注意力头会处理一部分 RBF 输出。具体来说,RBF 输出的维度
n_rbf被均匀分配给每个头,即:
[
\text{每个头的 RBF 维度} = \frac{n_rbf}{n_head} = \frac{16}{4} = 4
]
因此,第二个4是由n_rbf / n_head = 16 / 4 = 4得到的,表示每个注意力头会接收一个 4 维的向量作为输入。
维度变化步骤
在 RBF 处理后的维度变化如下:
-
D的初始维度:D最初是计算出来的原子对的距离,形状为[471988, 1],表示 471988 条边的距离。 -
RBF 处理:
D = self.rbf(D.view(-1)).view(D.shape[0], n_channel, self.n_head, -1)self.rbf(D)会将D从[471988, 1]映射到[471988, 16],即每条边的距离被映射到一个 16 维的向量。- 然后,这个 16 维的向量会被重塑为
[471988, 1, 4, 4],其中:471988:是边的数量。1:表示通道数量(在这种情况下,可能表示这是单通道)。4:来自n_head = 4,表示 4 个注意力头。4:来自n_rbf / n_head = 4,表示每个注意力头的 RBF 维度。
-
调整维度:
D = D.transpose(1, 2).reshape(D.shape[0], self.n_head, -1) # [Eu, n_head, n_channel * n_rbf / n_head]D经过transpose和reshape操作后,将维度变为[471988, 4, 4],即:471988:是边的数量。- 第一个
4:表示 4 个注意力头(n_head)。 - 第二个
4:每个注意力头的 RBF 输出维度(n_rbf / n_head)。
总结
- 第一个
4:来源于 多头注意力机制中的头的数量(n_head = 4),表示每条边有 4 个注意力头分别处理。 - 第二个
4:来源于 每个头上的 RBF 输出维度(n_rbf / n_head = 16 / 4 = 4),表示每个注意力头处理 4 维的 RBF 特征。
因此,D 的最终维度 (471988, 4, 4) 表示:
- 471988 条边,
- 每条边有 4 个注意力头,
- 每个头在处理 4 维的 RBF 特征。
坐标恢复
在这个函数 recover_scale 中,对原子或残基的坐标 Z 进行了 重新缩放 和 中心化 处理。以下是对函数逐步解析的详细解释:
1. unit_batch_id = batch_id[block_id]
- 通过
block_id将batch_id映射到更细粒度的单元(unit)上,即每个原子对应的批次 ID(batch_id)。 block_id表示每个原子所属的残基,batch_id表示整个批次中每个残基的 ID。unit_batch_id是原子级别的批次 ID(即每个原子所属的批次 ID)。
2. Z_c = scatter_mean(Z, unit_batch_id, dim=0)
- 这里的
scatter_mean操作是对每个批次中的原子坐标Z进行求平均,以计算该批次内的所有原子在空间中的中心坐标(Z_c)。 Z是每个原子的三维坐标,unit_batch_id用来将原子归到不同的批次(batch_id)。- 结果
Z_c的形状为[bs, n_channel, 3],其中bs表示批次的数量,n_channel表示通道数(通常为 1),3表示坐标的三维(x, y, z)。
3. Z_c = Z_c[unit_batch_id]
- 这一行的操作是将每个原子对应的批次中心坐标
Z_c映射回每个原子。换句话说,对于每个原子,它的Z_c是该原子所在批次的中心坐标。 - 结果
Z_c的形状是[N, n_channel, 3],其中N是原子的总数。
4. Z_centered = Z - Z_c
- 这一行对每个原子的坐标
Z进行了 中心化 处理,即减去它所在批次的中心坐标Z_c,得到每个原子相对于批次中心的位置。 - 结果
Z_centered表示原子的位置相对于其批次中心的偏移量。
5. Z = Z_c + Z_centered / record_scale[unit_batch_id]
- 这里的操作是对 中心化的坐标
Z_centered进行 缩放恢复 操作:Z_centered / record_scale[unit_batch_id]是将中心化后的坐标按照record_scale进行缩放恢复。record_scale[unit_batch_id]表示对每个原子所在批次应用的缩放比例。
- 最后,通过
Z_c + Z_centered / record_scale[unit_batch_id],将缩放后的中心化坐标重新加回批次中心坐标,恢复到原始坐标系中的位置。
总结
这个函数的目的是:
- 中心化:将每个原子的坐标
Z中心化,即每个原子的位置相对于它所属的批次中心进行重新表示。 - 缩放恢复:通过
record_scale将中心化的坐标重新缩放,恢复到原始的坐标比例。 - 坐标复原:将中心化且经过缩放的坐标加回原始批次中心,得到重新缩放并复原的坐标
Z。
这个过程可能用于在特定情况下对坐标进行归一化和缩放操作,最后再将坐标恢复到某种尺度上。
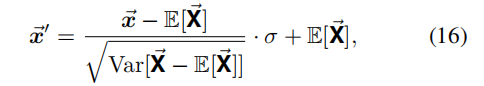
是类似于公式16这种缩放么
是的,recover_scale 函数的操作与公式 (16) 类似。公式 (16) 描述了一个常见的缩放和归一化过程,其中:
- 中心化:通过减去均值((\mathbb{E}[\vec{X}])),将数据移到以零为中心的位置。
- 标准化:除以数据的标准差 (\sqrt{\text{Var}[\vec{X} - \mathbb{E}[\vec{X}]]}),以调整数据的方差(或幅度)。
- 缩放恢复:乘以一个缩放因子 (\sigma),将标准化后的数据恢复到特定的尺度。
- 均值恢复:最后再加回均值 (\mathbb{E}[\vec{X}]),将数据重新定位到原来的中心位置。
公式 (16) 实际上描述的是对一个向量 (\vec{x}) 进行标准化(减去均值并除以标准差),然后再根据标准差 (\sigma) 和均值 (\mathbb{E}[\vec{X}]) 将其恢复到一个特定的尺度。
在你的代码中,recover_scale 函数执行的过程也很类似:
- 中心化:通过减去批次的中心坐标
Z_c,将坐标Z中心化。 - 缩放:通过除以
record_scale进行缩放处理,类似于公式中的标准化步骤。 - 均值恢复:加回批次的中心坐标
Z_c,类似于公式中将均值恢复的步骤。
因此,recover_scale 函数的逻辑基本上与这个公式的缩放和恢复过程是一致的。
边的映射
在这段代码中,edge_id 表示 每个原子对(unit pair)所属的边的编号。具体来说,edge_id 是用来追踪每个原子对(unit_src, unit_dst)属于哪个 block-level 的边。
为了更清楚地解释 edge_id 的含义,以下是其生成和使用的过程:
1. block_src_dst:block-level 的边
block_src_dst是输入的 block-level 的边,表示哪些残基(block)之间有连接。它的形状是[Eb, 2],其中Eb是 block 之间的边的数量,每个元素是一个长度为 2 的向量,表示两个 block 之间有边。
2. edge_id 的生成
- 在代码中,
edge_id用来记录 unit-level 边 对应的 block-level 边。具体来说,对于每对原子(unit)之间的连接,edge_id记录了这对原子所属的 block-level 边的索引。
具体的计算过程如下:
edge_id = torch.zeros(edge_n_pairs.sum(), dtype=torch.long, device=edge_n_pairs.device) # [Eu], which edge each unit pair belongs to
edge_id是一个长度为Eu的张量,Eu表示所有 unit 级别边的数量。- 这个张量用于追踪每对原子对(unit pair)属于哪个 block-level 的边。比如,如果一个 block-level 边
block_src_dst[i]包含 4 对原子对,那么这 4 对原子对的edge_id都会设置为i,表明它们属于同一个 block-level 的边。
3. edge_id 的生成过程细节
通过以下步骤生成:
edge_start_index = torch.cumsum(edge_n_pairs, dim=0)[:-1] # [Eb - 1], start index of each edge (without the first edge as it starts with 0) in unit_src_dst
edge_id[edge_start_index] = 1
edge_id = torch.cumsum(edge_id, dim=0) # [Eu], which edge each unit pair belongs to, start from 0, end with Eb - 1
edge_start_index是每个 block-level 边的 unit 对的起始位置。edge_id[edge_start_index] = 1将每个 block-level 边的起始位置标记为 1。torch.cumsum(edge_id, dim=0)使用累积求和的方式,将edge_id中的 1 扩展为对每个原子对所属的 block-level 边进行编号。
4. edge_id 的作用
- 这个
edge_id主要是用于追踪每对原子对(unit pair)属于哪个 block-level 的边。在后续代码中,它会用于将特征从原子对的层面聚合到 block-level 的层面。 - 当你想处理某个 block-level 边时,你可以通过
edge_id找到所有与该 block-level 边相关的原子对。
总结
edge_id 记录的是 每个原子对(unit pair)属于哪个 block-level 的边。通过这个编号,你可以知道某个原子对是通过哪个 block-level 边连接起来的,并且可以在后续处理中根据这个编号对边进行聚合和特征处理。
原子-原子映射到残基-残基
是的,根据当前代码逻辑,edge_id 只会对应 block-level(残基级别) 的 跨残基的连接,即 inter_edges 类型的连接,而不会对应 同一残基内部的连接(即 intra_edges)。
让我们进一步解释为什么 edge_id 主要对应的是 跨残基的连接:
1. block_src_dst 描述的是跨残基的连接
block_src_dst是一个[Eb, 2]的张量,表示 残基之间 的连接(即inter_edges)。每个元素是两个残基的 ID,表示这两个残基之间有连接。- 基于
block_src_dst生成的edge_id,用于映射从这些跨残基的边生成的 unit-level(原子级别) 边对。
2. edge_id 追踪的是跨残基的边
edge_id的生成逻辑通过跨残基的连接(即block_src_dst)确定。edge_id的目的是追踪每对原子(unit pair)属于哪个跨残基的连接。- 由于
edge_id是根据block_src_dst构建的,而block_src_dst只描述 残基之间 的连接,因此edge_id只会映射到 跨残基的边,即inter_edges。
3. 不处理 intra_edges(残基内部的连接)
- 如果需要处理 残基内部的连接(即
intra_edges),则需要额外的逻辑。当前的edge_id和block_src_dst逻辑只处理 残基之间的连接,即 跨残基 的连接。 - 残基内部的连接可以通过不同的方式生成,比如基于化学键或空间距离的原子对,但这在
block_src_dst中并没有涉及。
总结
- **
edge_id只会对应 block-level 中的 跨残基的连接,即 残基与残基之间的边(inter_edges)。 - 代码当前的逻辑不处理 残基内部的连接(
intra_edges),这些连接如果需要,则需要通过额外的逻辑处理。
如果你想在 edge_id 中包含 残基内部的连接,需要引入相应的逻辑来生成并处理这些 intra_edges,然后将它们与 inter_edges 一起处理。



 浙公网安备 33010602011771号
浙公网安备 33010602011771号