易基因:河大蒋建军教授BS-seq揭示底物特异性和蛋白稳定性促进植物特异性DNA甲基转移酶分化|Sci Adv
大家好,这里是专注表观组学十余年,领跑多组学科研服务的易基因。
DNA甲基化是一种重要的表观遗传机制,对转座子沉默和基因组完整性至关重要。在进化过程中,DNA甲基化底物在不同生物界中发生多样化。在植物中,染色体甲基化酶3(CMT3)和CMT2分别介导CHG和CHH位点的甲基化。然而,这两种甲基转移酶在进化过程中如何分化其底物特异性仍然未知。
近日,河南大学蒋建军教授为第一作者,美国圣路易斯华盛顿大学钟雪花教授和加州大学河滨分校宋吉奎教授为共同通讯在Science子刊《Science Advances》上发表了题为“Substrate specificity and protein stability drive the divergence of plant-specific DNA methyltransferases”的研究论文,揭示了底物特异性和蛋白质稳定性如何驱动植物特异性DNA甲基转移酶分化,特别是CMT2和CMT3在进化过程中的底物特异性分化,阐明了CMT2和CMT3产生功能分化的表观遗传调控机制。

标题:Substrate specificity and protein stability drive the divergence of plant-specific DNA methyltransferases(底物特异性和蛋白质稳定性促进植物特异性DNA甲基转移酶分化)
发表时间:2024年11月6日
发表期刊:Science Advances
影响因子:IF 11.7 / 1区
应用技术:BS-seq、RNA-seq(易基因金牌技术)
本研究进行了全面的结构、功能和进化研究,分析了CMT2和CMT3分化背后的分子机制。研究首先揭示了对CMT3识别CHG至关重要的精氨酸残基(R745)在CMT2中表现出很大的变异,解释了其失去CHG特异性的原因。在拟南芥中,将CMT2中相应的残基突变为精氨酸(V1200R)获得了CHG甲基化活性,并在cmt2cmt3突变体中重新沉默一组转座元件(TEs),发挥了类似CMT3功能。CMT3有一个短的N末端,CMT2却包含一个长而无序的N末端,这是许多植物物种的共同特征。这个长的N末端调控CMT2稳定性,并介导热诱导的CMT2降解。此外由于自然界中CMT2的N端存在多种变异,CMT2的N端更具可塑性,能够高度容忍各种自然变异。总的来说,本研究揭示了植物种染色体甲基化酶靶向特定环境DNA甲基化的分化机制,并为植物中DNA甲基化的功能和进化提供了新的视角。
研究方法:
- 结构和功能研究:通过比较CMT3和CMT2的关键残基,构建了CMT2与DNA复合物的结构模型,并进行体外甲基化实验。
- 系统发育分析:通过系统发育分析探讨CMT2和CMT3的进化关系。
- 基因编辑:通过在拟南芥中引入V1200R突变,研究者们增强了CMT2的CHG甲基化活性。
- DNA甲基化分析:利用全基因组亚硫酸盐测序(WGBS-seq)技术分析CMT2和CMT2V1200R对全基因组甲基化水平的影响。
- 转录组测序:通过RNA-seq分析CMT2V1200R介导CHG甲基化对基因表达的影响。
- 自然变异分析:分析自然条件下的CMT2变异,以及这些变异对DNA甲基化和植物适应性影响。
结果图形
(1)CMT2起源于开花植物中CMT3的复制,CMT2V1200R在体外和体内诱导CHG甲基化增加
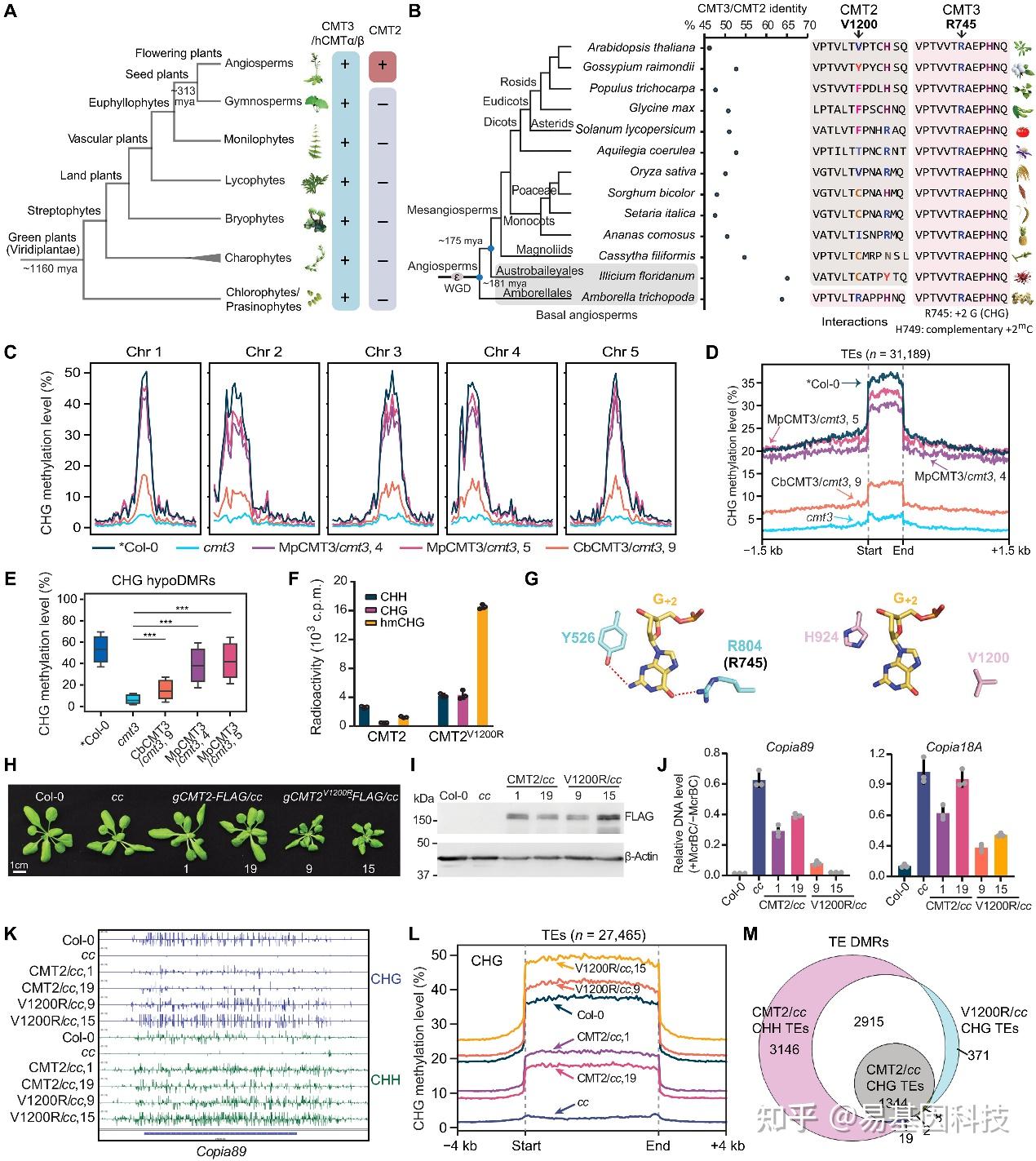
图1:精氨酸残基丢失使CMT2具有与CMT3不同的底物偏好。
A. 绿色植物(Viridiplantae)中CMT3(或其近缘同源物hCMTα/β)和CMT2的系统发育树。
B. CMT2和CMT3之间的蛋白质序列同一性(左图)以及在开花植物中与+2鸟嘌呤(G+2)接触的残基和CMT2中的相应残基(右图)。
C. 在cmt3突变背景下,M. polymorpha CMT3(MpCMT3)和C. braunii CMT3(CbCMT3)的CHG甲基化水平Meta图。
D. 拟南芥中所有转座元件(TEs)上的平均CHG甲基化水平Meta图。
E. CHG低甲基化区域(hypoDMRs)上的CHG甲基化水平箱线图。
F. CMT2和CMT2V1200R对CHH、CHG或半甲基化CHG(hmCHG)DNA底物的体外DNA甲基转移酶检测。
G. 玉米ZMET2-DNA复合物结构(蛋白质数据库7UBU)显示ZMET2 Y526和R804与CHG环境中目标胞嘧啶+2位点(G+2)鸟嘌呤之间的相互作用。
H. 两个独立的转基因CMT2和CMT2V1200R植物在cmt2cmt3(cc)突变背景下的表型图像。
I. 免疫印迹图显示(H)中植物的CMT2和CMT2V1200R蛋白水平。
J. McrBC-qPCR分析两个CMT2靶向TEs Copia89和Copia18A的DNA甲基化水平。
K. BS-seq中Copia89的CHG和CHH甲基化水平的基因组浏览器视图。
L. 拟南芥中所有TEs上的平均CHG甲基化水平Meta图。
M. 与Col-0比较时鉴定DMRs的重叠情况维恩图。
(2)CMT2V1200R介导的CHG甲基化抑制TE
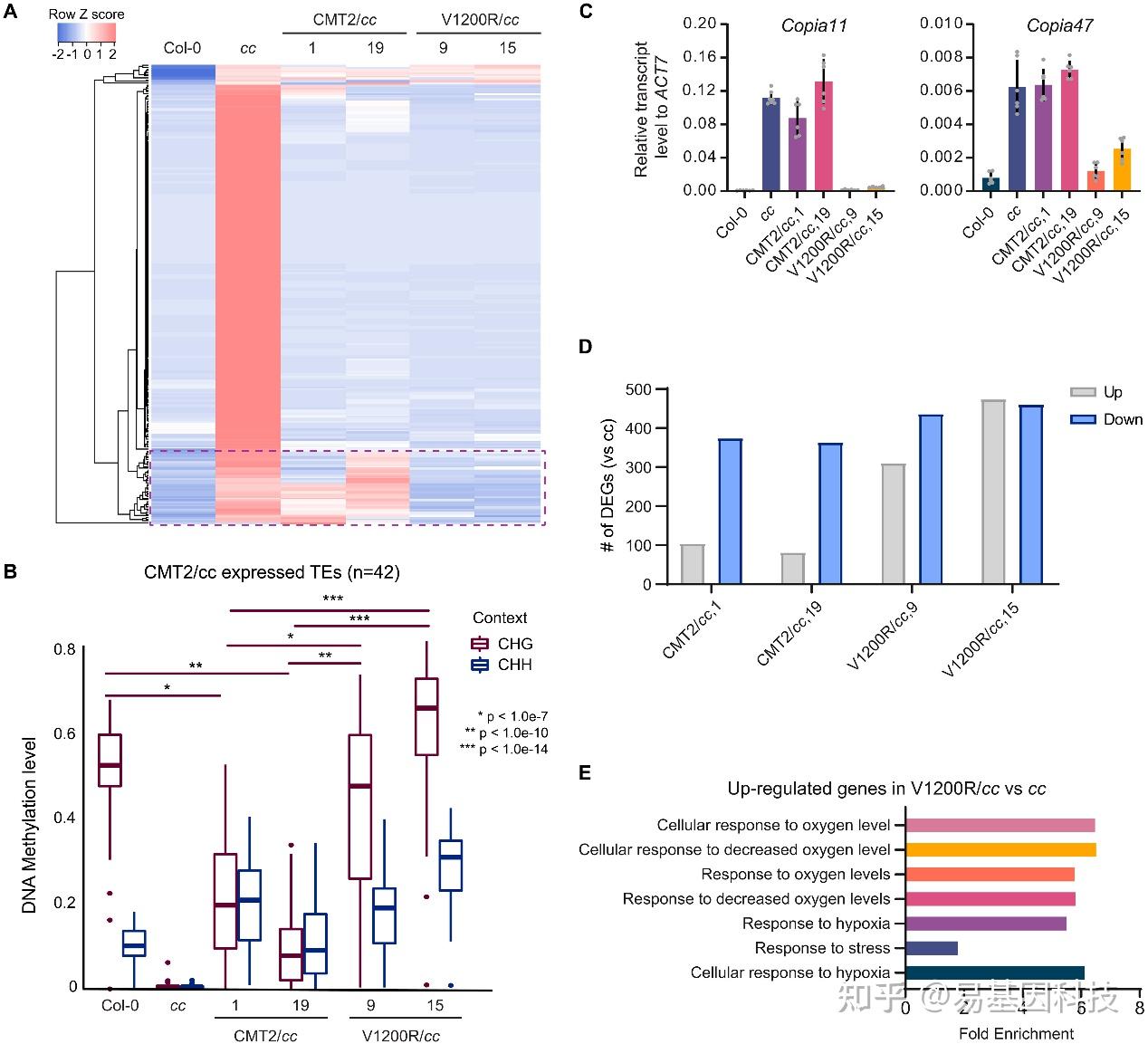
图2. CMT2V1200R抑制CMT2和CMT3靶向的转座元件(TEs)。
A. 两个独立的CMT2/cc和V1200R/cc转基因系中上调TEs的表达水平热图。cc代表cmt2cmt3。虚线框表示被CMT2V1200R重新沉默的TEs。
B. CMT2/cc中仍然表达的TEs的CHG和CHH DNA甲基化水平。
C. 在CMT2/cc和V1200R/cc转基因植物中,通过RT-qPCR检测Copia11和Copia47的相对转录水平。
D. CMT2/cc和V1200R/cc中差异表达基因(DEGs)数量。
E. V1200R/cc中上调基因的基因本体(GO)分析。
(3)CMT2包含一个对核定位很重要的长N末端
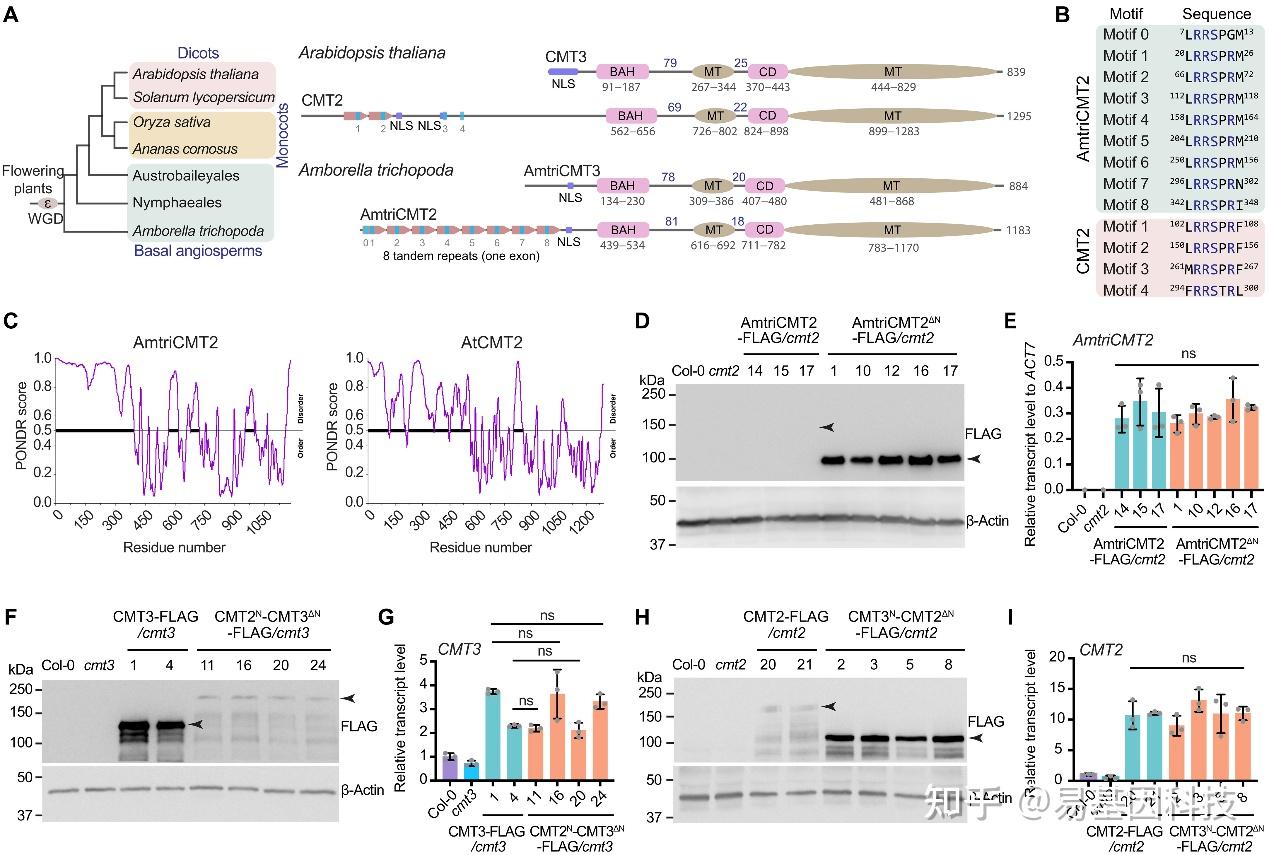
图3. CMT2的长N端无序,且调控蛋白水平。
A. 拟南芥(A. thaliana,双子叶植物)和A.trichopoda(基部被子植物)的CMT2和CMT3域结构图表。蓝色条表示串联重复。MT,甲基转移酶域;CD,染色质域。线上的数字表示域之间的氨基酸数量。WGD,全基因组复制。
B. A.trichopoda和A. thaliana CMT2 N端保守RRS motif比对。
C. A.trichopoda和A. thaliana CMT2的固有无序得分图,由PONDR评分。
D. 在A. thaliana cmt2突变背景下表达全长A.trichopoda CMT2(AmtriCMT2)和N端截短的A.trichopoda CMT2(AmtriCMT2ΔN)的蛋白水平免疫印迹。
E. RT-qPCR显示AmtriCMT2和AmtriCMT2ΔN转基因系中AmtriCMT2转录水平。
F. 野生型(CMT3)和N端转换的CMT3(CMT2N-CMT3ΔN)转基因系在cmt3突变体中的蛋白水平免疫印迹。
G. RT-qPCR显示CMT3和CMT2N-CMT3ΔN转基因植物中CMT3转录水平。
H. 野生型(CMT2)和N端转换的CMT2(CMT3N-CMT2ΔN)转基因系在A. thaliana cmt2突变体中的蛋白水平免疫印迹。
I. RT-qPCR显示CMT2和CMT3N-CMT2ΔN转基因系中CMT2转录水平。
转录水平首先归一化到Col-0中的CMT2水平,然后归一化到Col-0中的ACT7水平。ns,无显著性差异。
(4)CMT2的N末端调控蛋白质稳定性
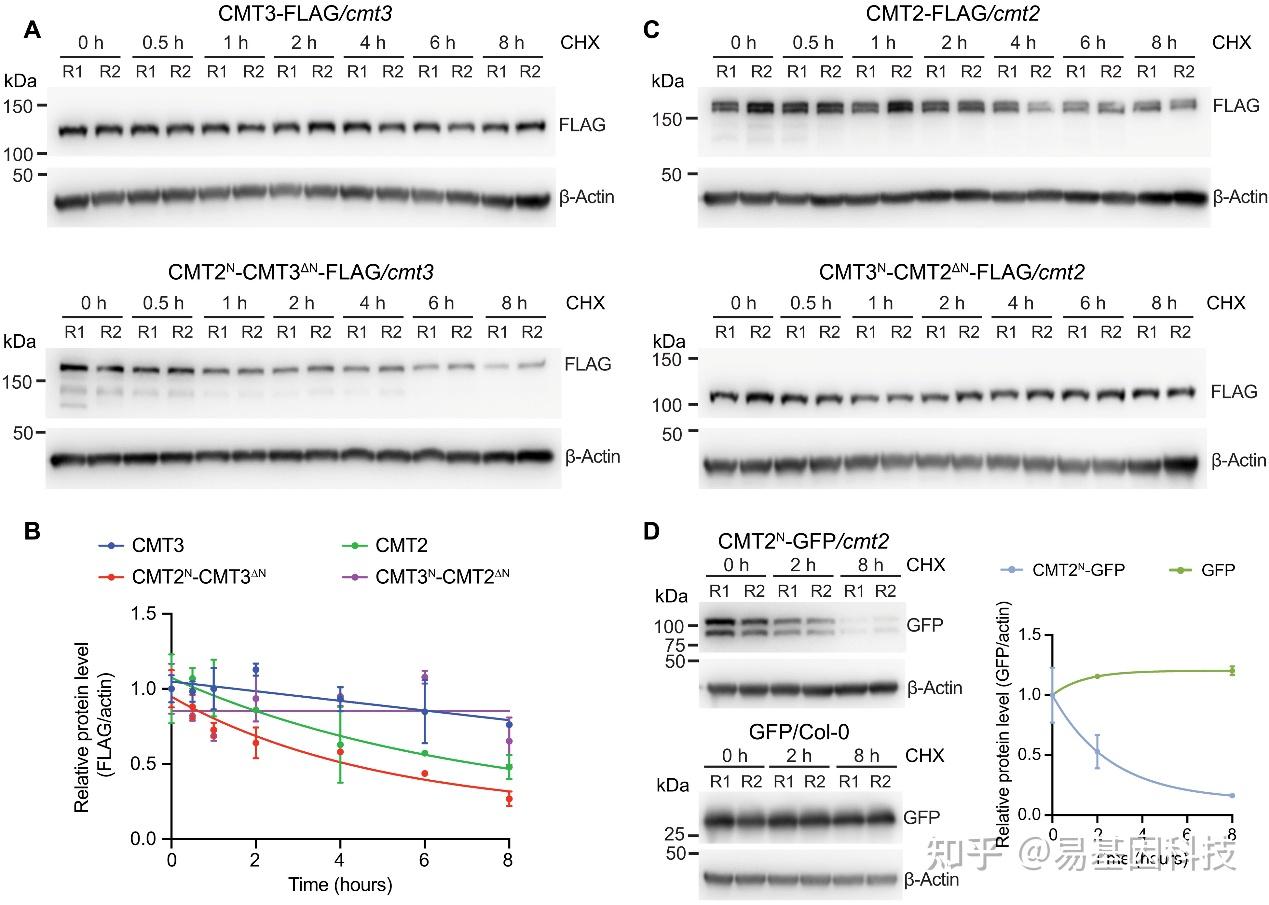
图4. CMT2的N末端调控其蛋白稳定性。
A. 用500 μM环己酰亚胺(CHX)处理指定时间后CMT3(顶部)和CMT2N-CMT3ΔN(底部)的水平免疫印迹图。使用7日龄幼苗。肌动蛋白作为对照。R1和R2代表两个生物学重复。
B. 相对于肌动蛋白归一化的蛋白水平定量。
C. 免疫印迹图显示用500 μM环己酰亚胺处理指定时间后CMT2(顶部)和CMT3N-CMT2ΔN(底部)的水平。
D. 免疫印迹图显示用500 μM环己酰亚胺处理后CMT2N-GFP(顶部)和仅GFP(底部)的水平。右侧是通过GFP信号相对于肌动蛋白标准化的蛋白水平定量。
(5)N末端介导热诱导的CMT2降解
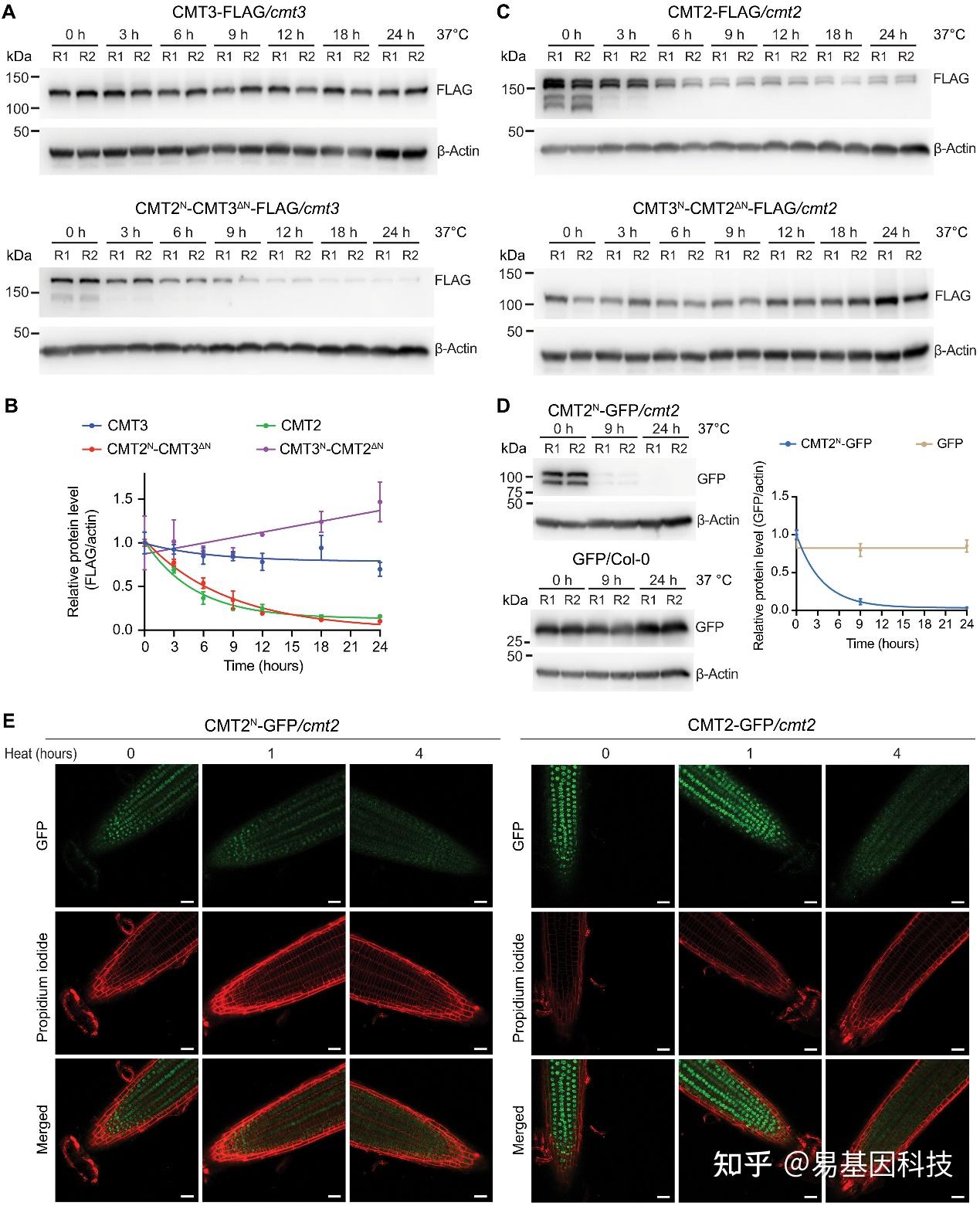
图5. 长N末端介导热诱导的CMT2蛋白降解。
A. 免疫印迹图显示在37°C热处理指定时间后CMT3(顶部)和CMT2N-CMT3ΔN(底部)的水平。使用10日龄幼苗。
B. 相对于肌动蛋白归一化的蛋白水平定量。
C. 免疫印迹图显示在37°C热处理指定时间后CMT2(顶部)和CMT3N-CMT2ΔN(底部)的水平。
D. 免疫印迹图显示在37°C热处理后CMT2N-GFP(顶部)和仅GFP(底部)的水平。
E. 共聚焦显微镜图像显示在37°C热处理指定时间后,拟南芥根尖中CMT2N-GFP和CMT2-GFP的定位和强度。
(6)CMT2自然变异表现出对环境胁迫的耐受性
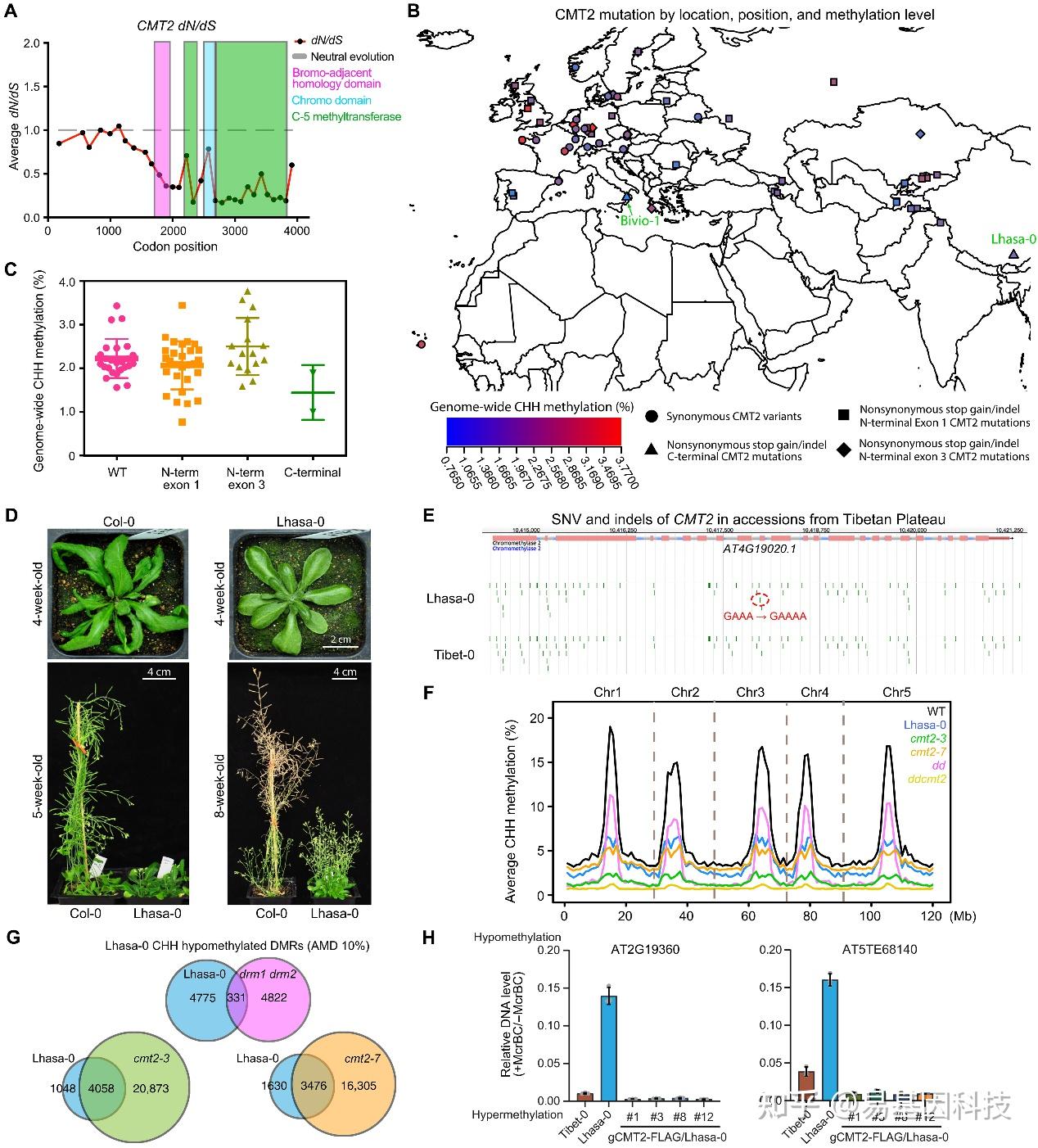
图6. 拟南芥中CMT2的自然变异。
A. 拟南芥近缘种CMT2基因的dN/dS绘图。
B. 不同CMT2基因突变的品系全基因组CHH甲基化水平。
C. 野生型(WT;n=25)、N端(第1外显子(n=31)和第3外显子(n=16)的AT4G19020.1)和C端(n=2)CMT2突变的CHH甲基化点图。
D. 拟南芥生态型Col-0和Lhasa-0的表型比较。
E. 来自青藏高原的自然品系Lhasa-0和Tibet-0的基因组浏览器视图,显示CMT2外显子中的单核苷酸变异(SNV)和插入。
F. 与野生型Col-0和CHH DNA甲基转移酶突变体相比,Lhasa-0生态型在拟南芥染色体上的平均水平CHH甲基化。
G. Lhasa-0与CHH DNA甲基转移酶突变体之间低甲基化差异甲基化区域(DMRs)的重叠。
H. McrBC-qPCR分析显示在两个代表性位点的DNA甲基化互补。含有CMT2基因组序列(gCMT2)的转基因系可以挽救Lhasa-0中丢失的CHH甲基化。Tibet-0用作对照。AMD,绝对甲基化差异。
易小结
本研究通过WGBS+RNA-seq等分析揭示了植物中CMTs的分化机制,为理解DNA甲基化在植物进化和功能中的作用提供了重要见解。
研究亮点
1) CMT2的起源和进化:研究揭示了CMT2是如何从CMT3分化而来,以及这一分化如何影响了植物的DNA甲基化模式。
2) V1200R突变的功能研究:通过V1200R突变,研究者们成功地改变了CMT2的底物特异性,这一发现对于理解酶活性的调控具有重要意义。
3) CMT2 N端的稳定性研究:研究者们发现CMT2的长N末端在热应激下对其稳定性至关重要,这一发现为理解环境因素如何影响表观遗传机制提供了新的视角。
4) 自然变异与环境适应性:通过分析自然条件下CMT2的变异,研究者们揭示了这些变异如何影响植物对环境应激的适应性,这对于理解植物进化和育种具有重要意义。
这项研究不仅增进了对植物DNA甲基化机制的理解,还为农业育种和作物改良提供了潜在的分子靶标。
关于易基因全基因组重亚硫酸盐测序(WGBS)
全基因组重亚硫酸盐甲基化测序(WGBS)可以在全基因组范围内精确的检测所有单个胞嘧啶碱基(C碱基)的甲基化水平,是DNA甲基化研究的金标准。WGBS能为基因组DNA甲基化时空特异性修饰的研究提供重要技术支持,能广泛应用在个体发育、衰老和疾病等生命过程的机制研究中,也是各物种甲基化图谱研究的首选方法。
易基因全基因组甲基化测序技术通过T4-DNA连接酶,在超声波打断基因组DNA片段的两端连接接头序列,连接产物通过重亚硫酸盐处理将未甲基化修饰的胞嘧啶C转变为尿嘧啶U,进而通过接头序列介导的 PCR 技术将尿嘧啶U转变为胸腺嘧啶T。
应用方向:
WGBS广泛用于各种物种,要求全基因组扫描(不错过关键位点)
- 全基因组甲基化图谱课题
- 标志物筛选课题
- 小规模研究课题
技术优势:
- 应用范围广:适用于所有参考基因组已知物种的甲基化研究;
- 全基因组覆盖:最大限度地获取完整的全基因组甲基化信息,精确绘制甲基化图谱;
- 单碱基分辨率:可精确分析每一个C碱基的甲基化状态。

易基因提供全面的表观基因组学(DNA甲基化、DNA羟甲基化)和表观转录组学(m6A、m5C、m1A、m7G、ac4C)、染色质结构与功能组学技术方案(ChIP-seq、ATAC-seq),详询易基因:0755-28317900。
参考文献:
Jiang J, Gwee J, Fang J, Leichter SM, Sanders D, Ji X, Song J, Zhong X. Substrate specificity and protein stability drive the divergence of plant-specific DNA methyltransferases. Sci Adv. 2024 Nov 8;10(45):eadr2222. doi: 10.1126/sciadv.adr2222. PubMed PMID: 39504374.
相关阅读:
WGBS+RNA-seq揭示松材线虫JIII阶段形成过程中的DNA甲基化差异
WGBS+RNA-seq揭示黄瓜作物的“源-库”关系受DNA甲基化调控


