易基因:朱健康团队WGBS揭示主动DNA去甲基化缺失植物的DNA甲基化跨代增加|PNAS
大家好,这里是专注表观组学十余年,领跑多组学科研服务的易基因。
DNA甲基化是一种在植物和哺乳动物中保守的表观遗传标记,对生长、发育、疾病和寿命具有重要作用。植物和动物基因组中DNA甲基化的主动获得或丢失,以及DNA甲基化水平变化可能导致减数分裂稳定的表观遗传变异,这些变异产生可遗传的表型多样性。尽管对跨代表观遗传已经了解很多,但目前尚不清楚跨代表观遗传稳定性是否可能受到哪些细胞因子的调控。
2024年5月20日,,南方科技大学朱健康院士团队在《PNAS》 (Proceedings of the National Academy of Sciences of the United States of America)杂志上发表题为“Transgenerational increases in DNA methylation in Arabidopsis plants defective in active DNA demethylation”的研究成果,该研究以模式生物拟南芥为研究对象,通过WGBS等分析揭示了DNA去甲基化酶ROS1突变导致基因组水平上DNA甲基化水平逐代升高。研究表明了ROS1在阻止随机形成的DNA甲基化的跨代遗传、维持表观基因组稳定中发挥重要监控功能。本研究对理解基因组DNA甲基化的进化以及生存环境如何通过表观遗传影响后代具有重要意义。

标题:Transgenerational increases in DNA methylation in Arabidopsis plants defective in active DNA demethylation(在主动DNA去甲基化缺陷的拟南芥植物中,DNA甲基化的跨代增加)
杂志:PNAS
影响因子:IF 9.4 /Q1
技术平台:WGBS(易基因优势技术)
研究摘要:
本研究分析了从单粒种子后代连续繁殖10代(G1-G10)的野生型和ros1突变体拟南芥植物在正常和盐胁迫两种生长条件下主动发生的DNA甲基化跨代变化。分析结果揭示了主动DNA去甲基化功能缺失的ros1突变体显示出增加的跨代表观遗传突变率。与野生型相比,ros1突变体在个别胞嘧啶上获得的甲基化比丢失的甲基化要多,而野生型则在主动获得和丢失的甲基化胞嘧啶数量上相似。同样的,跨代表观遗传差异甲基化区域在ros1突变体中也表现出高甲基化。本研究结果揭示了ROS1 DNA去甲基化酶对跨代表观遗传稳定性的遗传贡献,并表明ROS1可能具有防止跨代DNA甲基化增加的意外监控功能。
研究方法:
样本选择:Col-0 野生型和 ros1 突变体,正常和盐胁迫条件下从单粒拟南芥种子连续繁殖10代(G1-G10),每一代创建5个独立株系。通过WGBS分析G1-G10的差异甲基化位点(DMCs)和差异甲基化区域(DMRs)。
干货分享 》》易基因: DNA甲基化差异水平分析中的DMC、DMR、DMG鉴定
结果图形:
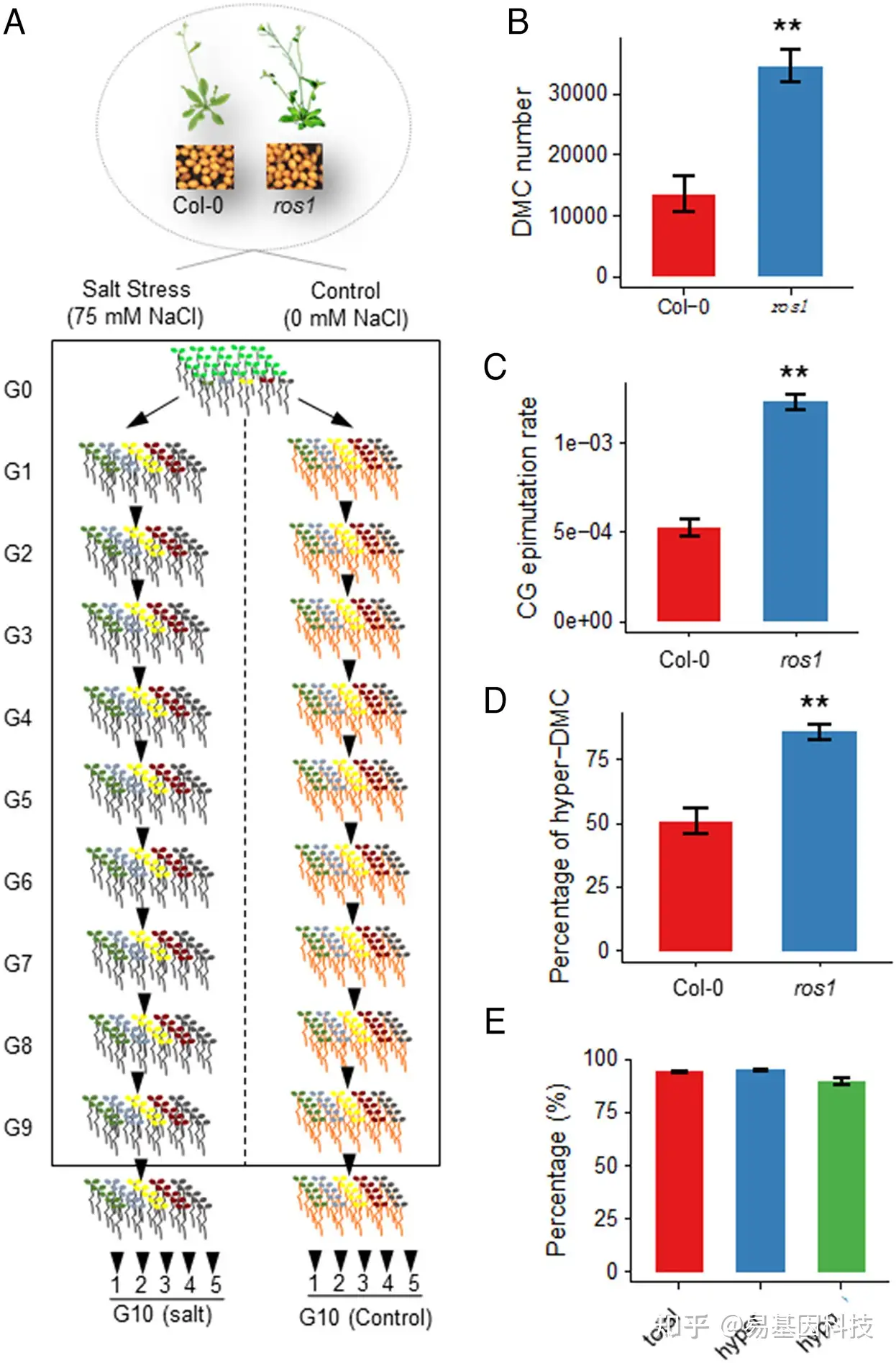
图1:研究的实验设计与跨代差异甲基化位点(DMC)分析:
- 实验设计:从单一祖先植物收获种子,并在MS培养基上发芽。将10天大的幼苗移植到土壤中。在blotting开始时,从五株标记的植物(由五株不同颜色的幼苗表示)收集莲座叶(rosette leaf),并将其汇总为G1样本。盐处理组和对照组(CK)分别从G1代衍生出五条独立系。盐处理组在每一代持续用75 mM NaCl溶液灌溉。CK组用0 mM NaCl水灌溉。G10(无盐)和盐处理-G10样本是在blotting开始时从盐处理组和CK组的五条独立系的第十代收集的莲座叶。
- 在正常生长条件下,鉴定出的G1与后代(G10)之间跨代DMCs数量。
- 每个基因型的CG表观遗传突变率。
- 每个基因型中跨代高甲基化差异位点hyper-DMCs百分比。
- 不位于ros1 G1高甲基化差异区域(hyper DMRs)中的跨代甲基化差异位点DMCs(高甲基化、低甲基化和结合)的比例。
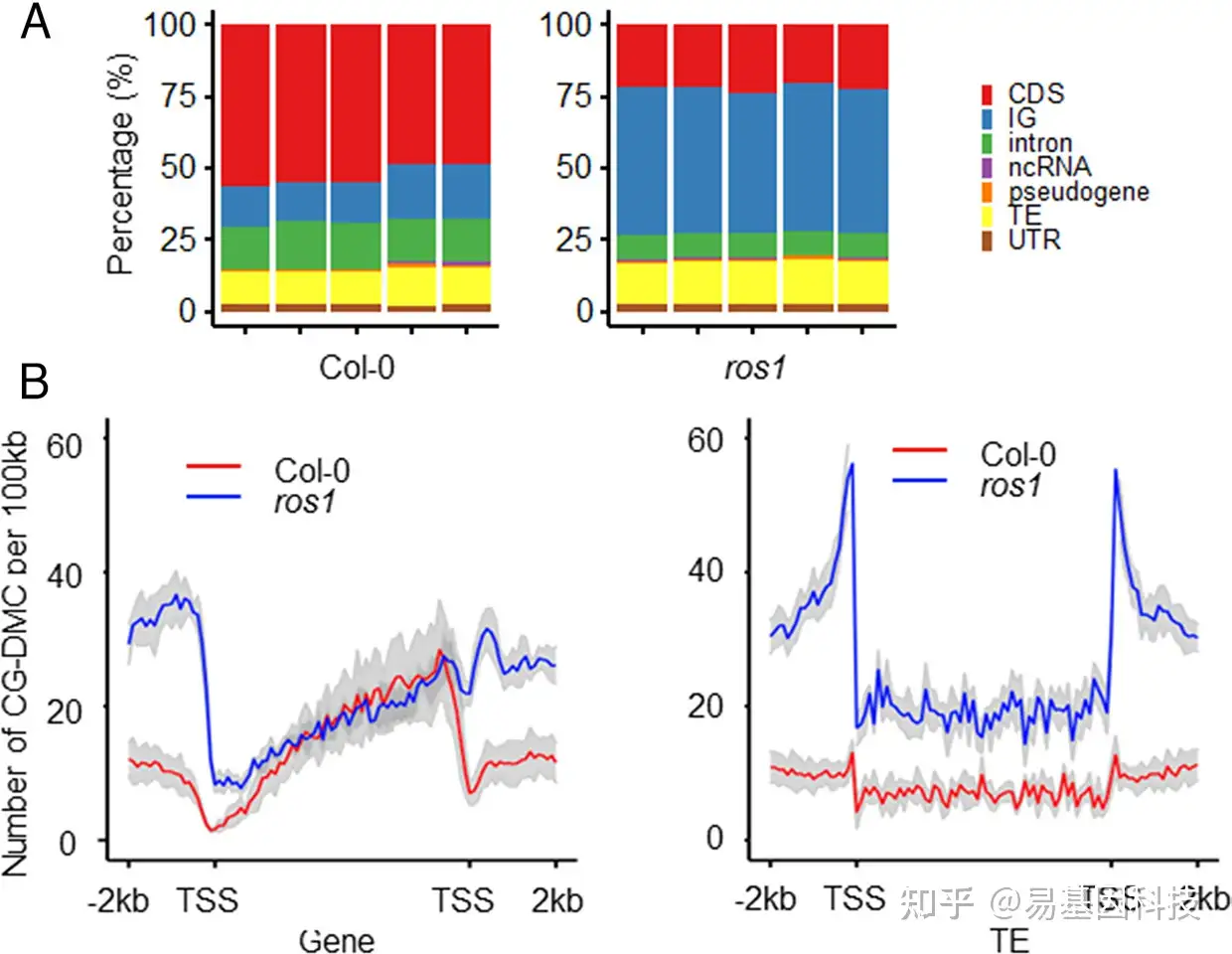
图2:在正常生长条件下植物跨代CG-DMCs表征
- 基于基因组特征对跨代表观遗传CG-DMCs进行注释。
- 跨代表观遗传CG-DMCs在基因和转座元件(TEs)上的分布。
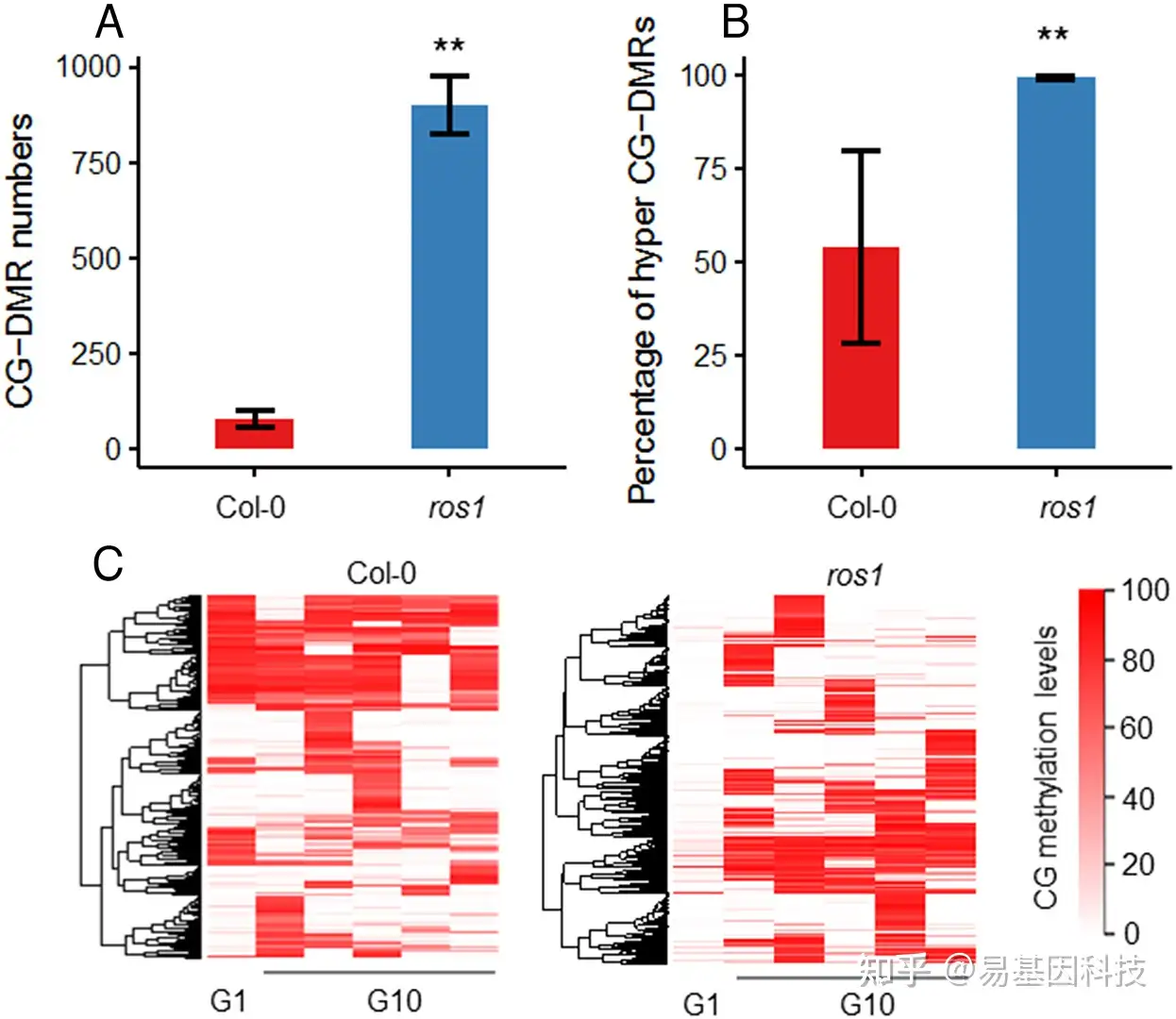
图3:跨代表观遗传CG-DMRs的统计数据和热图:
- Col-0和ros1中跨代表观遗传CG-DMRs数量。
- Col-0和ros1中高甲基化跨代表观遗传CG-DMRs百分比。
- Col-0和ros1中跨代表观遗传CG-DMRs DNA甲基化模式热图。
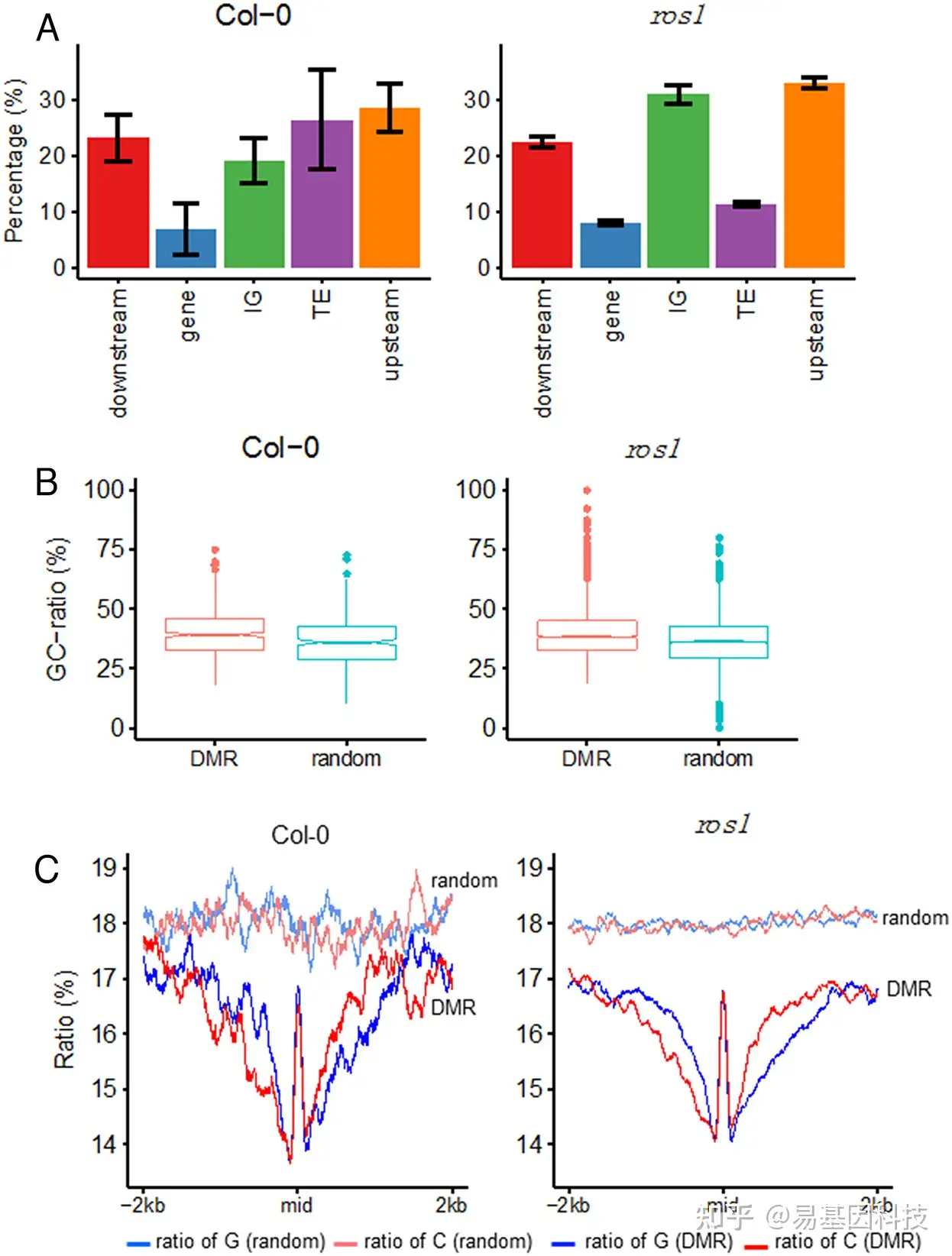
图4:在正常生长条件下植物跨代表观遗传CG-DMRs表征
- 基于基因组特征对跨代表观遗传CG-DMRs进行注释。
- Col-0和ros1中跨代表观遗传CG-DMRs与随机选择区域的GC含量比率。每个基因型的五个CG-DMRs合并分析。随机选择区域与合并的CG-DMRs具有相同的长度分布。
- Col-0和ros1中跨代表观遗传CG-DMRs中G和C的比例。使用随机选择的区域作为对照。所使用的区域是CG-DMR中点周围±2kb侧翼区域。
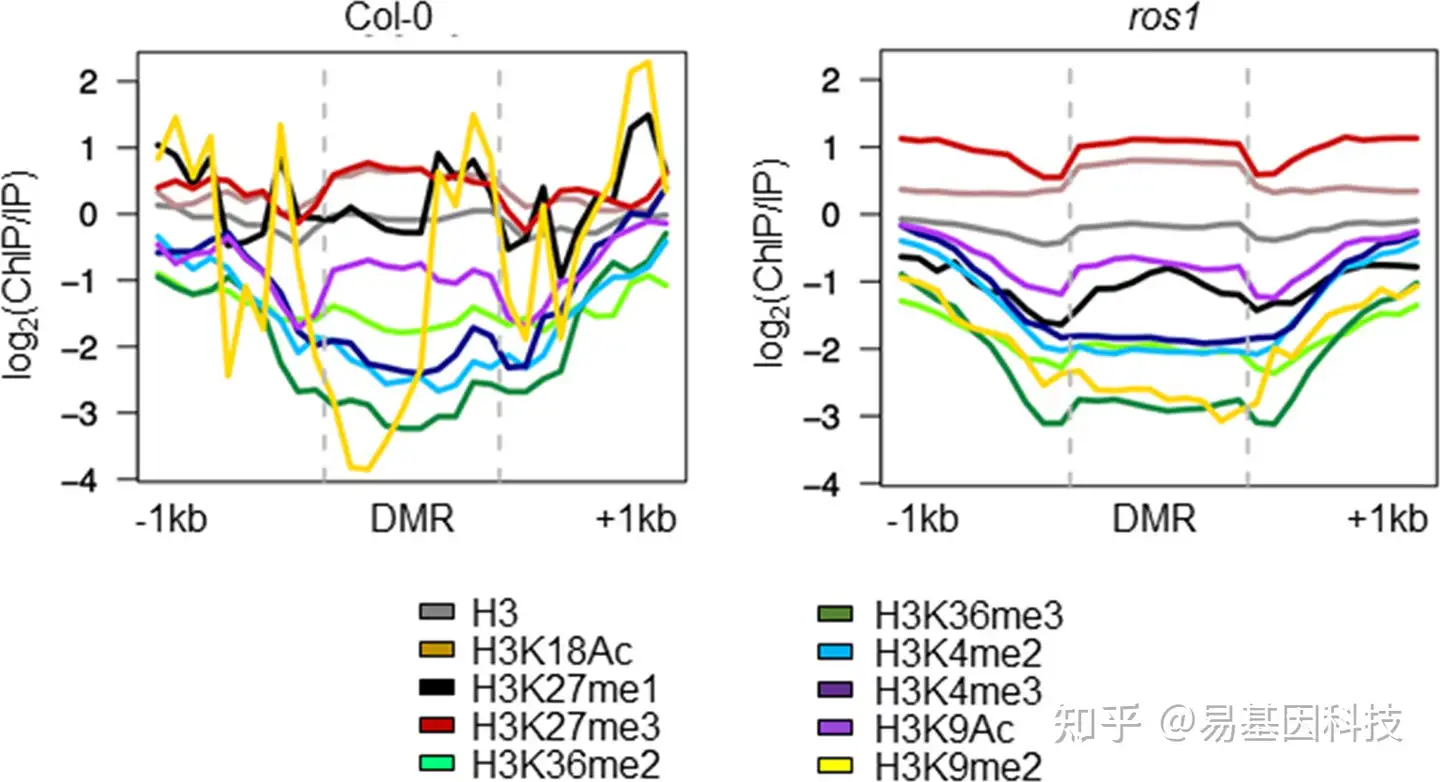
图5:在正常生长条件下,Col-0和ros1中跨代表观遗传CG-DMR周围不同组蛋白修饰信号分布。
关于易基因全基因组重亚硫酸盐测序(WGBS)
全基因组重亚硫酸盐甲基化测序(WGBS)可以在全基因组范围内精确的检测所有单个胞嘧啶碱基(C碱基)的甲基化水平,是DNA甲基化研究的金标准。WGBS能为基因组DNA甲基化时空特异性修饰的研究提供重要技术支持,能广泛应用在个体发育、衰老和疾病等生命过程的机制研究中,也是各物种甲基化图谱研究的首选方法。
易基因全基因组甲基化测序技术通过T4-DNA连接酶,在超声波打断基因组DNA片段的两端连接接头序列,连接产物通过重亚硫酸盐处理将未甲基化修饰的胞嘧啶C转变为尿嘧啶U,进而通过接头序列介导的 PCR 技术将尿嘧啶U转变为胸腺嘧啶T。
应用方向:
WGBS广泛用于各种物种,要求全基因组扫描(不错过关键位点)
- 全基因组甲基化图谱课题
- 标志物筛选课题
- 小规模研究课题
技术优势:
- 应用范围广:适用于所有参考基因组已知物种的甲基化研究;
- 全基因组覆盖:最大限度地获取完整的全基因组甲基化信息,精确绘制甲基化图谱;
- 单碱基分辨率:可精确分析每一个C碱基的甲基化状态。

易基因提供全面的表观基因组学(DNA甲基化、DNA羟甲基化)和表观转录组学(m6A、m5C、m1A、m7G、ac4C)、染色质结构与功能组学技术方案(ChIP-seq、ATAC-seq),详询易基因:0755-28317900。
相关阅读:
项目文章 | WGBS+RNA-seq揭示松材线虫JIII阶段形成过程中的DNA甲基化差异
Cell|易基因微量DNA甲基化测序助力中国科学家成功构建胚胎干细胞嵌合体猴,登上《细胞》封面
Nature | 易基因DNA甲基化测序助力人多能干细胞向胚胎全能8细胞的人工诱导
参考文献:
Tang K, Zhu X, Xie S, Lang Z, Zhu JK. Transgenerational increases in DNA methylation in Arabidopsis plants defective in active DNA demethylation. Proc Natl Acad Sci U S A. 2024 May 28;121(22):e2320468121. doi: 10.1073/pnas.2320468121. PubMed PMID: 38768356


