易基因:血液Target-BS揭示靶向DNA甲基化标记水平与患者乳腺癌风险相关|Nature子刊
大家好,这里是专注表观组学十余年,领跑多组学科研服务的易基因。
符合遗传性乳腺癌和卵巢癌(HBOC)标准的患者中,只有不到15-20%携带致病性编码基因突变,意味着可能存在其他分子机制导致这种疾病的风险增加。外周血DNA甲基化是乳腺癌(BC)风险的潜在表观遗传标记。在分子水平上,BC 组织中DNA甲基化标记变化主要发生在致癌发展的早期阶段。但当前的研究主要集中在有限数量的基因和HBOC患者上,并没有在大规模基因Panel中分析没有遗传致病变异的患者DNA甲基化情况。
2024年6月,墨西哥国立自治大学Iztacala高等研究学院Felipe Vaca-Paniagua团队通过靶向亚硫酸盐测序(targeted bisulfite sequencing)对18个肿瘤抑制基因(抑癌基因)启动子(330个CpG位点)进行甲基化分析。揭示了:① EPCAM(17个CpG位点;p=0.017)和RAD51C(27个CpG位点;p=0.048)中的高甲基化;② BC患者中36个CpG特异性位点的高甲基化(FDR q<0.05);③ 四个特定CpG位点与更高BC风险相关(FDR q<0.01, Bonferroni p<0.001):cg89786999-FANCI(OR=1.65;95% CI:1.2–2.2)、cg23652916-PALB2(OR=2.83;95% CI:1.7–4.7)、cg47630224-MSH2(OR=4.17;95% CI:2.1–8.5)和cg47596828-EPCAM(OR=1.84;95% CI:1.5–2.3)。在澳大利亚的一个队列中对cg47630224-MSH2甲基化进行验证,显示BC风险增加3倍(AUC: 0.929;95% CI: 0.904–0.955)。表明四个DNA甲基化CpG位点可能与更高的BC风险相关,可能作为未检测到编码突变的患者的生物标志物。相关研究成果以“Methylation marks in blood DNA reveal breast cancer risk in patients fulfilling hereditary disease criteria”发表在《npj Precision Oncology》(IF 6.8)期刊上。

研究方法:
- 研究对象包括231名符合HBOC标准的乳腺癌患者(这些患者未检测到编码致病变异)和156名健康对照组。
- 通过靶向亚硫酸盐测序(targeted bisulfite sequencing,Target-BS)对18个肿瘤抑制基因启动子区域(330个CpG位点)进行甲基化分析。
- 使用统计和生物信息学方法分析甲基化数据,包括Wilcoxon秩和检验、多元逻辑回归分析、ROC曲线分析等。
- 独立队列验证。
研究结果:
(1)实验设计
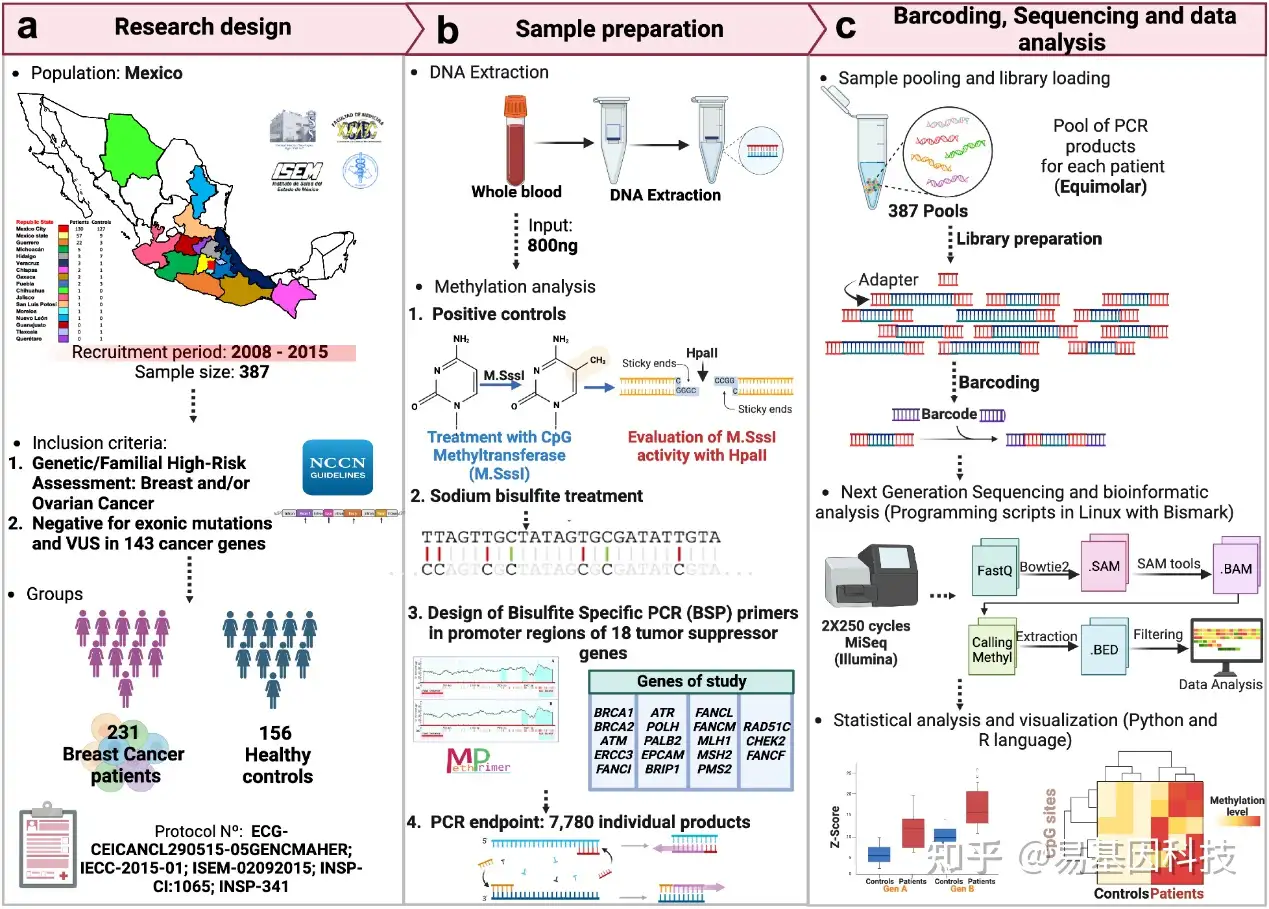
图1:实验设计
a. 患者选择的招募和入选标准
b. Target-BS样本准备、亚硫酸盐处理、靶向启动子和总扩增分析
c. DNA文库制备workflow、条形码、自动化生物信息学分析和生物统计分析
(2)高通量DNA甲基化测序揭示高风险乳腺癌患者抑癌基因中的异常甲基化。
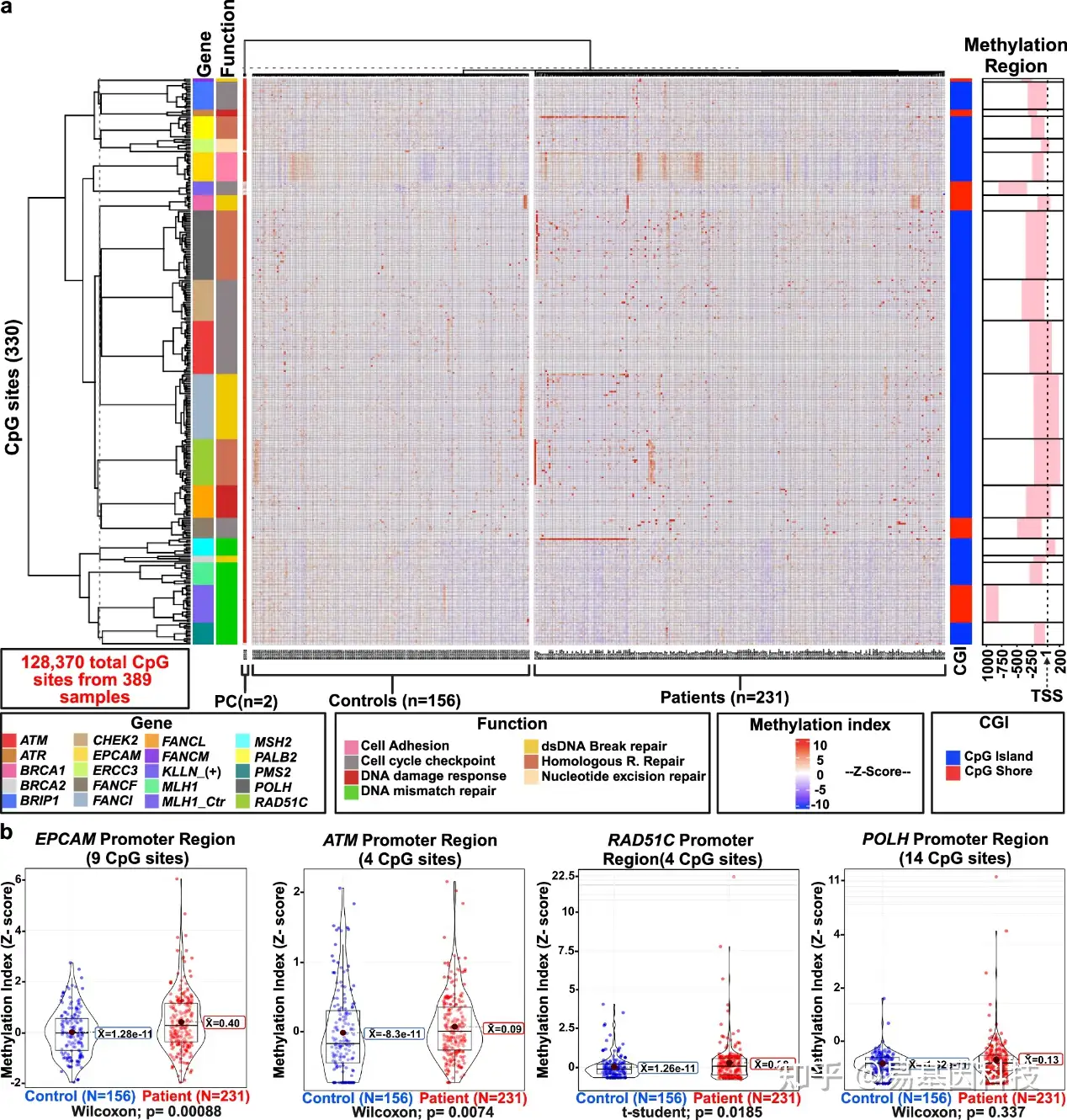
图2:乳腺癌抑癌基因启动子区域的高甲基化CpG位点。
a.分析抑癌基因(TSGs)启动子区域的位点特异性甲基化状态。
b. EPCAM、ATM、RAD51C和POLH中高甲基化CpG位点的差异水平。TSS表示转录起始位点,CGI表示CpG岛相对位点,PC表示阳性对照,TSGs代表抑癌基因。
(3)乳腺癌患者在四个特定CpG位点表现出高甲基化富集
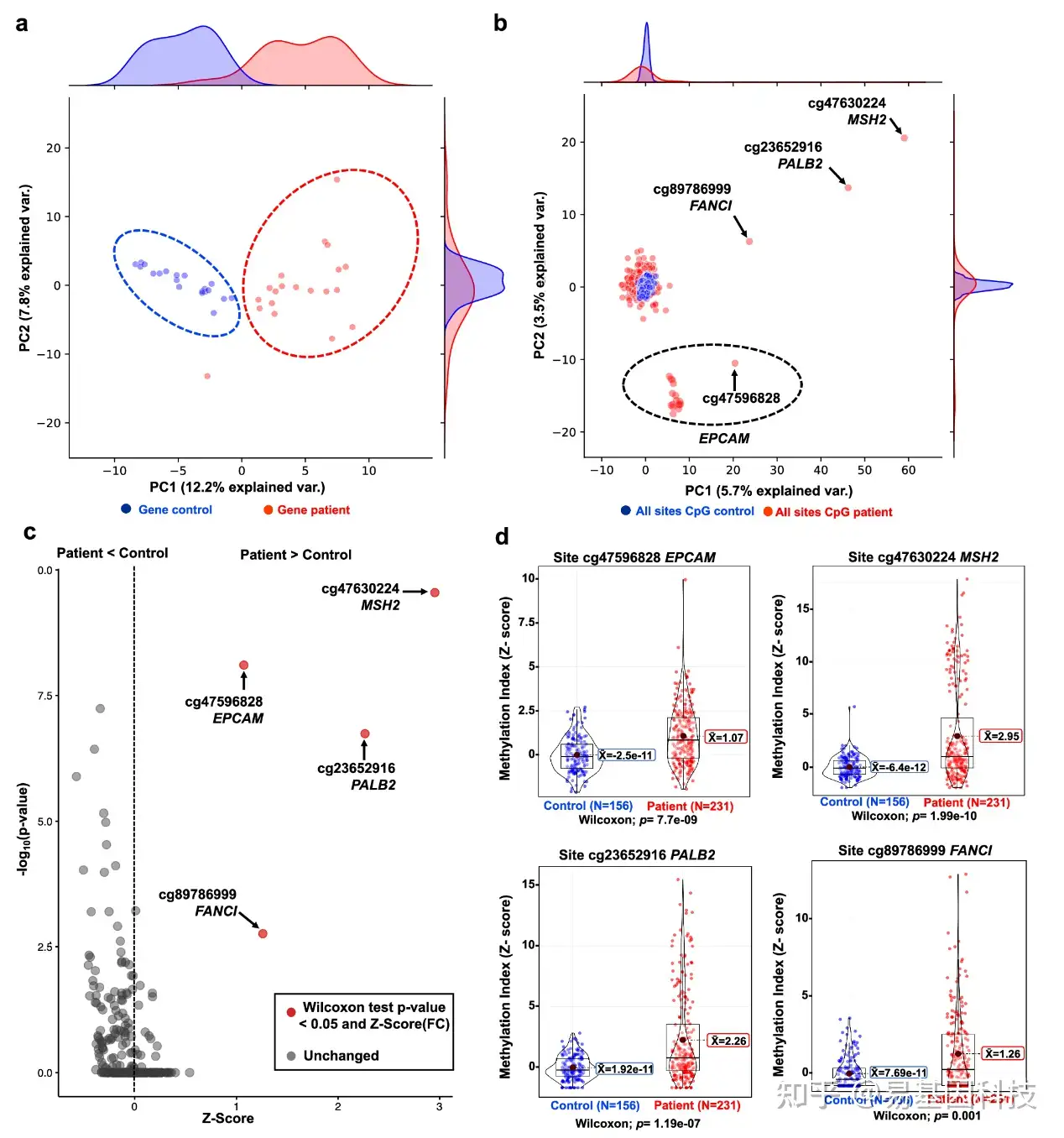
图3:四种异常DNA甲基化标记与乳腺癌相关。
a. 每个启动子的平均Z分数的主成分分析(PCA)。
b. 330个特异性CpG位点的PCA分析。cg8978699-FANCI、cg23652916-PALB2、cg47630224-MSH2和cg47596828-EPCAM的差异最大(箭头所示)。所有EPCAM位点形成了一个独立的组。
c. 各个CpG位点的差异高甲基化水平。红点代表显著位点。
d. 与对照组相比,显著CpG位点的差异高甲基化。
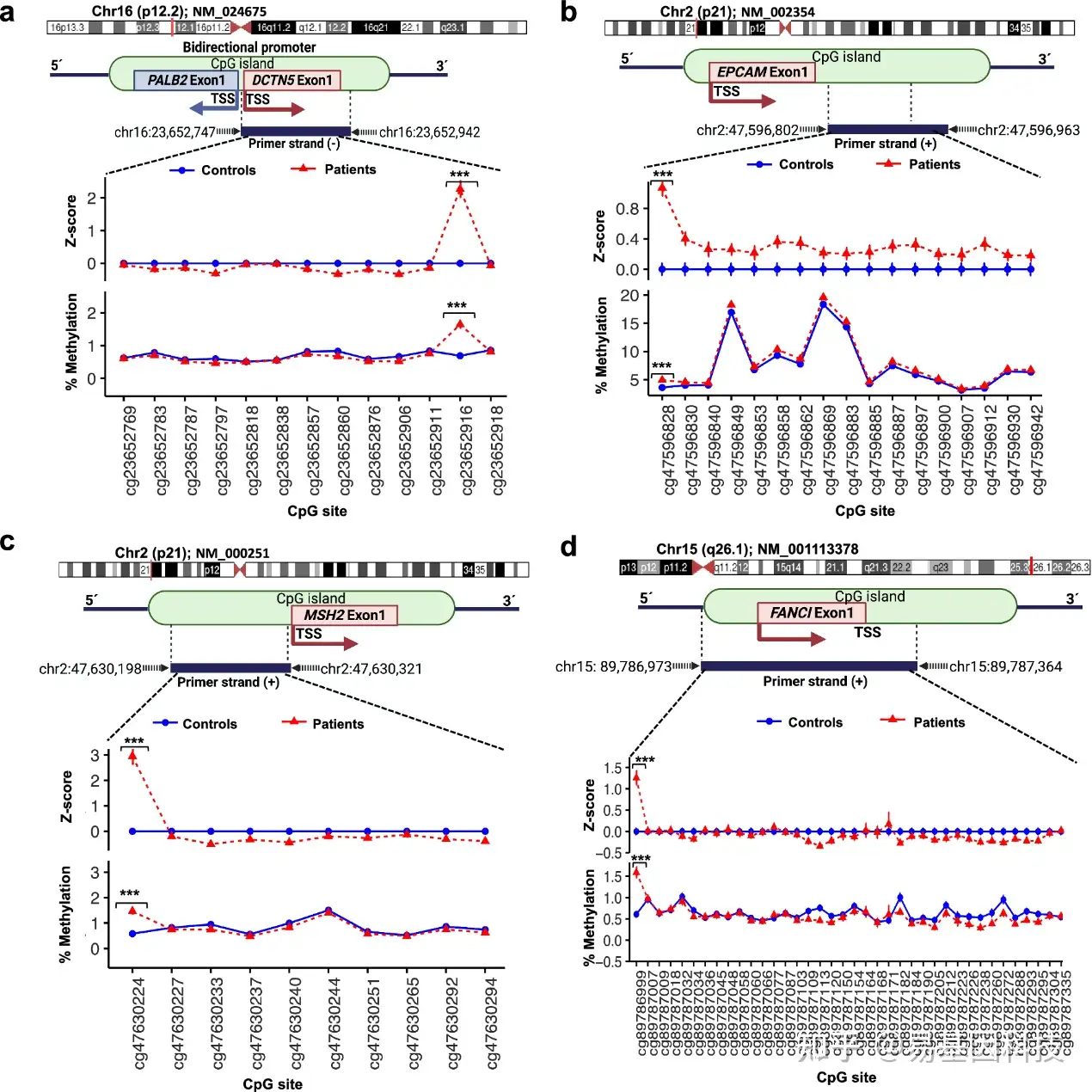
图4:比较患者组与对照组中PALB2、EPCAM、MSH2和FANCI启动子区域的位点特异性种系甲基化水平。
a-d. 显示四个基因位点。蓝色实线表示来自血液对照的平均甲基化值。橙色虚线表示患者平均甲基化值。位点cg47596828-EPCAM、cg47630224-MSH2、cg23652916-PALB2和cg89786999-FANCI在患者中显示出统计学上显著的甲基化水平增加。基因区域的示意图表示CpG岛和转录起始位点(TSS)。
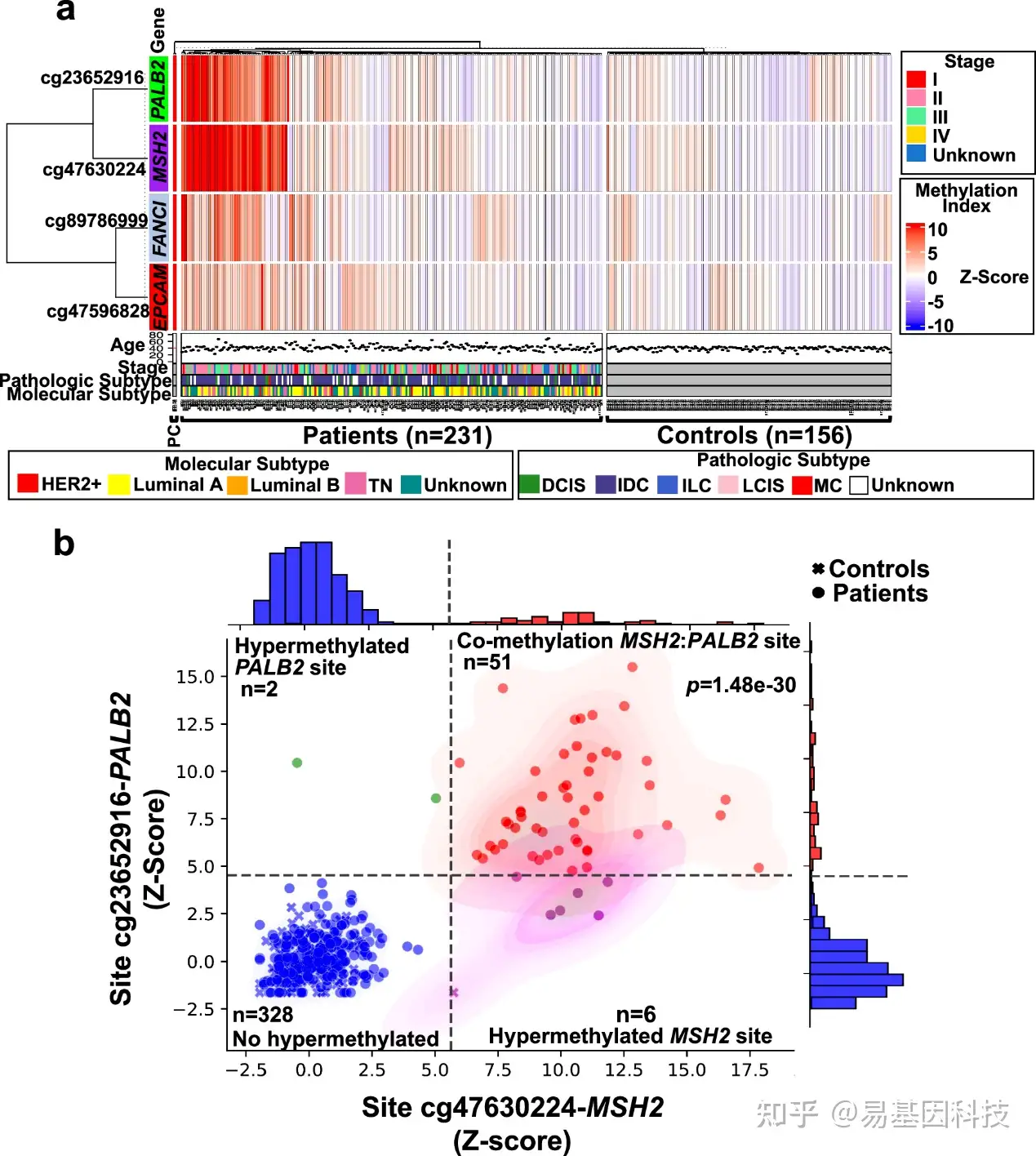
图5:cg23652916-PALB2和cg47630224-MSH2位点的共甲基化。
a. 监督分层聚类热图显示51名患者中MSH2-PALB2共甲基化亚群。Y轴:CpG位点。X轴:对照组和患者。下方的条形显示每个患者与每个高甲基化位点相关的年龄、组织病理学和分子亚型。DCIS代表导管原位癌,LCIS代表小叶原位癌,IDC代表浸润性导管癌,ILC代表浸润性小叶癌,MC代表髓样癌,PC代表阳性对照。
b. cg23652916-PALB2和cg47630224-MSH2的Z分数共甲基化水平。
(4)四个特异性高甲基化CpG位点作为乳腺癌风险的潜在生物标志物
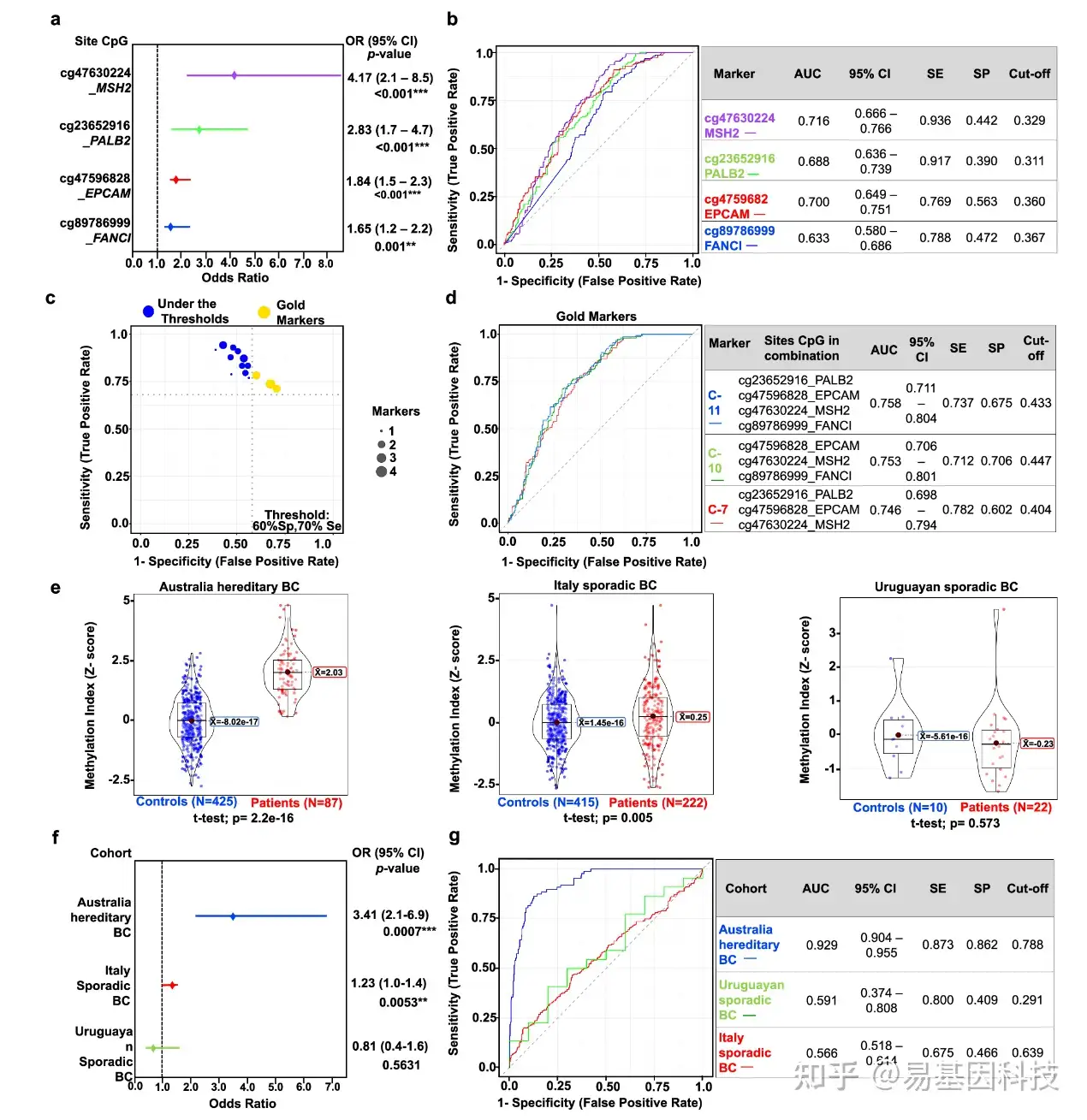
图6:独立验证队列中四个CpG位点的生物标志物潜力以及 cg47630224-MSH2 与乳腺癌风险之间的关联。
a. 四个高甲基化CpG位点的乳腺癌风险。
b. 四个CpG位点的ROC性能。
c. 对11个CpG位点组合的灵敏度和特异性进行综合分析。黄色气泡代表最佳标记组合(Se>70%,Sp>60%),称为“金标准标记”。
d. 金标准标记组合7、10和11的生物标志物能力。
e. 在三个国际队列中对cg47630224-MSH2进行差异甲基化分析。
f. cg47630224-MSH2位点与三个队列中BC风险的关联分析。
g. 所有队列中cg47630224-MSH2位点的生物标志物能力:澳大利亚(蓝色)、乌拉圭(绿色)、意大利(红色)。
易小结
本研究在单个碱基分辨率下对大型病例对照数据集中的高风险基因启动子区域进行全面分析,利用健康人群对照来最小化非遗传和环境因素引起的甲基化水平变异。提供了关于符合HBOC标准的乳腺癌患者种系甲基化与癌症易感性基因之间的相关性证据(这些患者没有可检测到的编码致病变异)。研究共鉴定出四个新的潜在表观遗传标记,与更高的乳腺癌风险相关,这些标记在独立队列中得到了验证。总体而言,这项工作有助于提高对高风险乳腺癌患者的表观遗传谱的理解,这可能是病因学的其他机制。进一步研究表观遗传标记有潜力增强风险评估,并促进乳腺癌管理中的个性化方法。
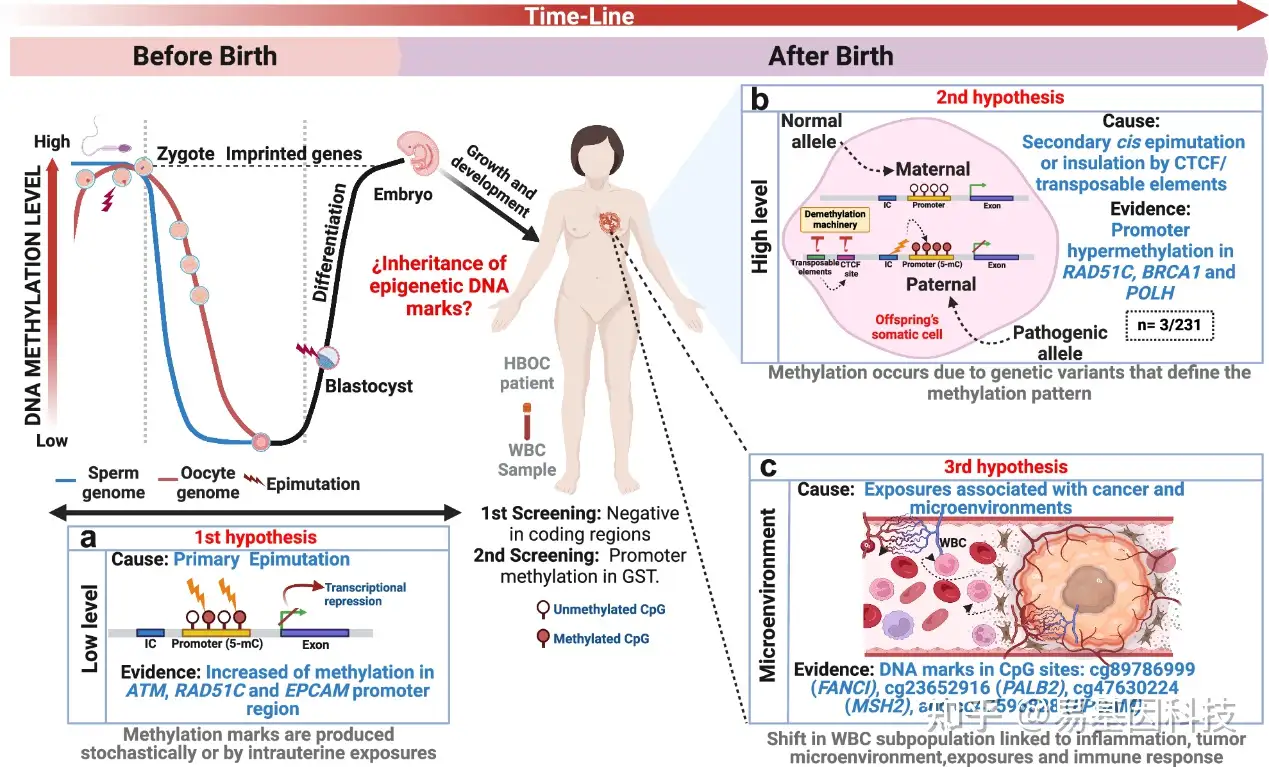
图7:与乳腺癌风险相关的异常甲基化标记模型。
关于靶基因DNA甲基化测序(Target-BS)
对目标基因/CpG 位点甲基化检测,建立灵活的靶向甲基化测序技术。易基因建立的靶基因甲基化高通量测序(Target Bisulfite sequencing,Target-BS)是针对已有目标基因panel组合,运用易基因自主研发的甲基化特异性多重PCR引物设计软件,对亚硫酸盐转化后的目标基因多重扩增,建库后进行超高深度(1000X以上)甲基化精准检测。
应用方向:
Target-BS广泛用于已知位点的甲基化Biomarker筛选、验证及临床转化应用
- 后续目标基因的甲基化验证
技术优势:
- 多基因多重扩增,检测通量高;
- 超高深度亚硫酸盐甲基化测序,相对于传统BSP克隆测序,检测灵敏度、准确性高,可准确检测到样本中低于1%的甲基化水平;
- 样本需求量低,20ng基因组DNA可实现多基因扩增;
- 适用范围广,对基因组DNA、FFPE样本、游离cfDNA等样本均适用;
- 检测费用显著低于现有技术、快周期、超高性价比。
参考文献:
Ruiz-De La Cruz M,et al. Methylation marks in blood DNA reveal breast cancer risk in patients fulfilling hereditary disease criteria. NPJ Precis Oncol. 2024 Jun 19;8(1):136. pii: 10.1038/s41698-024-00611-z. doi: 10.1038/s41698-024-00611-z. PubMed PMID: 38898118.
相关阅读:


