易基因: RRBS揭示基于DNA甲基化驱动基因的肾透明细胞癌预后模型的鉴定和验证|项目文章
大家好,这里是专注表观组学十余年,领跑多组学科研服务的易基因。
肾细胞癌(RCC)是最常见的肾癌亚型,每年超400万例新发病例,是泌尿系统恶性肿瘤导致的第二大死因。2%-70%的RCC为透明细胞RCC(Clear cell renal cell carcinoma,ccRCC)。DNA甲基化(DNA methylation,DNAm)是主要的表观遗传修饰之一,在维持基因转录和基因组稳定性方面具有至关重要的作用。DNAm在ccRCC的发病机制中起着重要作用,许多研究集中于DNAm异常变化与ccRCC预后之间的相关性,但基于DNAm驱动基因的ccRCC预后模型鲜有报道。
2023年6月7日,南方医科大学附属龙华人民医院泌尿外科邓琼研究员等在《BMC Genomics》发表题为“Identification and validation of a DNA methylation-driven gene-based prognostic model for clear cell renal cell carcinoma”的研究论文,该研究以ccRCC患者的DNA为对象,通过简化基因组重亚硫酸盐测序(RRBS)等技术方法,鉴定出ccRCC患者的ccRCC早期诊断和预后的DNA甲基化生物标志物。深圳市易基因科技有限公司为本研究提供RRBS建库测序分析服务。

标题:Identification and validation of a DNA methylation-driven gene-based prognostic model for clear cell renal cell carcinoma(基于DNA甲基化驱动基因的肾透明细胞癌预后模型的鉴定和验证)
时间:2023-06-07
期刊:BMC Genomics
影响因子:IF 4.4
技术平台:RRBS等

摘要:
肾透明细胞癌(ccRCC)是一种形态异质且预后不良的恶性肿瘤。本研究旨在建立DNA甲基化(DNAm)驱动的ccRCC基因预后模型。本研究对ccRCC患者的DNA提取物进行了RRBS测序分析。对10对患者样本的RRBS数据进行分析以筛选候选CpG位点,然后训练并验证18个CpG位点模型,并结合临床特征建立了用于ccRCC预后或风险评估的列线图(Nomogram model)模型。研究在启动子区域鉴定出2261个DMR。经DMR筛选后,共筛选出578个候选基因,与450K芯片中的408个CpG二核苷酸相对应。从TCGA数据集中收集了478个ccRCC样本的DNAm图谱。通过单因素Cox回归、LASSO回归和多因素Cox比例风险回归分析对319个样本的training集进行分析,共鉴定出18个CpG的预后组。结合临床特征构建了预后模型,在测试集(test set,159个样本)和整组(whole set,478个样本)中,Kaplan-Meier曲线差异显著;ROC曲线和生存分析显示AUC>0.7。结合临床病理特征和甲基化风险评分的列线图表现较好,决策曲线分析也显示出有益的效果。
本研究深入了解了高甲基化在ccRCC中的作用。鉴定出的靶标可作为ccRCC早期诊断和预后判断的生物标志物。本研究对更好的风险分层和个性化治疗具有重要意义。
研究结果
(1)RRBS质控结果

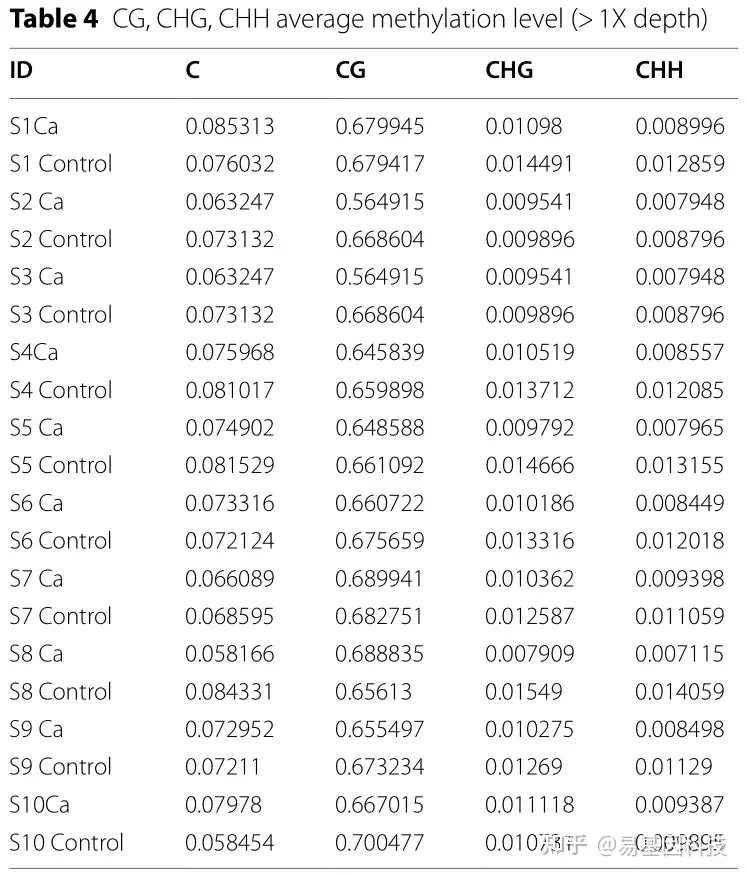
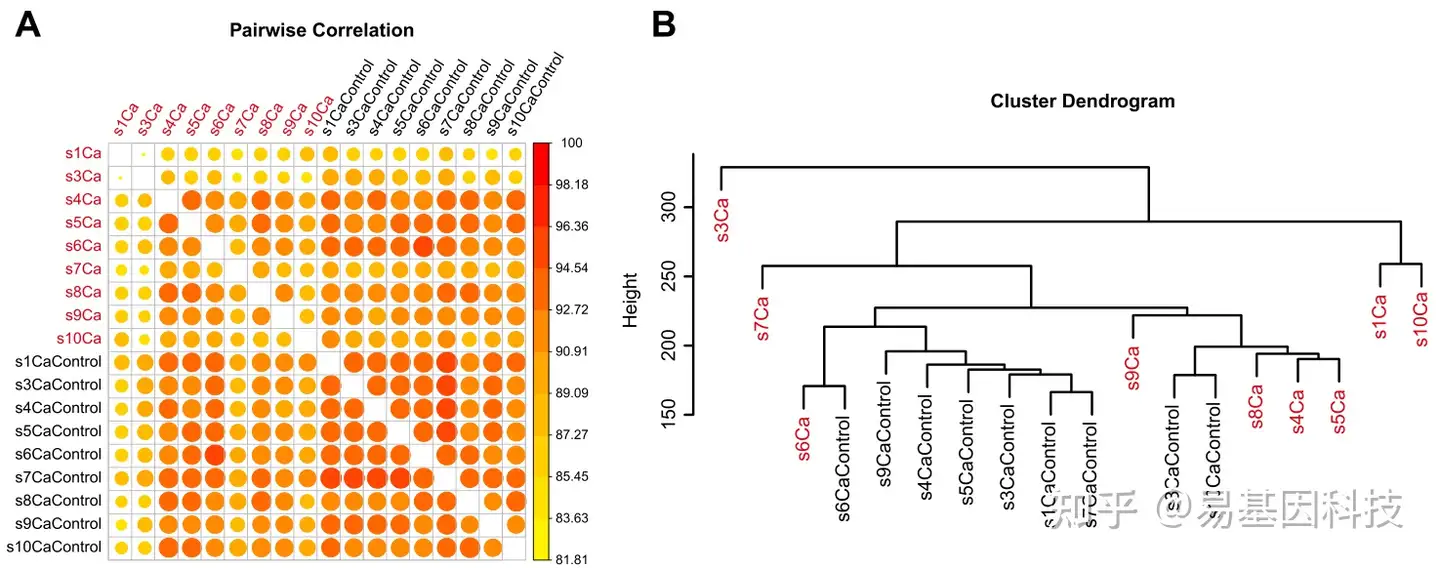
图1:样本间全基因组甲基化差异。
- 样本中CpG甲基化水平相关性。较大的圆形区域和较深的颜色表示高相关性。
- 聚类树状图。根据欧几里得距离,使用R.Ward.d2的hclust函数对样本进行聚类。选择最小方差方法作为聚类方法
(2)DMR鉴定
图2:DMR分析
- 平均甲基化差异。X轴代表甲基化差异。负值表示低甲基化。正值表示高甲基化。Y轴表示相应横坐标的DMR数量。
- DMR甲基化分布密度。X轴代表Ca组中的DMR甲基化水平;Y轴代表对照组中的DMR甲基化水平。DMR密度从低到高(白色到红色)。
- DMR在基因元件上的分布
(3)ccRCC预后模型构建

图3:差异甲基化CpG(differentially methylated CpG,dmCpG)位点鉴定。
A、 使用调谐参数(lambda)对CpG位点进行LASSO回归分析。
B、 蛋白质-蛋白质互作。
C、 18个dmCpG位点的DisGeNet疾病富集分析。
D、 18个dmCpG位点的GO富集分析
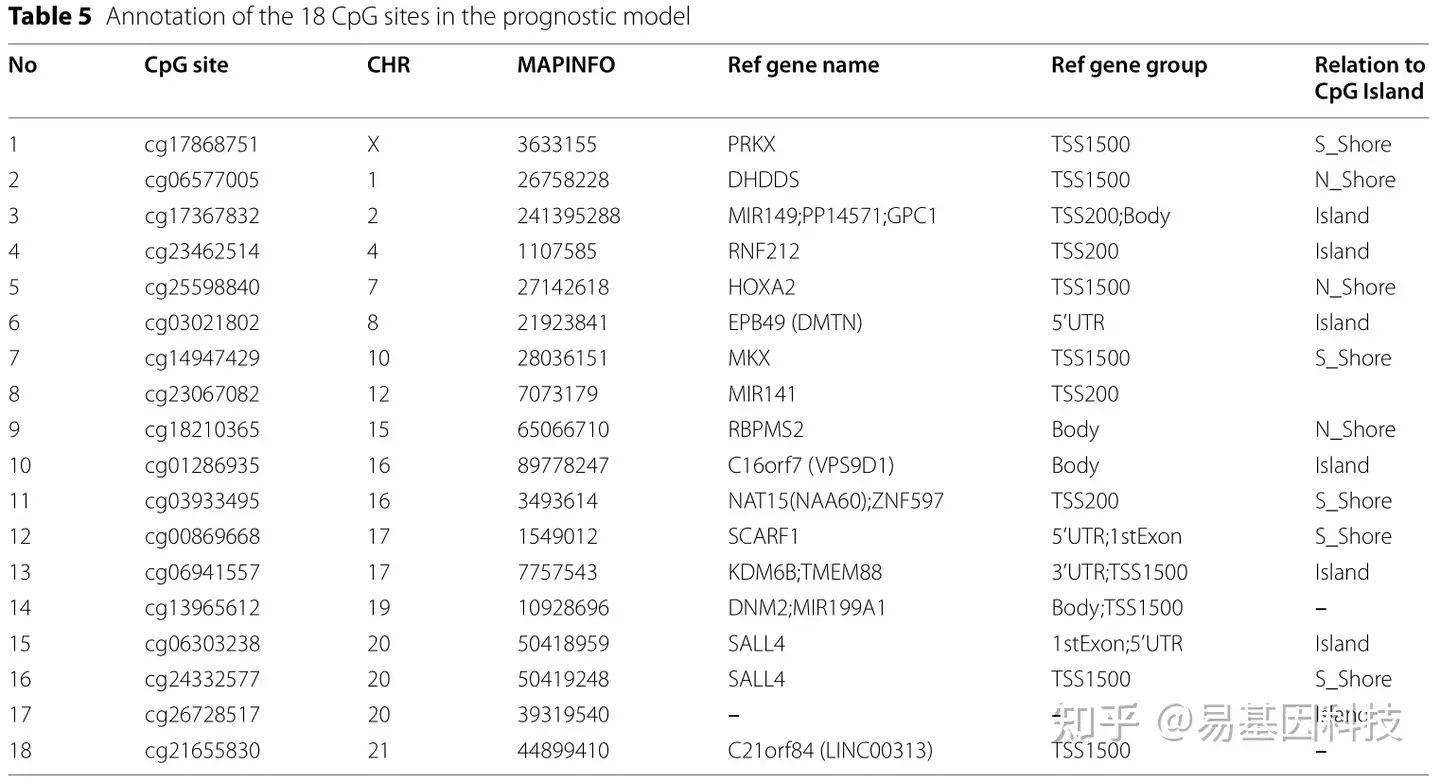
表5:预后模型中18个CpG位点的注释
(4)预后模型的Training
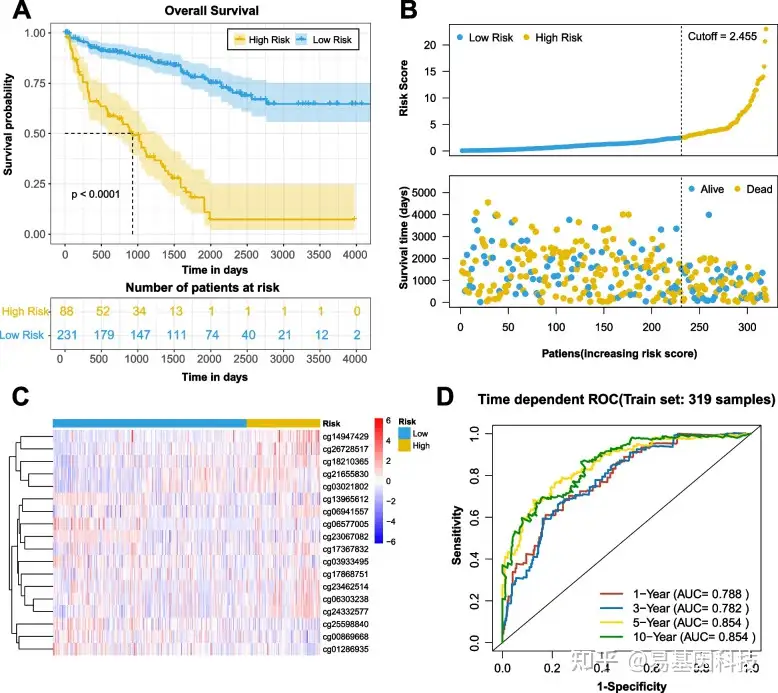
图4:training集中的风险评分
- 高风险组和低风险组患者的KM生存曲线。数据显示为四分位间距的中位数。使用Log-rank检验评估统计学显著性。虚线显示50%生存概率的显著性。
- 计算高风险和低风险患者的风险评分和生存状态等级。虚线显示区分ccRCC高风险和低风险患者的临界值。
- C.18个CpG位点甲基化水平热图。
- 风险评分的1年、3年、5年和10年ROC曲线。以临界值决定该模型的敏感性和特异性
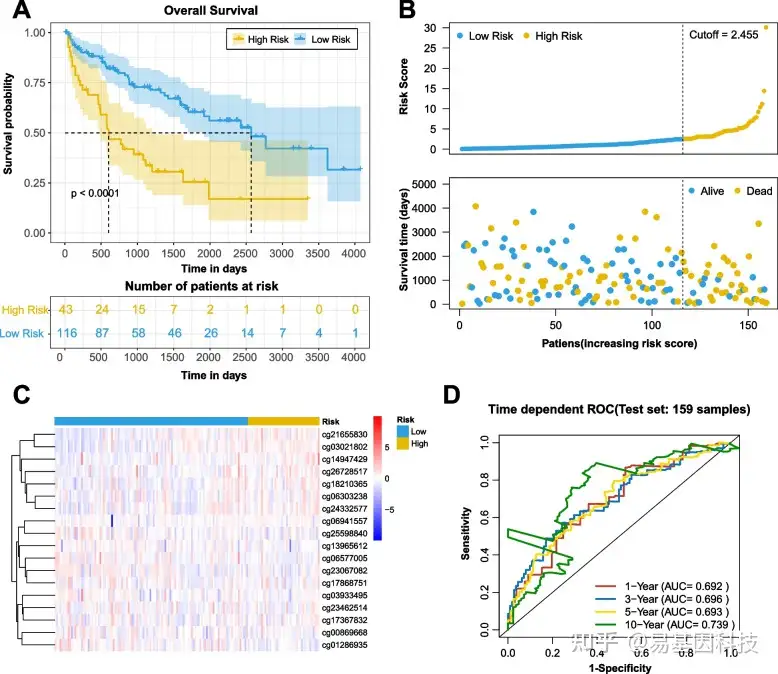
图5:test集中的风险评分
(5)预后模型的验证

图6:基于预后模型和临床特征的列线图
预后模型和临床特征的单因素(A)和多因素(B)回归分析。(C)预测ccRCC患者1年、3年、5年和10年生存时间概率列线图
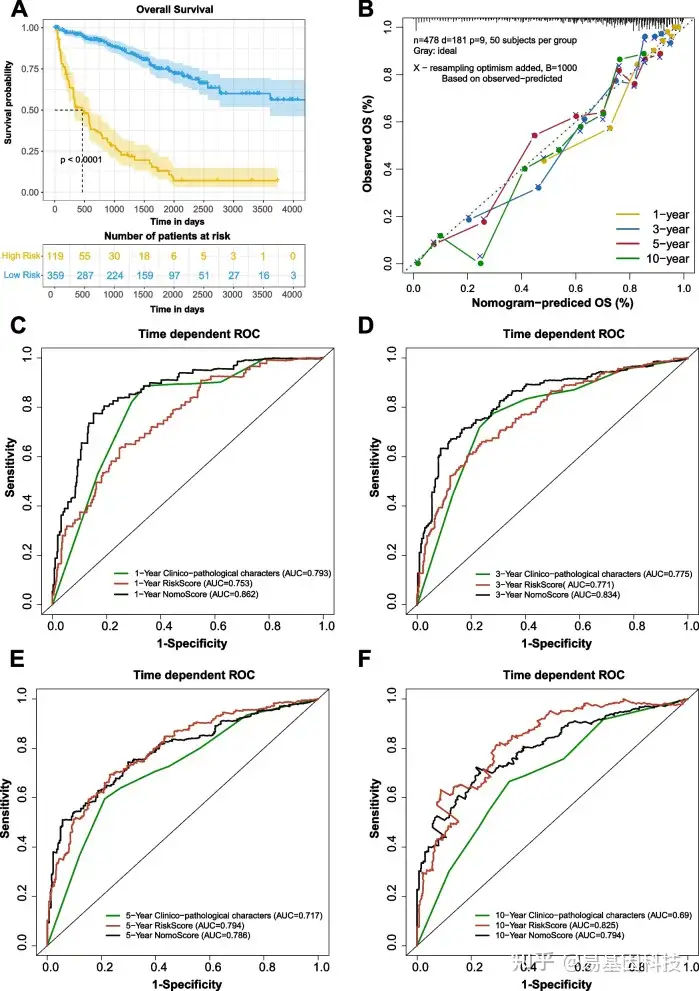
图7:模型校准和ROC评估
- 高风险组和低风险组患者的KM生存曲线。
- 预测ccRCC患者1年、3年、5年和10年总生存期(OS)的列线图。
C-F. 1年(C)、3年(D)、5年(E)、10年(F)的临床病理特征(绿色,与肿瘤分期诊断、年龄和肿瘤组织学分级相结合),RiskScore(红色)和NomoScore(黑色)的ROC曲线
结论
本研究分析了患者样本中的RRBS数据以筛选原始候选CpG位点,然后利用TCGA-KIRC数据训练和验证了18个CpG位点模型,并结合临床特征建立了用于ccRCC预后或风险评估的列线图模型。
基于该结果,研究开发并验证了这种新型预后模型是一种实用、可靠的ccRCC患者预后工具。本研究结果支持异常DNAm变化与肿瘤发生密切相关的观点,并为ccRCC提供了潜在的新型预后生物标志物,对更好的风险分层和个性化治疗具有重要意义。
关于易基因简化基因组甲基化测序研究解决方案
简化甲基化测序(Reduced Representation Bisulfite Sequencing,RRBS)是利用限制性内切酶对基因组进行酶切,富集启动子及CpG岛等重要的表观调控区域并进行重亚硫酸盐测序。该技术显著提高了高CpG区域的测序深度,在CpG岛、启动子区域和增强子元件区域可以获得高精度的分辨率,是一种准确、高效、经济的DNA甲基化研究方法,在大规模临床样本的研究中具有广泛的应用前景。
为适应科研技术的需要,易基因进一步开发了可在更大区域内捕获CpG位点的双酶切RRBS(dRRBS),可研究更广泛区域的甲基化,包括CGI shore等区域。
为助力适用低起始量DNA样本(5ng)量多维度甲基化分析,易基因开发了富集覆盖CpG岛、启动子、增强子、CTCF结合位点的甲基化靶向基因组测序方法:extended-representation bisulfite sequencing(XRBS),实现了高灵敏度和微量样本复用检测,使其具有高度可扩展性,并适用于有限的样本和单个细胞基因组CG位点覆盖高达15M以上。
技术优势:
- 起始量:100ng gDNA;
- 单碱基分辨率;
- 多样本的覆盖区域重复性可达到85%-95%、测序区域针对高CpG调控区域,数据利用率更高;
- 针对性强,成本较低;
- 基因组CG位点覆盖高达10-15M,显著优于850K芯片。
应用方向:
RRBS/dRRBS/XRBS广泛应用于动物,要求全基因组扫描(覆盖关键调控位点)的:
- 队列研究、疾病分子分型、临床样本的甲基化 Biomarker 筛选
- 复杂疾病及肿瘤发病机制等甲基化研究
- 模式动物发育和疾病甲基化研究
| 技术参数 | RRBS | DRRBS | cfDNA-RBS | Micro-RBS | sc-RBS |
|---|---|---|---|---|---|
| 原理 | MspI酶切+连接接头 | 多酶切+连接接头 | CCGG邻近片段连接接头 | CCGG邻近片段连接接头 | CCGG邻近片段连接接头 |
| 样本要求 | 1μg | 1μg | 1ng | 1ng | 单细胞/1-10个细胞 |
| 测序数据量 | 10G | 15G | 20G | 20G | 2G |
| 5XCG位点覆盖 | 6M | 8M | 6M | 10M | 4-8M |
易基因科技提供全面的DNA甲基化研究整体解决方案。
参考文献:
Deng Q, Du Y, Wang Z, Chen Y, Wang J, Liang H, Zhang D. Identification and validation of a DNA methylation-driven gene-based prognostic model for clear cell renal cell carcinoma. BMC Genomics. 2023 Jun 7;24(1):307.
相关阅读:
oxRRBS+RRBS揭示牦牛下丘脑在神经调节和髓鞘形成中的表观调控机制


 浙公网安备 33010602011771号
浙公网安备 33010602011771号