易基因|动物发育过程中顺式调控区域的活性DNA去甲基化早于脊椎动物起源:重磅研究
易基因|动物发育过程中顺式调控区域的活性DNA去甲基化早于脊椎动物起源:重磅研究
大家好,这里是专注表观组学十余年,领跑多组学科研服务的易基因。
2022年12月02日,澳大利亚悉尼加尔文医学研究所基因组学和表观遗传学系Ozren Bogdanovic研究团队在《SCIENCE ADVANCES》杂志发表了题为“Active DNA demethylation of developmental cis-regulatory regions predates vertebrate origins”的研究论文,该研究通过WGBS、ACE-seq、hMeDIP-seq、ATAC-seq等技术对海胆(sea urchin,Strongylocentrotus purpuratus)、文昌鱼(lancelet,Branchiostoma lanceolatum)等动物胚胎和成体组织进行测序分析,揭示了动物发育过程中从调控区域主动去除5mC早于脊椎动物起源。

标题:Active DNA demethylation of developmental cis-regulatory regions predates vertebrate origins
时间:2022.12.02
期刊:SCIENCE ADVANCES
影响因子:IF 14.957
技术平台:WGBS、ACE-seq、hMeDIP-seq、ATAC-seq
样本实验:
海胆、文昌鱼胚胎和成体组织进行WGBS、ACE-seq、hMeDIP-seq、ATAC-seq测序分析,实验各设置2个生物学重复
研究思路:
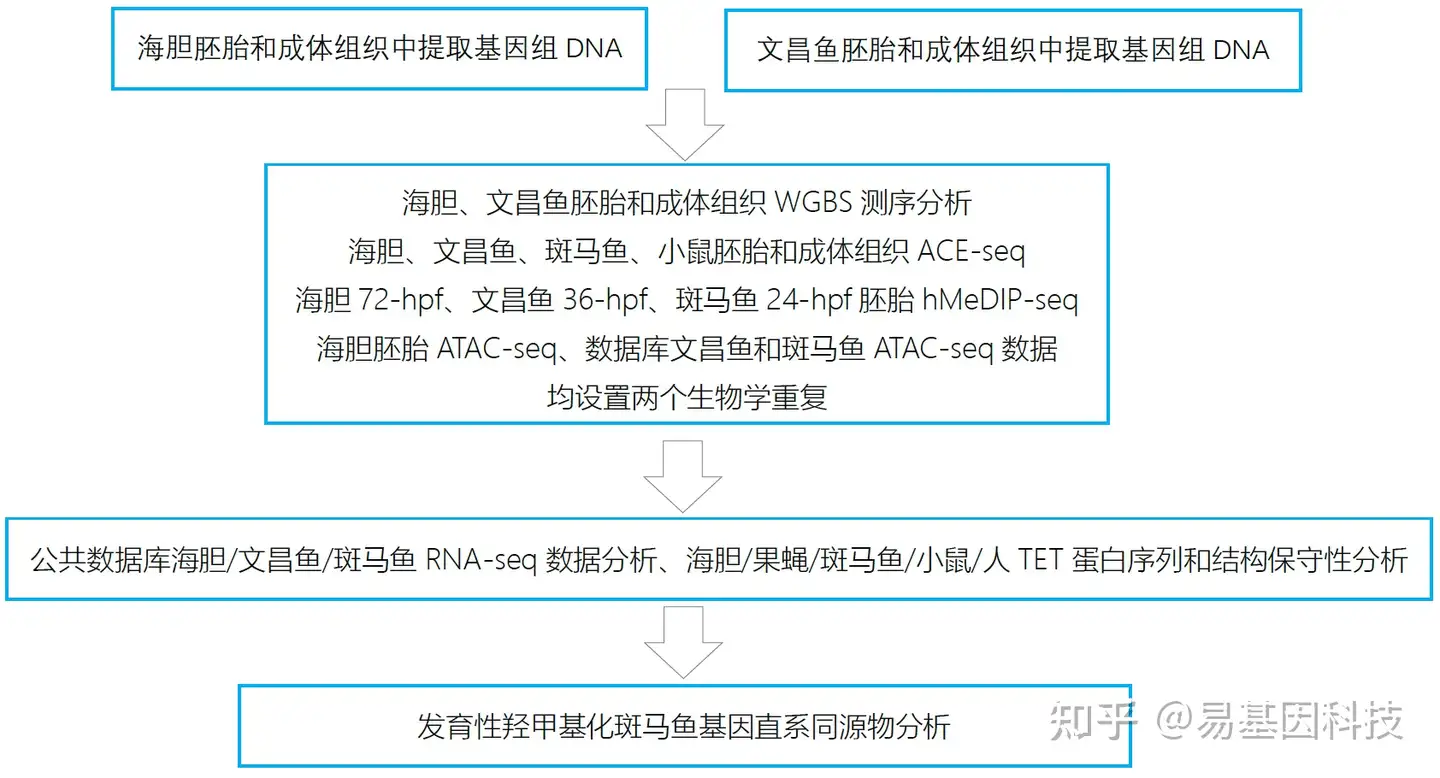
摘要:
DNA甲基化(5-甲基胞嘧啶(5mC))是脊椎动物胚胎发生所需的抑制基因调控标记。基因组5mC通过DNA甲基转移酶(沉积5mC)和TET酶(通过形成5-羟甲基胞嘧啶(5hmC)参与其活性去除)的作用而受到严格调控。TET酶对哺乳动物原肠胚形成和脊椎动物发育增强子的激活至关重要,然而迄今为止仍对无脊椎动物谱系中的5hmC功能、丰度和基因组分布缺乏清晰的了解。本研究通过对海胆和文昌鱼胚胎发生过程中的5mC和5hmC谱进行单碱基定量分析,阐明了无脊椎动物5hmC和TET酶的作用。研究发现无脊椎后口动物(invertebrate deuterostomes)通过TET酶靶向去甲基化与发育基因相关的调控区域,并表明已鉴定的5hmC调控基因的补体在脊椎动物中保守。本研究表明,从调控区域主动去除5mC是后口动物胚胎发生的共同特征,暗示了主要基因调控模块的意外深度保守。
结果图形:
(1)TET酶的结构和功能保守
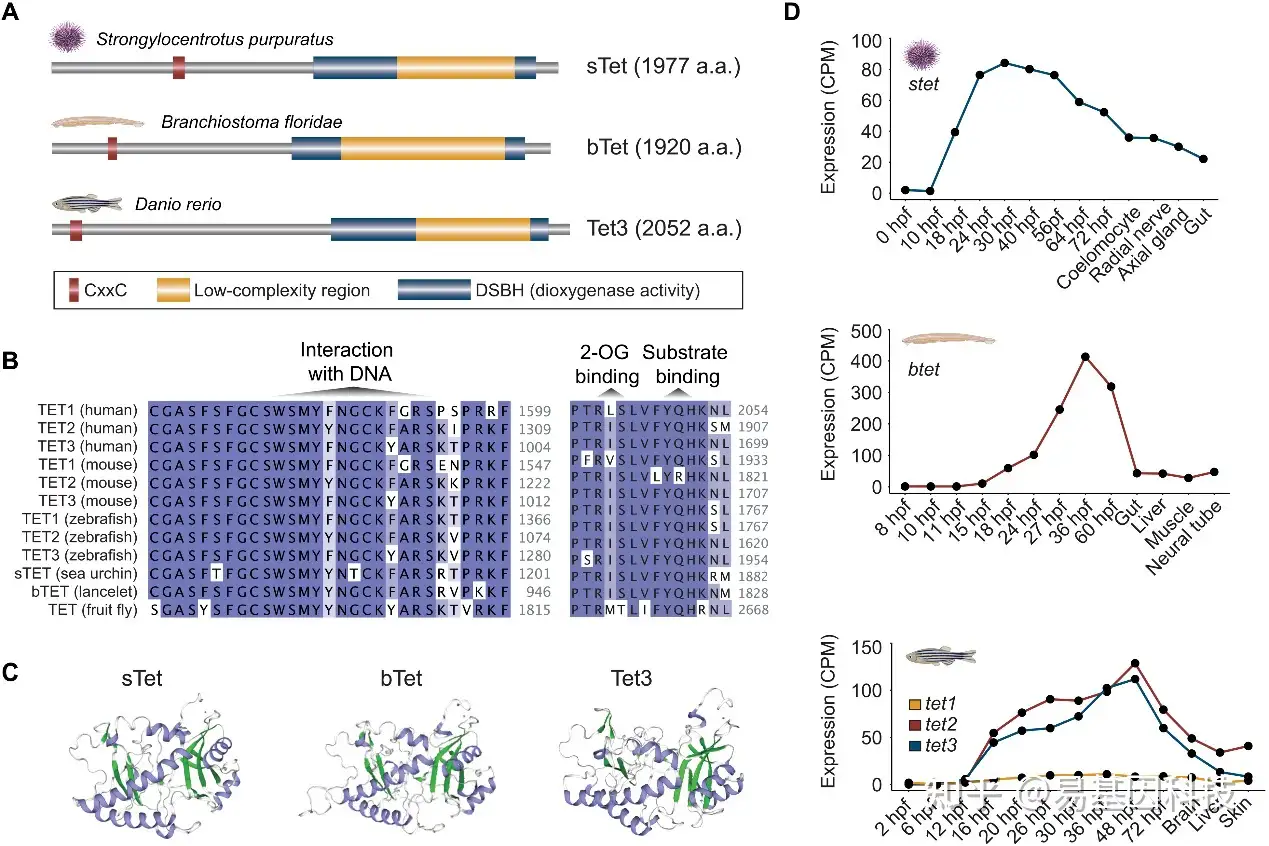
图1:TET蛋白序列、催化结构域结构和发育表达的进化保守性
(A) 使用SWISS-MODEL和HMMER预测海胆、文昌鱼和斑马鱼TET蛋白结构域结构。海胆sTet、佛罗里达文昌鱼bTet和斑马鱼Tet3含有N-端DNA结合CxxC结构域和包含大型低复杂性插入片段的C-端催化DSBH结构域。
(B) 人/小鼠/斑马鱼TET1/TET2/TET3以及海胆/文昌鱼/果蝇TET催化核心结构域的多序列比对。显示了DSBH的一部分。每种氨基酸的颜色表示百分比同一性(PID),深蓝表示高PID,浅蓝表示低PID。(C) 使用SWISS-MODEL进行海胆sTet、文昌鱼bTet和斑马鱼Tet3的甲基胞嘧啶双加氧酶结构域的三维模型。人TET2-5fC复合体(5d9y.1.A)晶体结构用于模型构建。蓝色表示ɑHelix,绿色表示βsheet。
(D) TET基因在海胆、文昌鱼和斑马鱼发育过程中的表达变化。a.a表示amino acids;2-OG表示2-oxoglutarate;CPM表示百万计数(counts per million);hpf表示受精后小时数(hours post fertilization)。
(2)WGBS揭示海胆和文昌鱼基因组的DNA甲基化含量和变化
为量化可作为TET氧化和5hmC形成基底的基因组5mC数量,作者利用WGBS绘制了海胆发育过程的4个时期相对应的5mC谱,分别为受精后24小时(24hpf,囊胚)、受精后48小时(48 hpf,原肠胚)、受精后72小时(72hpf,pluteus)和成体时期(tube feet)。此外还分析了8 hpf(囊胚,G3)、15 hpf(原肠胚,G6)、36 hpf(神经胚,T0)和成体(肝脏,A)时期文昌鱼胚胎发生的WGBS数据(图2A)。结果与先前研究一致,平均甲基化水平(海胆23-28%,文昌鱼21-25%)与典型的无脊椎动物甲基体模式一致,大多数基因组5mC位于表达基因的基因体内。值得注意的是,海胆和文昌鱼的成体样本显示出非常低的5mC水平。鉴于无脊椎动物中基因表达与基因体5mC之间的正相关关系,成体组织中的5mC水平较低可能由这些组织的表达基因数量少或总转录output低的特异性转录谱导致。为此作者分析了多个发育时期和成体组织的转录组,揭示了海胆和文昌鱼两种生物所有检测样本的平均表达谱(图2B)。结果表明在成体组织中观察到的5mC丢失可能并非由复杂转录的重大变化引起,而是与更频繁使用顺式调控元件相关的染色质可及性逐渐增加有关。根据这些结果,作者无法在成体组织中检测到海胆和文昌鱼DNMT转录本的任何主要发育下调。
由于海胆和文昌鱼DNMT对脊椎动物的高度结构保守,为进一步了解5mC的发育动态变化,作者鉴定出基因组区域在5mC水平上显示出显著的发育变化(错误发现率<0.05,ΔmCG/CG≥ 0.2)(图2C)。研究分别在海胆和文昌鱼基因组中鉴定20072和8136个差异甲基化区域(DMR),其中大多数对应于幼体到成体(larva/pluteus-to-adult)的转变(图2D)。大部分(86%的海胆和80%的文昌鱼)已鉴定的DMR与发育性低甲基化相关(图2C和D)。结果与海胆和文昌鱼两种生物成体组织中5mC的整体较低水平一致(图2A)。总之,研究发现采样的无脊椎后口动物的胚胎发生存在显著的5mC发育丢失,且不能通过转录活性或DNMT表达的发育变化来解释。
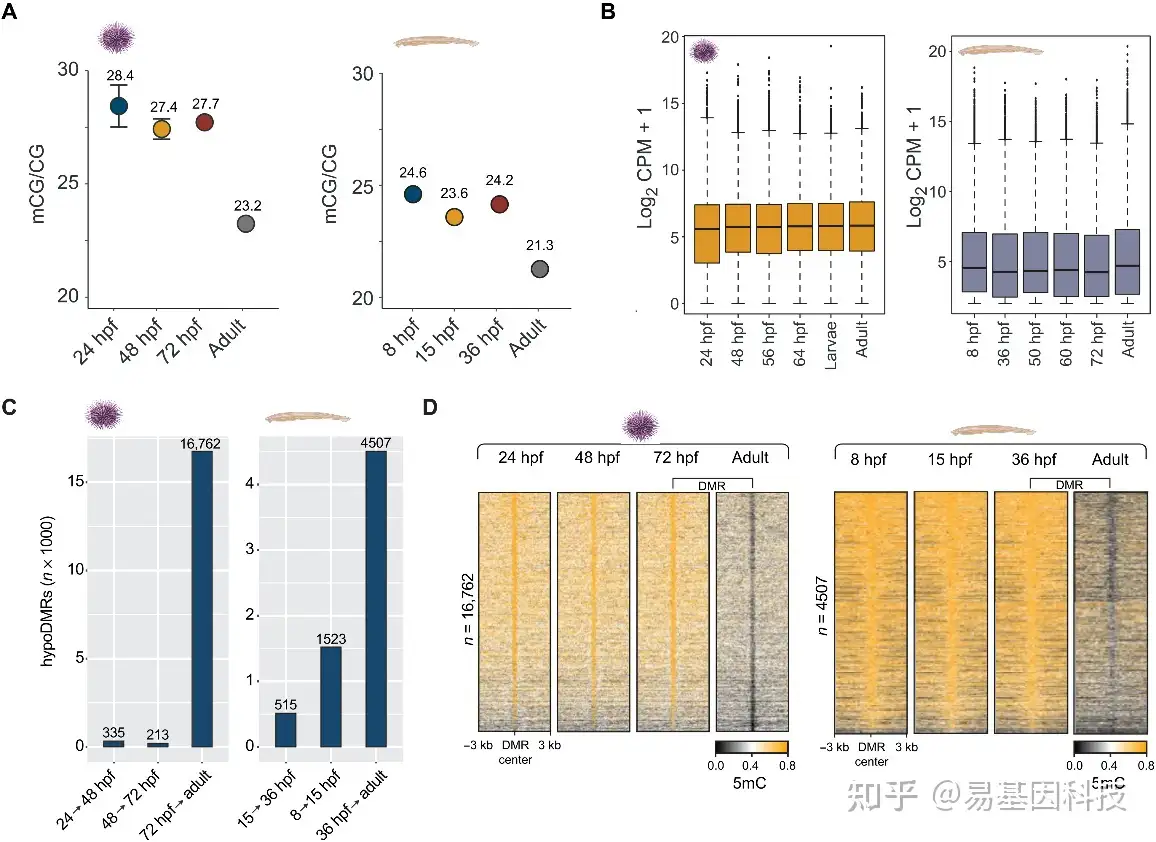
图2:海胆和文昌鱼发育过程中的DNA甲基化变化
(A) 海胆和文昌鱼发育过程中的平均DNA甲基化(mCG/CG)水平。
(B) 海胆和文昌鱼发育过程中的平均基因表达(CPM+1)水平。
(C) 海胆和文昌鱼基因组的胚胎和成体阶段之间鉴定的低甲基化DMR(hypoDMR)数量。
(D) 海胆和文昌鱼基因组中成体hypoDMR的DNA甲基化(mCG/CG)热图。
(3)ACE-seq和hMeDIP-seq揭示5hmC景观的发展动态变化
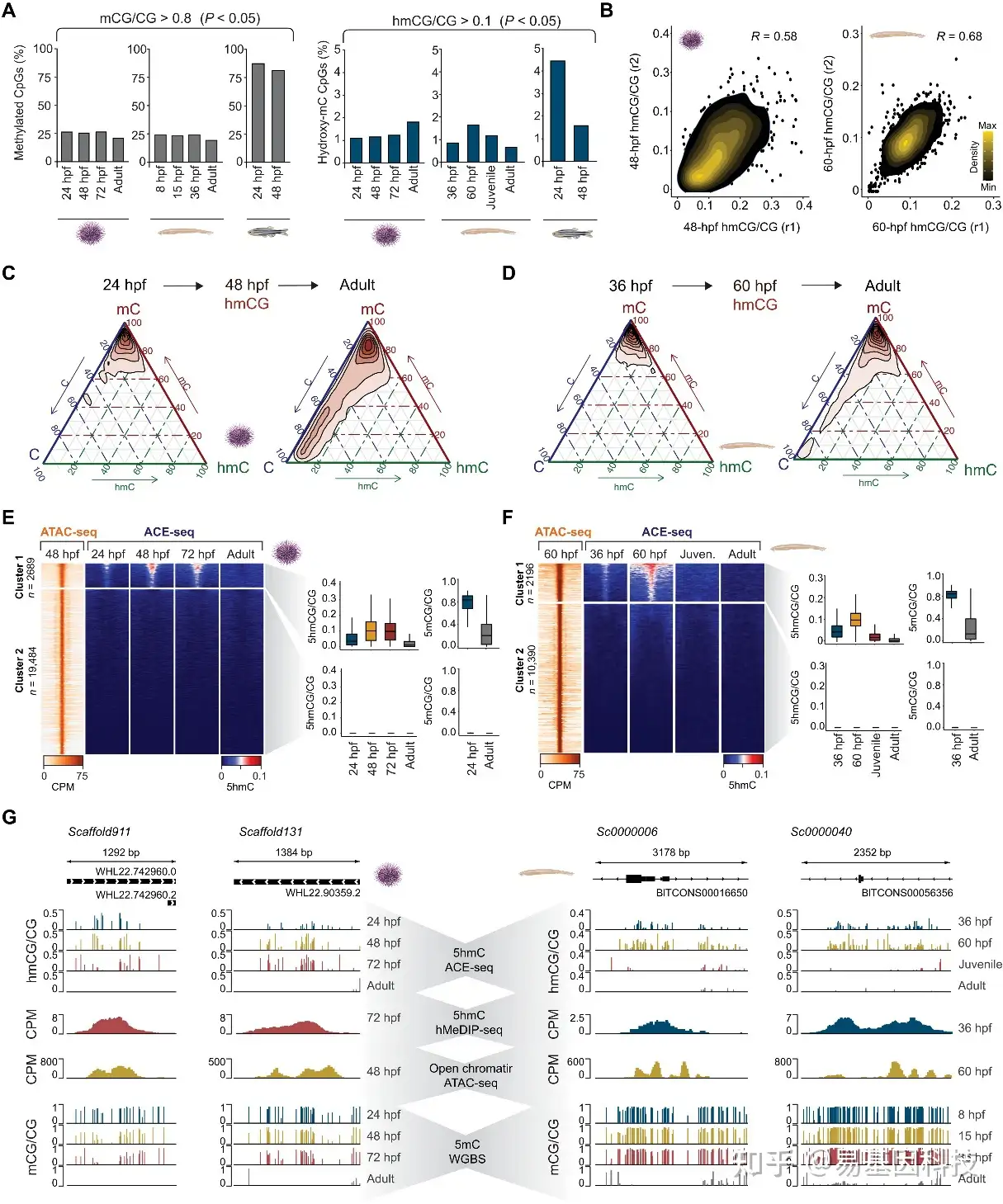
图3:海胆和文昌鱼发育的DNA羟甲基化景观
- 海胆、文昌鱼和斑马鱼的胚胎和成体组织中甲基化和羟甲基化CpG百分比。分析包括甲基化至少80% CpG位点和羟甲基化至少10% CpG位点(P adj.<0.05)。仅考虑覆盖率>10X的CpG位点。
- 海胆48hpf和文昌鱼60hpf样品生物学重复之间羟甲基化水平的一致性。
(C-D) 海胆(C)和文昌鱼(D)胚胎和成体组织中甲基化(mC)、羟甲基化(hmC)、未甲基化(C)CpG状态之间关系的三元图。图中显示海胆48hpf(C)和文昌鱼60hpf(D)羟甲基化的CpG位点(P<0.05)。
(E-F) 海胆(E)和文昌鱼(F)中开放染色质区域(ATAC-seq)的DNA羟甲基化变化(ACE-seq)热图。ACE-seq和ATAC-seq信号在ATAC-seq peaks上的K均值聚类(K=2)。海胆(E)和文昌鱼(F)发育过程中DNA羟甲基化和DNA甲基化变化箱形图。
(G) IGV浏览器跟踪描绘开放染色质区域(ATAC-seq)的DNA羟甲基化(ACE-seq和hMeDIP-seq)富集与海胆和文昌鱼基因组中的DNA去甲基化(WGBS)一致。
(4)ATAC-seq揭示5hmC与发育基因激活
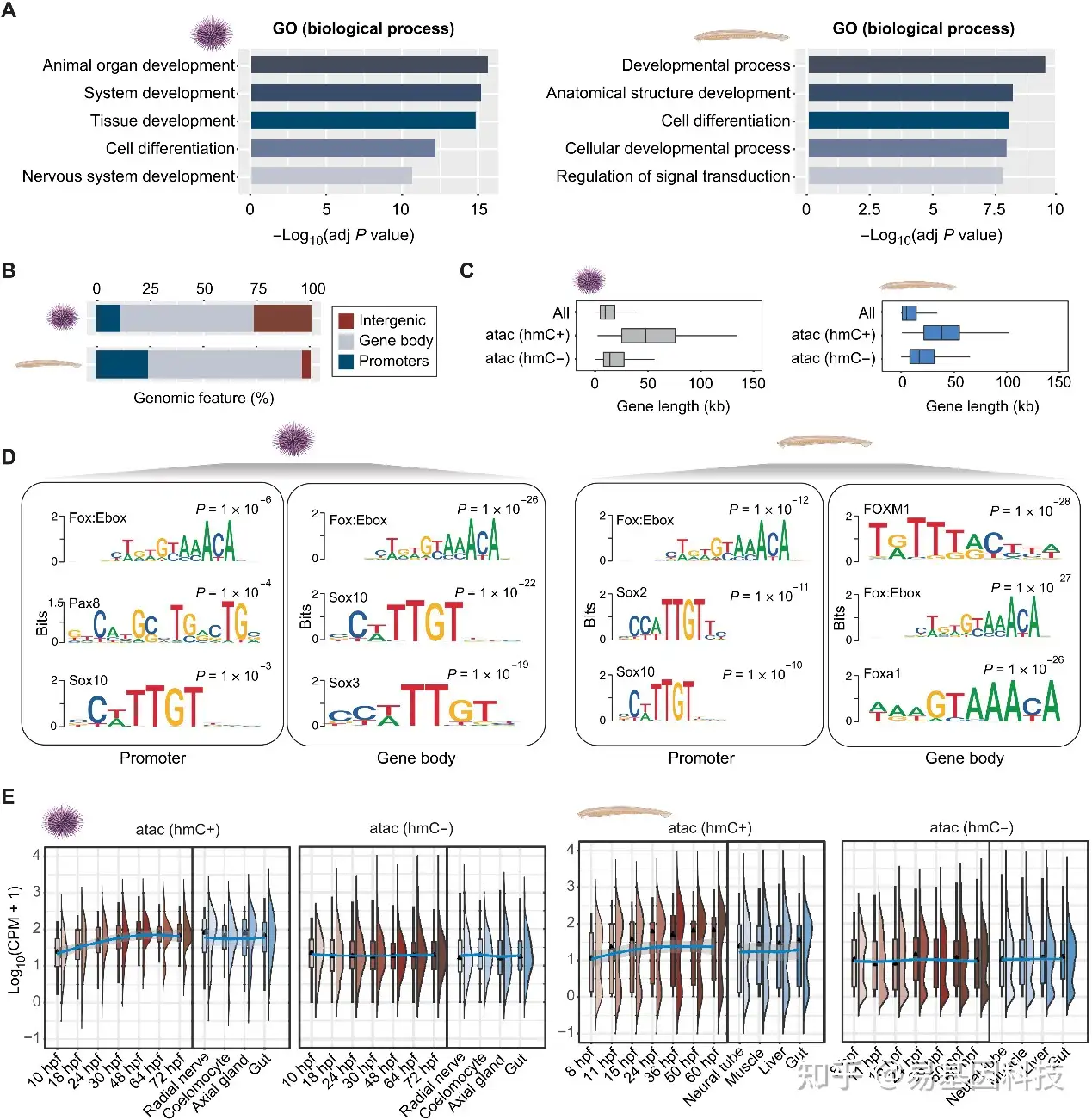
图4:5hmC标记基因的发育表达
(A) 海胆和文昌鱼在启动子和基因体区域5hmC标记的ATAC-seq peaks基因的GO分析。
(B) 不同基因组区域5hmC标记的ATAC-seq peaks百分比。
(C) 启动子相关5hmC标记的ATAC-seq peaks(启动子ATAC-hmC+)和非5hmC ATAC-seq peaks(启动子ATAC-hmC−)的基因长度。
(D) 与基因启动子或基因体相关的ATAC-hmC+区域的HOMER motif富集分析。
(E) 启动子ATAC-hmC+基因与启动子ATAC-hmC-基因在海胆和文昌鱼胚胎和成体组织中的表达变化。
(5)5hmC标记基因的保守调控机制
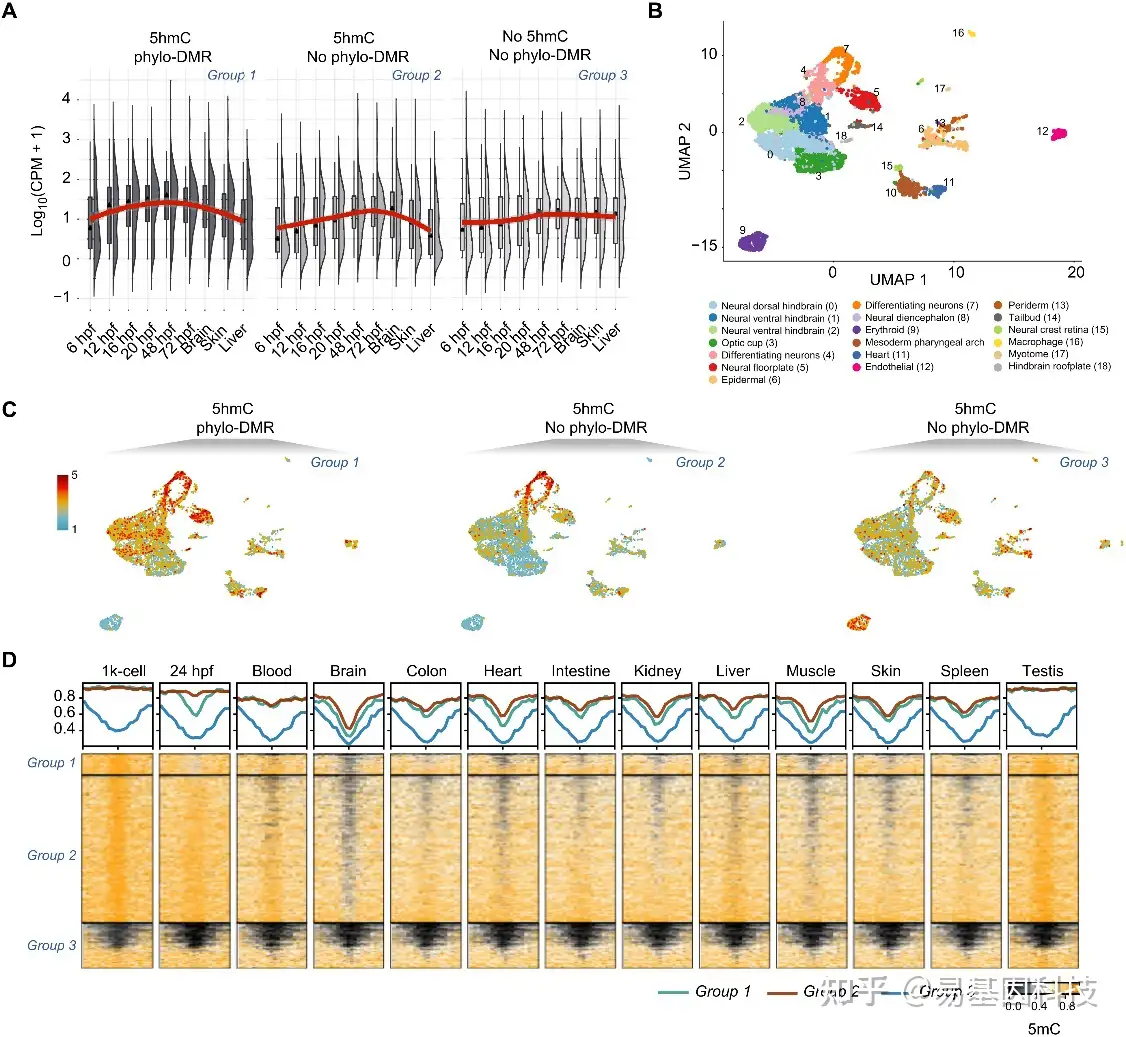
图5:斑马鱼5hmC标记基因的保守发育调控方式
(A) 2R/3R ohnolog斑马鱼在胚胎和成体组织中5hmC标记的ATAC-seq peaks与phylo DMR重叠区域(5hmC/phylo DMR)、5hmC标记的ATAC-seq peaks与非phylo DMR重叠区域(5hmC/No phylo DMR)、非5hmC标记的ATAC-seq peaks与非phylo DMR重叠区域(No 5hmC/ No phylo DMR)的基因表达。
(B) 24 hpf斑马鱼胚胎的7738个单细胞的UMAP投影。细胞按组织类型进行颜色编码。
(C) 24hpf斑马鱼胚胎的5hmC/phylo DMR、5hmC/No phylo DMR和No 5hmC/No phylo DMR三个基因组的z-scores得分。模块评分描述了每个基因组的平均基因表达与随机对照基因之间的差异。
(D) 1k细胞和24hpf斑马鱼胚胎以及多个成体组织中5hmC/phylo DMR、5hmC/No phylo DMR和No 5hmC/No phylo DMR ATAC-seq peaks的DNA甲基化谱热图。
结论:
本研究使用全基因组重亚硫酸盐测序(WGBS)和ACE-seq在海胆和文昌鱼四个发育时期分别生成5mC和5hmC的单碱基分辨率图谱。将这些数据集与开放染色质图谱(ATAC-seq)数据进行整合分析,表明在两种海胆和文昌鱼两种生物的调控区域DNA去甲基化显著,有力地支持了无脊椎动物后口动物使用TET蛋白和5hmC进行发育基因调控的观点。另外研究发现5hmC标记基因的脊椎动物同源性与5hmC富集有关,并定义了斑马鱼的时空调控方式。研究表明至少在一些无脊椎动物谱系中,5mC不仅可以预防假转录起始所需的基因活性,还可以作为调控标记在与关键发育过程相关的调控区域被积极重塑。总的来说,本研究为无脊椎动物-脊椎动物的5mC/5hmC调控逻辑提供了新见解,并证明在动物发育过程中从调控区域主动去除5mC早于脊椎动物起源。
关于易基因全基因组重亚硫酸盐测序(WGBS)技术
全基因组重亚硫酸盐甲基化测序(WGBS)可以在全基因组范围内精确的检测所有单个胞嘧啶碱基(C碱基)的甲基化水平,是DNA甲基化研究的金标准。WGBS能为基因组DNA甲基化时空特异性修饰的研究提供重要技术支持,能广泛应用在个体发育、衰老和疾病等生命过程的机制研究中,也是各物种甲基化图谱研究的首选方法。
易基因提供的全基因组甲基化测序技术通过T4-DNA连接酶,在超声波打断基因组DNA片段的两端连接接头序列,连接产物通过重亚硫酸盐处理将未甲基化修饰的胞嘧啶C转变为尿嘧啶U,进而通过接头序列介导的 PCR 技术将尿嘧啶U转变为胸腺嘧啶T。
应用方向:
WGBS广泛用于各种物种,要求全基因组扫描(不错过关键位点)
- 全基因组甲基化图谱课题
- 标志物筛选课题
- 小规模研究课题
技术优势:
- 应用范围广:适用于所有参考基因组已知物种的甲基化研究;
- 全基因组覆盖:最大限度地获取完整的全基因组甲基化信息,精确绘制甲基化图谱;
- 单碱基分辨率:可精确分析每一个C碱基的甲基化状态。
易基因科技提供全面的DNA甲基化研究整体解决方案,技术详情了解请致电易基因。
参考文献:
Skvortsova K, Bertrand S, Voronov D, Duckett PE, Ross SE, Magri MS, Maeso I, Weatheritt RJ, Gómez Skarmeta JL, Arnone MI, Escriva H, Bogdanovic O. Active DNA demethylation of developmental cis-regulatory regions predates vertebrate origins. Sci Adv. 2022 Dec 2;8(48):eabn2258.
相关阅读:
动物发育:DNA甲基化组与转录组综合分析绒山羊胚胎期毛囊发育的调控机制
