易基因|3文一览:表观遗传时钟(甲基化年龄)在衰老和肿瘤中的作用
大家好,这里是专注表观组学十余年,领跑多组学科研服务的易基因。
早在2018年,加州大学洛杉矶分校生物统计学家Steve Horvath研究发现:随着年龄的增加,某些基因的甲基化增加了,而另一些基因的甲基化会减少,处于浮动状态。在此基础上他设计开发了表观遗传时钟(Epigenetic clock),即利用基因的甲基化图谱来判断生理年龄,且精确度高达98%。本期我们通过3篇研究论文说说表观遗传时钟(或DNA甲基化年龄)的作用,它和衰老相关,也和癌症高度相关。
01
小鼠:表观遗传时钟与衰老

标题:Tick tock, tick tock: Mouse culture and tissue aging captured by an epigenetic clock.
期刊:Aging Cell
影响因子:IF 9.304
发表时间:2022.01.25
技术平台:RRBS
摘要
衰老与DNA甲基化的动态变化相关,尽管这种变化的前因后果尚不清楚,通过体外模型研究控制这些变化,可以从实验中大大提高揭示表观遗传衰老机制的能力。然而目前尚不清楚培养细胞引起的变化是否可以作为体内衰老组织中观察到的模型。为此作者对小鼠胚胎成纤维细胞(MEFs)传代培养并通过简化代表性重亚硫酸盐测序(reduced representation bisulfite sequencing,RRBS)评估每个阶段的DNA甲基化水平变化。此外,作者开发一种被称为“CultureAGE”的体外追踪细胞衰老方法,检测能否追踪各种小鼠组织的生理衰老,以及抗衰老干预能否调控这种追踪方法。结果显示,随着年龄的增长,多个组织(肝、肺、肾、血液和脂肪)中CultureAGE值显著增加。且通过对照验证CultureAGE值不是细胞衰老的标志物,表明CultureAGE揭示了一种可以在体外诱导的独特但渐进的细胞衰老现象。此外研究还证明,当MEFs重编程为诱导多能干细胞(iPSCs)时,在热量限制的动物中表观遗传衰老速度较慢。富集和聚类分析表明,EED和PcGs(Polycomb group)蛋白是翻译culture aging表型中潜在的重要染色质调控因子。总之,本研究证实可以体外诱导生理相关衰老变化,并揭示表观遗传衰老机制。
实验方法
在两只雌性小鼠妊娠第12.5天提取分离MEF细胞。将细胞进行分裂/传代培养六次,每次传代均进行流式细胞术/共聚焦显微镜和RRBS测序。通过RRBS在三个生物重复的每次传代中评估DNA甲基化变化。
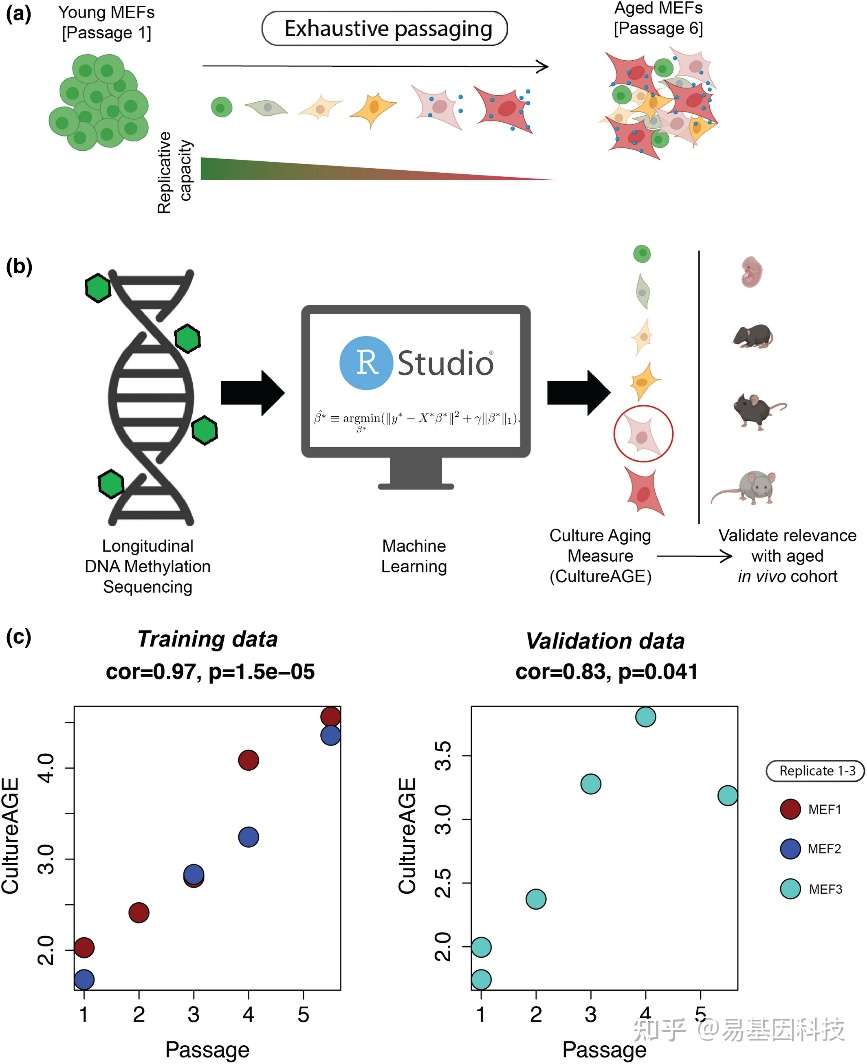
图1:在MEF中测定DNA甲基化CultureAGE值
(a)在常氧(20%O2)状态下对MEF培养,直至细胞终末停滞状态(细胞衰老),其复制能力逐渐降低。
(b)RRBS测序的DNA甲基化数据中产生CultureAGE,随后在衰老活体队列中进行生理相关性测试。
(c)红色=MEF1细胞系,蓝色=MEF2细胞系,绿色=MEF3细胞系重复验证。利用皮尔逊相关系数分析法确定通路相关性和统计显著性。
关键图形
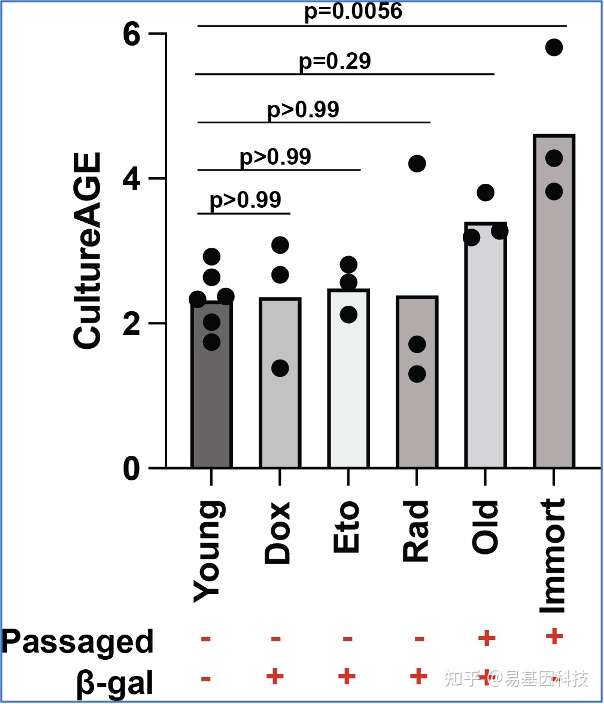 CultureAGE表型独立于细胞衰老表型,需要复制扩增
CultureAGE表型独立于细胞衰老表型,需要复制扩增
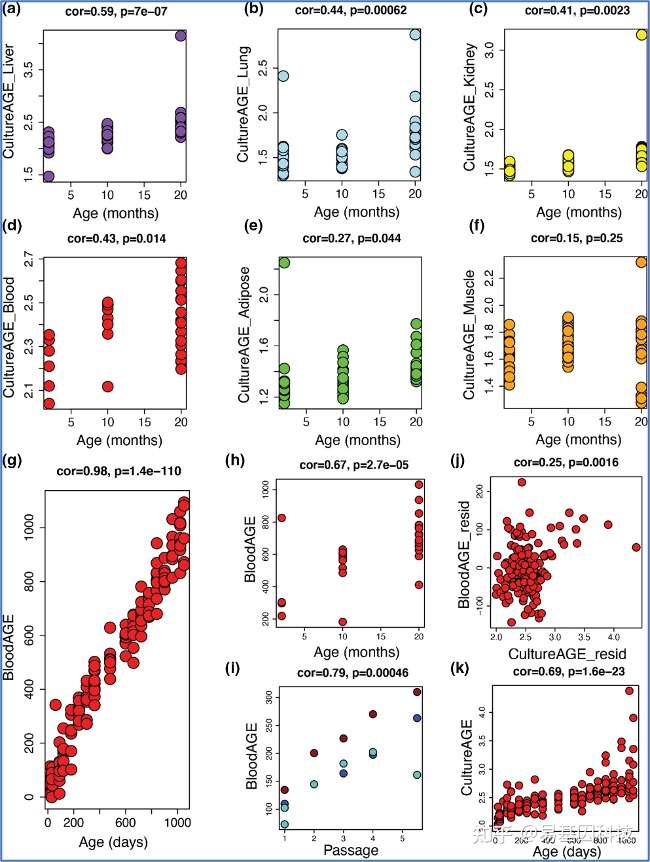 通过CultureAGE测定多组织生理衰老模式
通过CultureAGE测定多组织生理衰老模式
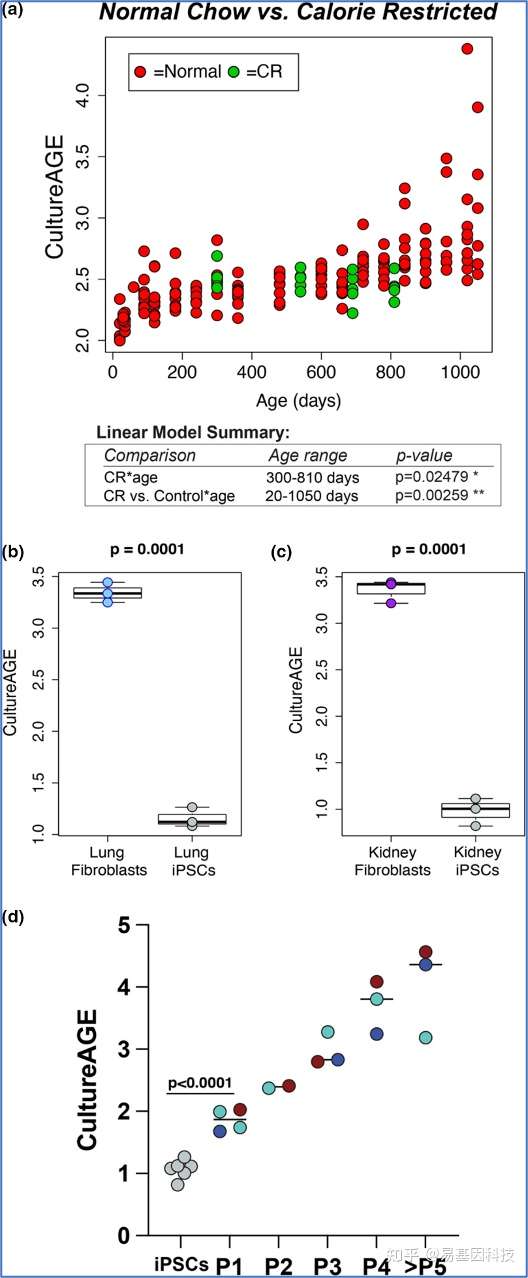 CultureAGE预测热量限制小鼠和重编程的MEF细胞培养状态
CultureAGE预测热量限制小鼠和重编程的MEF细胞培养状态
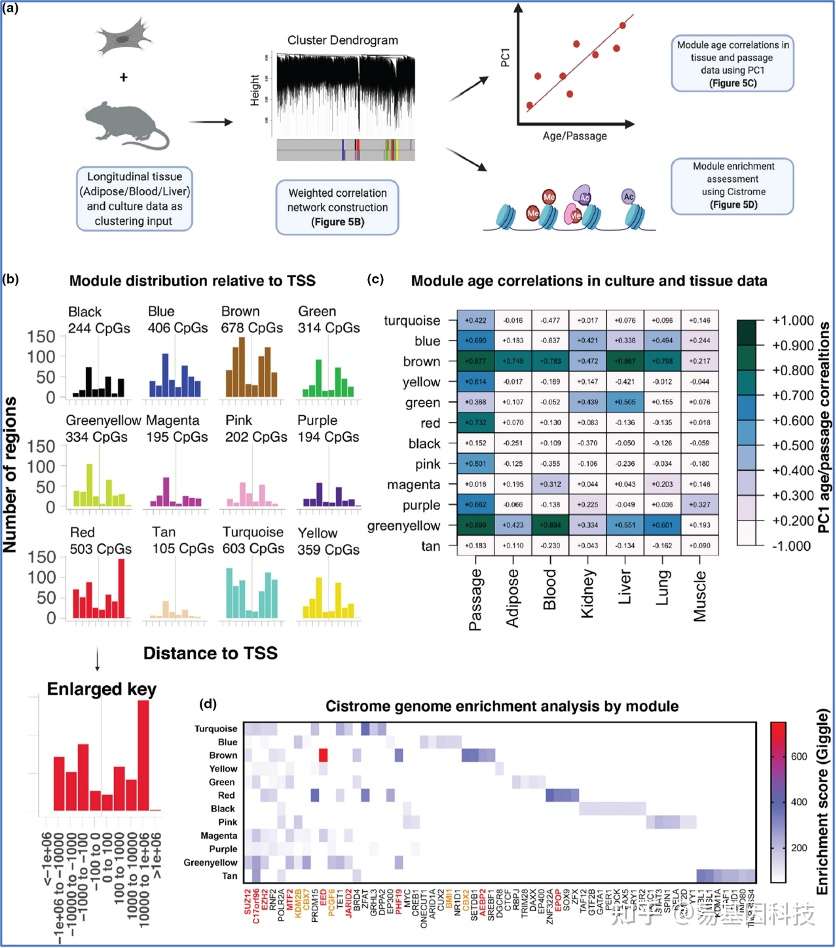 聚类分析证实culture aging存在于生理环境中,并强调PcG蛋白是重要的culture aging调控因子
聚类分析证实culture aging存在于生理环境中,并强调PcG蛋白是重要的culture aging调控因子
02
F344大鼠:表观遗传时钟与大颗粒淋巴细胞白血病
标题:Impact of Large Granular Lymphocyte Leukemia on Blood DNA Methylation and Epigenetic Clock Modeling in Fischer 344 Rats
期刊:J Gerontol A Biol Sci Med Sci
影响因子:IF 6.053
发表时间:2021.10.28
技术平台:RRBS
摘要
特定CpG位点的甲基化年龄依赖性差异已在“表观遗传时钟”公式中用以预测年龄。表观遗传年龄与实际年龄的偏差情况与不良健康结果相关,有助于了解健康状况。在大多数情况下,表观遗传时钟通过从循环血细胞中提取DNA进行甲基化水平评估。然而,表观遗传时钟对循环肿瘤细胞的影响尚不清楚。本研究使用常发生大颗粒淋巴细胞白血病(LGLL)的61只17-27个月大的Fischer 344(F344)大鼠为样本,检测在27只大鼠的脾脏和肝脏中发现明确的LGLL病理学标记。作者利用覆盖300万个CpG位点的简化甲基化测序(RRBS)来评估DNA甲基化。尽管LGLL广泛增加DNA甲基化的变异性,但并没有改变表观遗传衰老状态。将LGLL大鼠纳入时钟训练集,结果显著改变了121个常用CPG位点中的83个预测因子。此外在包含LGLL的大鼠样本上的训练模型比不含LGLL的训练模型具有更大的绝对年龄差(增加39%;p<0.0001)。表明衰老和LGLL的表观遗传学信号不同,因此LGLL评估不是F344大鼠中表观遗传年龄的必需有效测定,不过表观遗传时钟公式的精度和结构可能会受到训练集中肿瘤造血细胞的影响。
实验方法
该研究共使用162只雄性F344 CDF大鼠(1-27个月,每个月龄6只)。将大鼠单独饲养,可随意摄入标准的室内食物和水,并保持12小时开灯-12小时关灯循环。评估LGLL的大鼠在抽血后4周内进行安乐死,解剖内部器官并储存在中性缓冲的10%福尔马林中用于随后的病理学分析。此前研究表明在18月龄之前存在 LGLL 极为罕见,因此本研究只评估17-27月龄大鼠 (n = 61) 的 LGLL 病理学,并评估六只 6月龄大鼠和六只 12月龄大鼠的脾脏和肝脏病理学作为阴性对照。提取大鼠血液DNA进行简化甲基化测序(RRBS)和高性能统计分析。
结果
LGLL病理学
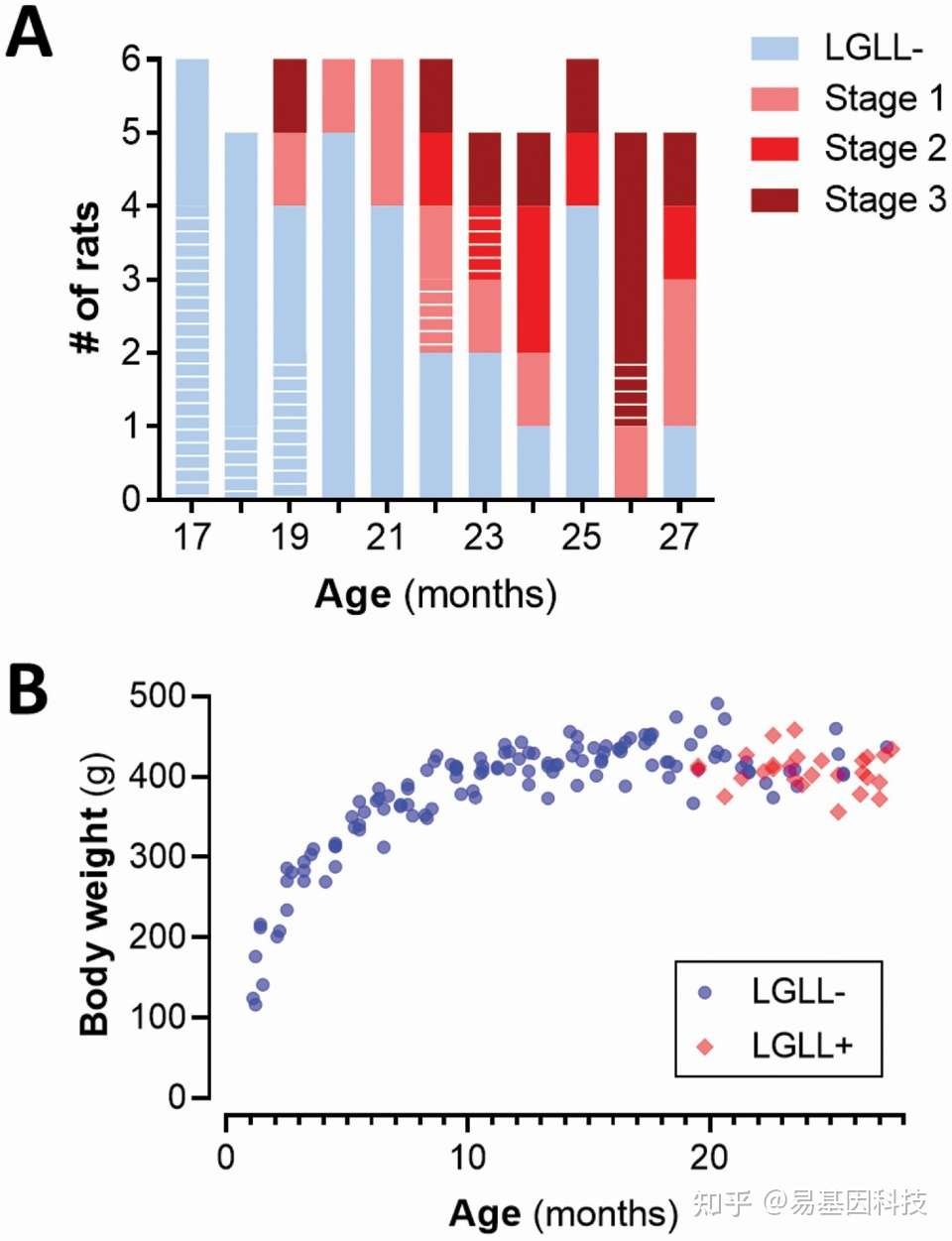 按年龄划分的大鼠 LGLL 患病率按年龄划分的大鼠 LGLL 患病率
按年龄划分的大鼠 LGLL 患病率按年龄划分的大鼠 LGLL 患病率
LGLL相关的DNA甲基化水平变化
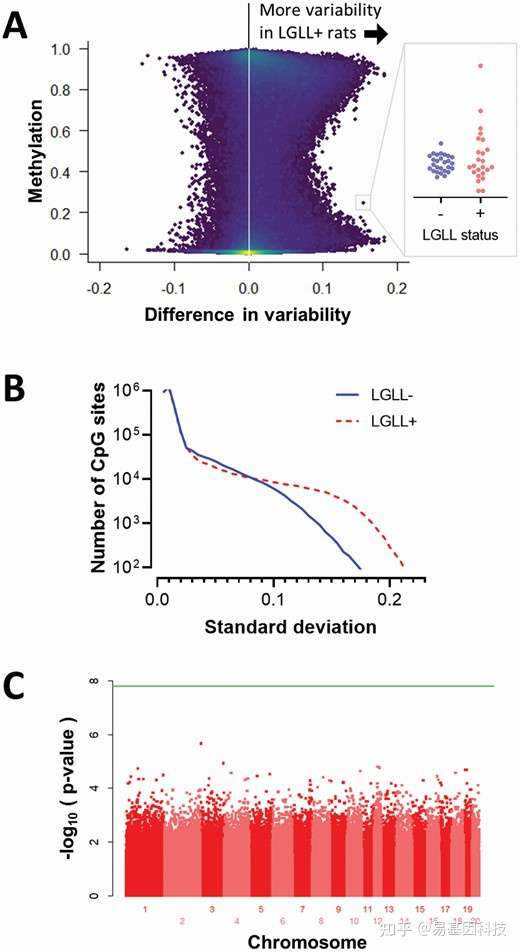 LGLL大鼠CpG甲基化的变异性增加
LGLL大鼠CpG甲基化的变异性增加
LGLL对表观遗传钟的影响
LGLL降低了表观遗传时钟精度,不会加速表观遗传衰老
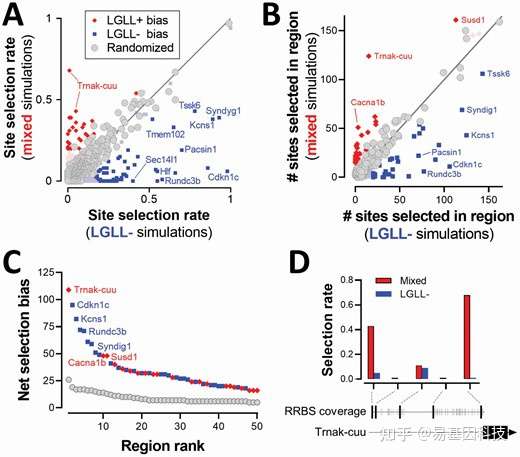
LGLL影响表观遗传时钟生成过程中的预测因子选择
03
人样本:乳腺癌与复制相关的表观遗传时钟

标题:DNA methylation landscapes of 1538 breast cancers reveal a replication-linked clock, epigenomic instability and cis-regulation.
期刊:nature communications
影响因子:IF 14.919
发表时间:2021.09.13
技术平台:RRBS
摘要
DNA甲基化在癌症中是异常的,但这种表观遗传学变化的动力学作用、调控作用和临床意义仍然知之甚少。
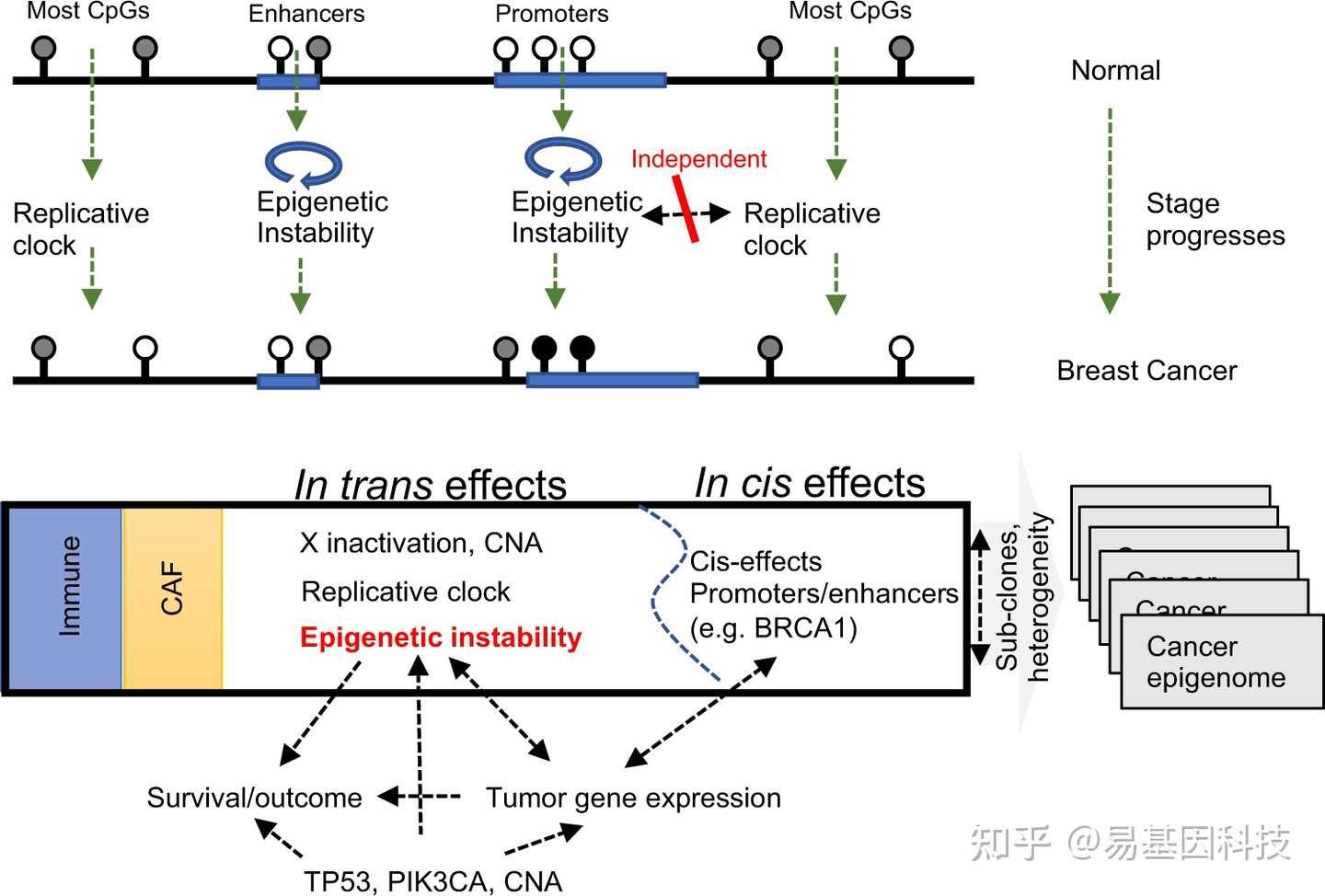
METABRIC队列中包含了2000多个乳腺癌样本,这些样本此前已在临床、遗传和转录方面进行了广泛表征。本文作者使用RRBS测序分析技术对其DNA甲基化状态进行研究。来自METABRIC队列的1538例乳腺癌组织和244个相邻正常乳腺组织的简化甲基化测序(RRBS)谱,在丰富的基因组、转录组和临床数据背景下对DNA甲基化进行深度分析。来自免疫和间质标记物的肿瘤DNA甲基化状态被反褶积(deconvoluted),从而导致在非CpG位点发现与全基因组甲基化丢失的肿瘤复制相关时钟(replication-linked clock)。出乎意料的是,在大部分CpG区域甲基化遵循两个独立于复制的获得(MG)或丢失(ML)过程,称之为表观基因组不稳定性,表观基因组不稳定性与肿瘤分级/分期、TP53突变和较差预后相关。另外,研究人员在数百个启动子和数千个远端元件中发现了顺式特异性甲基化(cis-specific methylation)和表达相关,包括一些已知的肿瘤抑制因子和致癌基因,证明了数百个启动子和数千个远端元件中的甲基化水平和特异性顺式作用下的基因表达相关,突出了全基因组甲基化水平变化在肿瘤转录改变中的重要作用,包括典型的BRCA1高甲基化效应。
实验方法
从METABRIC数据库中选取1538个原发性乳腺癌组织样本和244个相邻组织的正常样本,共1782个样本进行简化DNA甲基化测序分析(RRBS),并建立导致乳腺癌DNA甲基化过程的多因素统一模型。
结果
乳腺癌与复制相关的DNA甲基化时钟过程相关
1.METABRIC 队列的甲基化分析
从METABRIC队列中选取1538个原发性乳腺癌组织样本和244个相邻组织的正常样本,利用可获得的临床、基因组和转录组数据来分析甲基化过程(图1a),研究人员利用RRBS测序方法对这1782个样本以30.4B reads来覆盖广泛的基因组分布,有助于分析全基因组甲基化变化趋势及调控元件和启动子的甲基化变化。RRBS测序方法使93%的样本被超过1 M CpG位点的10个以上reads覆盖(图1b),9%的reads被映射到真正的启动子区域(图1c),75%的启动子区域平均覆盖超过20个reads(平均覆盖246个),有助于下游的定量分析(图1d)。
2.乳腺癌甲基化的分层建模
以METABRIC数据库为模型,研究人员开发了一种半监督算法(Methylayer),用于肿瘤甲基化动力学的分层建模(图1e)。Methylayer的基本原理依赖于基因表达、遗传学和临床信息的整合,计算混杂因素(肿瘤微环境[TME]效应),以此推断可随机影响全基因组甲基化变化趋势,Methylayer可以强有力地筛选表观遗传顺式调控的候选基因,并得出预后指标。将Methylayer分别应用于METABRIC队列的ER+和ER-肿瘤样本,并比较两类肿瘤样本的动力学。
数据结果发现,基因表达的整合使Methyllayer将TME效应识别为主要数据混杂因素,从而促进从肿瘤活检中获得的甲基化图谱多样化(图1f)。该算法在基因表达和启动子甲基化状态的相互关联中检测到一个强大的免疫标记,即TME标记与肿瘤分级相关(图1g),并通过独立的反褶积表达谱和病理指标进行验证。在对TME标记进行推断之后,研究人员应用了一种新的K-nn归一化算法(K-nn normalization algorithm Methods,图1h),该算法在推断下游肿瘤甲基化区域时,验证了Methyllayer显著降低了TME偏差。
3. 复制相关的甲基化时钟过程与肿瘤中甲基化丢失相关
与对照组相比,基于TME标准甲基化的Methyllayer聚类在肿瘤样本中鉴定出一组高度相关的CpG区域 (图1i),这一甲基化区域与肿瘤分级不相关(图1j),将其标记为时钟层(clock layer),尽管与启动子相关的远端定位相关,但时钟层CpG显示出较低的CpG含量(图1k),因此在推定的调控元件(基于组蛋白修饰)中代表性不足。
基因组通过定义早期和晚期复制域的调控过程在S期复制。有趣的是,在S晚期复制域中,时钟层的肿瘤甲基化减少更为强烈(图1l,m)。这与此前研究一致,表明衰老和癌症中DNA甲基化的丢失可能与复制过程中相关的甲基化异常积累(“epi-mutations”,表突变)相关。筛选跨METABRIC的基因表达特征并未发现与甲基化时钟层相关的常规转录程序。总之,这些数据共同表明癌症中甲基化丢失时钟的动力学与基因组复制过程密切相关。
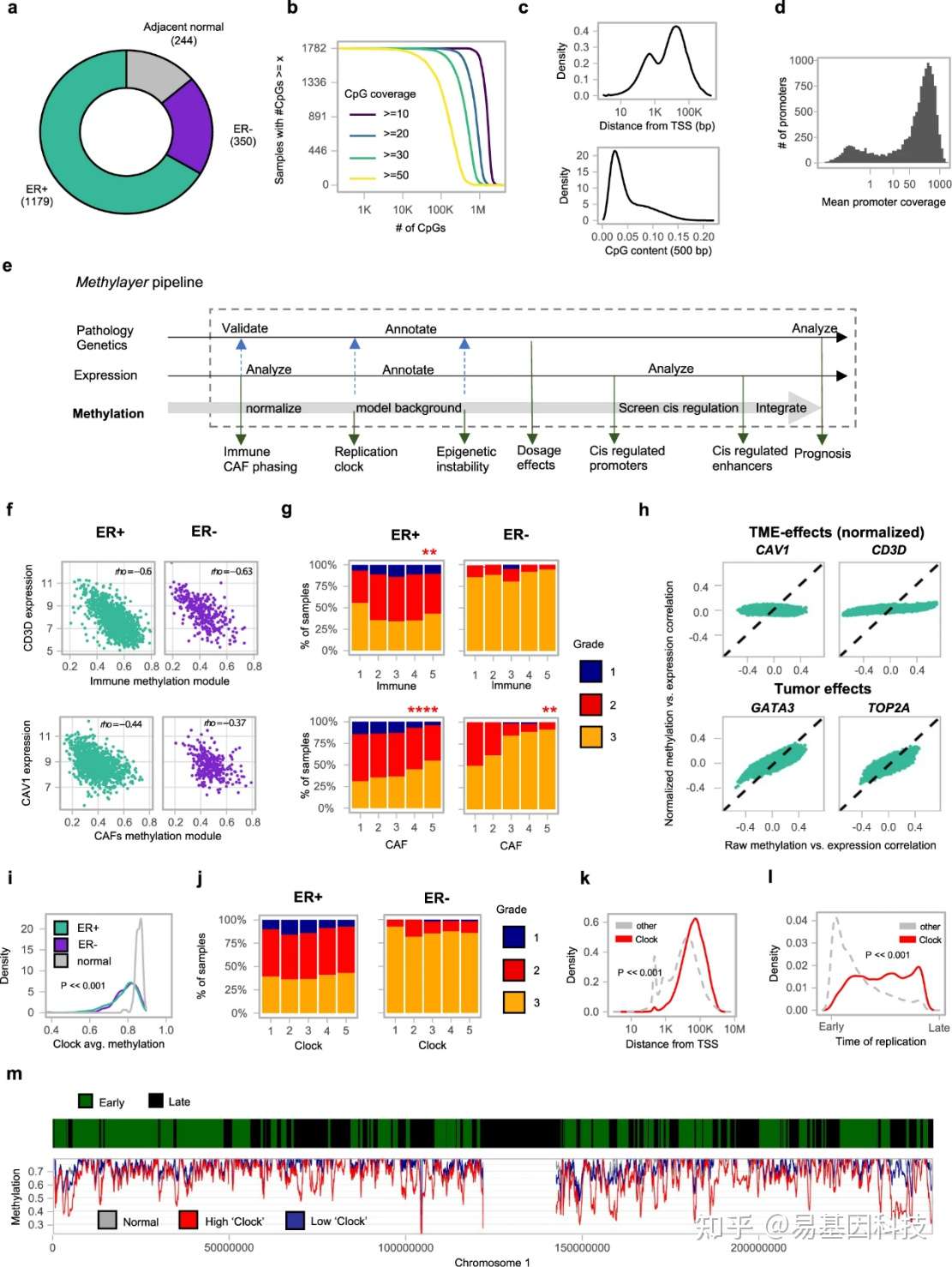 图: 在 METABRIC 队列中解剖肿瘤、免疫学和 CAF 甲基化
图: 在 METABRIC 队列中解剖肿瘤、免疫学和 CAF 甲基化
易基因小结:表观遗传时钟的作用
表观遗传时钟(epigenetic clock)可以根据DNA的特定变化(甲基化)来预测年龄,是预测衰老、疾病及死亡的个体化生物时钟。表观遗传时钟研究成果为比较表观基因组学的研究提供了大量资源,并且一系列基于此的新思路新方法将会诞生、拓展开来,成为开启表观遗传研究领域新世界大门的密匙。
关于3文共同采用的测序技术
— RRBS —
简化甲基化测序(Reduced Representation Bisulfite Sequencing,RRBS)是利用限制性内切酶对基因组进行酶切,富集启动子及CpG岛等重要的表观调控区域并进行重亚硫酸盐测序。该技术显著提高了高CpG区域的测序深度,在CpG岛、启动子区域和增强子元件区域可以获得高精度的分辨率,是一种准确、高效、经济的DNA甲基化研究方法,在大规模临床样本的研究中具有广泛的应用前景。为了适应科研技术的需要,易基因进一步开发了可在更大区域内捕获CpG位点的双酶切RRBS(dRRBS),可研究更广泛区域的甲基化,包括CGI shore等区域。
为助力低样本量多维度分析,易基因开发了富集覆盖CpG岛、启动子、增强子、CTCF结合位点的甲基化靶向测序方法:extended-representation bisulfite sequencing(XRBS),实现了高灵敏度和样本复用,使其具有高度可扩展性,并适用于有限的样本和单个细胞。
有DNA甲基化测序需求的老师可以联系我们哦

参考文献
DOI:10.1111/acel.13553
DOI:10.1093/gerona/glab328
DOI:10.1038/s41467-021-25661-w
相关阅读:
3文聚焦:DNA甲基化对果实成熟的重要作用(橙+番茄+草莓)




【推荐】国内首个AI IDE,深度理解中文开发场景,立即下载体验Trae
【推荐】编程新体验,更懂你的AI,立即体验豆包MarsCode编程助手
【推荐】抖音旗下AI助手豆包,你的智能百科全书,全免费不限次数
【推荐】轻量又高性能的 SSH 工具 IShell:AI 加持,快人一步
· winform 绘制太阳,地球,月球 运作规律
· AI与.NET技术实操系列(五):向量存储与相似性搜索在 .NET 中的实现
· 超详细:普通电脑也行Windows部署deepseek R1训练数据并当服务器共享给他人
· 【硬核科普】Trae如何「偷看」你的代码?零基础破解AI编程运行原理
· 上周热点回顾(3.3-3.9)