第4章 探索性数据分析(多因子与复合分析)
4.1、多因子
4.1.1 假设检验与方差检验
假设检验适用于(数据样本较小时)
方差检验适用于(数据样本较大时)
import numpy as np
import scipy.stats as ss
#生成一20个数的标准正态分布
norm_dist = ss.norm.rvs(size=20)
#检测norm_dist是否是正态分布,使用的方法是基于峰度和偏度的
print(ss.normaltest(norm_dist))
#结果:NormaltestResult(statistic=0.2025598777545946, pvalue=0.9036800223028876)
#第一个是统计值,第二个值是p值
(1)P分布检验常用于比较两种样本是否一致(例如:临床医疗上药物是否有效);
(2)独立分布t检验用于检测两组值的均值是都有比较大的差异性

print(ss.ttest_ind(ss.norm.rvs(size=10),ss.norm.rvs(size=20)))
#结果:Ttest_indResult(statistic=-0.575484958550556, pvalue=0.5695598474341583)
由于p值大于0.05(假定),可以接受该假设
(3)卡方检验常常用于确定两因素件是否有比较强的联系


import scipy.stats as ss
print(ss.chi2_contingency([[15,95],[85,5]]))
#结果:(126.08080808080808, 2.9521414005078985e-29, 1, array([[55., 55.],[45., 45.]]))
#第一个值为卡方值,第二个值为P值,第三个值为自由度,第四个为与原数据数组同维度的对应理论值
由于p值小于0.05(假定),拒绝该假设
(4)F检验常用于方差分析
检测统计量F,做假设检验【F满足自由度(m-1,n-m)的F分布】
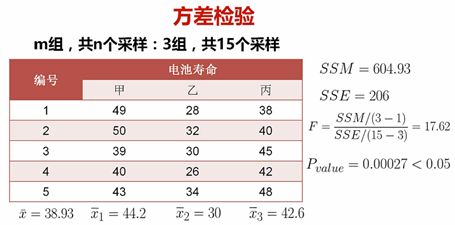
SST:总变差平方和
SSM:平均平方和(组间平方和)
SSE:残差平方和(组内平方和)
print(ss.f_oneway([49,50,39,40,43],[28,32,30,26,34,],[38,40,45,42,48]))
#结果:F_onewayResult(statistic=17.619417475728156, pvalue=0.0002687153079821641)
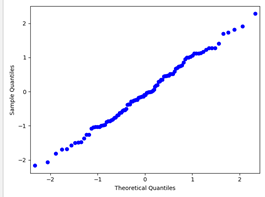
(5)使用qq图来对比一个分布是否属于一个已知的分布
方法:看散点图的是否在x,y轴的角平分线上
from statsmodels.graphics.api import qqplot import matplotlib.pyplot as plt qqplot(ss.norm.rvs(size=100)) plt.show()
4.1.2 相关系数
范围在[-1,1]----皮尔逊、斯皮尔曼(只与名次差有关,与具体数值相关性不强,故适用于相对比较的情况下)
衡量两组数据(或者两组样本)的分布趋势、变化趋势、一致趋势程度的因子,分为正相关(正向同步),负相关(反向同步)等。

其中:分子是协方差,分母是x,y的标准差,两者相乘相当于一个归一化因子

其中:n表示每组数据的数量,d值得是两组数据排名后的名次差

import pandas as pd
x = pd.Series([2.5,0.5,2.2,1.9,3.1,2.3,2,1,1.5,1.1])
y = pd.Series([2.4,0.7,2.9,2.2,3.0,2.7,1.6,1.1,1.6,0.9])
data = pd.DataFrame({'x':x,'y':y})
print('data:','\n',data)
print('Pearson相关系数:',x.corr(y))
print('Spearman相关系数:',x.corr(y,method='spearman'))
#Pearson相关系数: 0.9259292726922455
#Spearman相关系数: 0.9179373709568976
#DataFrame是针对于列进行相关性计算的,故需要对其进行转置
df = pd.DataFrame(np.array([x,y]).T)
print(df.corr())
'''结果
0 1
0 1.000000 0.925929
1 0.925929 1.000000
'''
4.1.3 回归
线性回归
线性回归最常用的是解法:最小二乘法,其本质是最小化误差平方


线性回归效果判定的关键指标:
决定系数越接近于1说明效果越好;
DW的范围在【0 , 4】,DW=2时表示残差不相关,接近于4表示正相关,接近于0表示残差负相关,好的回归一般残差是不相关的。
其中决定系数中的左R2:一元决定系数的定义;
右R2:多元决定系数的定义;
k:参数的个数
e:残差(预测值与实际值的差)
import numpy as np
import scipy.stats as ss
x = np.arange(10).astype(np.float).reshape((10,1))
y =x*3+4+np.random.random((10,1))
print('实际值:','\n',y)
from sklearn.linear_model import LinearRegression
reg = LinearRegression()
res = reg.fit(x,y)
y_pre = reg.predict(x)
print('预测值:','\n',y_pre)
score = reg.score(x,y)
print('效果:',score)
a = reg.coef_
b = reg.intercept_
print('參數:',a,';截距:',b)
import seaborn as sns
import matplotlib.pyplot as plt
plt.figure(figsize=(10,8))
plt.plot(x,y)
plt.plot(x,y_pre)
plt.show()

4.1.4 主成分分析(PCA)与奇异值分解(SVD)
主成分分析(PCA)步骤
· 求特征协方差矩阵
· 求协方差的特征值和特征向量
· 将特征值从大到小排序,选择其中最大的k个
· 将样本点投影到选取的特征向量上

自写版本(按照PCA步骤):
特别说明:np.linalg.eig(cov)之后的特征向量已按照对应的特征值从大至小进行排列
numpy.linalg模块包含线性代数的函数。使用这个模块,可以计算逆矩阵、求特征值、解线性方程组以及求解行列式等
eigvals函数可以计算矩阵的特征值,而eig函数可以返回一个包含特征值和对应的特征向量的元组
#-----特别说明:np.linalg.eig(cov)之后的特征向量已按照对应的特征值从大至小进行排列 import numpy as np import pandas as pd x=[2.5,0.5,2.2,1.9,3.1,2.3,2,1,1.5,1.1] y=[2.4,0.7,2.9,2.2,3.0,2.7,1.6,1.1,1.6,0.9] data = pd.DataFrame({'x':x,'y':y}) print('data:','\n',data) cov = data.cov() print('协方差矩阵:','\n',cov) #求协方差矩阵的特征值和特征向量 #numpy.linalg模块包含线性代数的函数。使用这个模块,可以计算逆矩阵、求特征值、解线性方程组以及求解行列式等 #eigvals函数可以计算矩阵的特征值,而eig函数可以返回一个包含特征值和对应的特征向量的元组 eigen = np.linalg.eig(cov) print(eigen) eigenvalues = eigen[0] print('eigenvalues:','\n',eigen[0]) #选取比较大的特征值所代表的特征向量 eigenvectors = eigen[1] print('选取的特征值:','\n',eigen[0][1]) print('选取的特征向量:','\n',eigen[1][0]) jian = data-np.mean(data) pcavectors = np.dot(jian,eigen[1][0]) print('主成分分析后的向量','\n',pcavectors)
自写版本(使用函数按照PCA步骤):
import numpy as np
import pandas as pd
def myPCA(data,n_components =1000000000):
mean_vals = np.mean(data,axis=0)
mid = data - mean_vals
#此处需要针对列进行协方差计算
cov_mat = np.cov(mid,rowvar=False)#rowvar默认为True,即对行求协方差
from scipy import linalg
eig_vals,eig_vects = linalg.eig(np.mat(cov_mat))
#取出最大的特征值所对应的特征向量
eig_val_index = np.argsort(eig_vals)#argsort()得到排序之后的索引
eig_val_index = eig_val_index[:-(n_components+1):-1]
eig_vects = eig_vects[:,eig_val_index]
low_dim_mat = np.dot(mid,eig_vects)
return low_dim_mat,eig_vals
x=[2.5,0.5,2.2,1.9,3.1,2.3,2,1,1.5,1.1]
y=[2.4,0.7,2.9,2.2,3.0,2.7,1.6,1.1,1.6,0.9]
data = pd.DataFrame([x,y]).T
print(myPCA(data,n_components=1))
接口版本:
from sklearn.decomposition import PCA import numpy as np import pandas as pd x=[2.5,0.5,2.2,1.9,3.1,2.3,2,1,1.5,1.1] y=[2.4,0.7,2.9,2.2,3.0,2.7,1.6,1.1,1.6,0.9] data = pd.DataFrame([x,y]).T #降维 lower_dim = PCA(n_components=1) #降为1维 new = lower_dim.fit(data) print(new) #PCA(copy=True, iterated_power='auto', n_components=1, random_state=None, #svd_solver='auto', tol=0.0, whiten=False) #copy如果为False,则传递给fit的数据将被覆盖并且运行fit(X).t important = lower_dim.explained_variance_ratio_ print(important) #[0.96318131],说明得到了96.3%的信息量 newdata = lower_dim.transform(data) print(newdata) #转化后的数值
注意:sklearn中所用到的PCA方法是奇异值分解(SVD),与上述PCA的步骤不一样,所以两者所得出的结论会有些许不同
4.2、复合分析
4.2.1交叉分析
除了纵向分析横向分析外,可以使用交叉分析来分析数据属性和属性的关系
sns.heatmap()用于做热图-----两个不同属性间相互影响的情况!
import numpy as np
import pandas as pd
import scipy.stats as ss
import matplotlib.pyplot as plt
import seaborn as sns
df = pd.read_csv("HR.csv")
##查看各个部门的离职之间是否有明显差异----使用独立t检验方法
##得到各个部门的离职分布
#得到分组后的索引值
dp_indices = df.groupby(by = 'department').indices
# print(dp_indices)
sales_values = df["left"].iloc[dp_indices['sales']].values
technical_values = df["left"].iloc[dp_indices['technical']].values
print(ss.ttest_ind(sales_values,technical_values))
#求两两部门间的p值
dp_keys = list(dp_indices.keys())
print(dp_keys)
dp_t_mat = np.zeros([len(dp_keys),len(dp_keys)])
for i in range(len(dp_keys)):
for j in range(len(dp_keys)):
p_values = ss.ttest_ind(df['left'].iloc[dp_indices[dp_keys[i]]].values,
df['left'].iloc[dp_indices[dp_keys[j]]].values)[1]
if p_values<0.05:
dp_t_mat[i][j] = -1
else:
dp_t_mat[i][j] = p_values
sns.heatmap(dp_t_mat,xticklabels=dp_keys,yticklabels=dp_keys)
plt.show()
#只有为黑色的,两个部门的才有显著性差异

# 采用透视表
piv_tb = pd.pivot_table(df,values='left',index=['promotion_last_5years','salary'],
columns=['Work_accident'],aggfunc=np.mean)
print(piv_tb)
sns.heatmap(piv_tb,vmin=0,vmax=1,cmap=sns.color_palette("Reds",n_colors=256))#指定最大值为1,最小值为0
plt.show()

颜色越深,离职率越高
4.2.2分组与钻取
钻取是改变数据维度层次,变换分析粒度的过程,可分为向下和向上钻取。
连续属性在分组前需要进行离散化。
连续分组:
· 分割(一阶差分)、拐点(二阶差分)
· 聚类
· 不纯度(Gini系数)


#求Gini系数
s1 = pd.Series(['X1','X1','X2','X2','X2'])
s2 = pd.Series(['Y1','Y1','Y1','Y2','Y2'])
#求平方和:
def getProbSS(s):
if type(s)!= pd.core.series.Series:
s = pd.Series(s)
prt_ary = s.groupby(by=s).count().values/float(len(s))
return 1- sum(prt_ary**2)
def getGini(s1,s2):
d = dict()
for i in list(range(len(s1))):
d[s1[i]] = d.get(s1[i], []) + [s2[i]]
return 1 - sum([getProbSS(d[k]) * len(d[k])/float(len(s1)) for k in d])
print("Gini:",getGini(s1,s2))
#结果:Gini: 0.7333333333333334
sns.barplot(x='salary',y='left',hue='department',data=df) plt.show()

#拐点(二阶差分) sl_s = df['satisfaction_level'] sns.barplot(list(range(len(sl_s))),sl_s.sort_values()) plt.show()

4.2.3相关分析
#heatmap()会自动屏蔽离散化信息。例如,salary有low、medium、high
sns.heatmap(df.corr(),vmin=-1,vmax=1,cmap=sns.color_palette("RdBu",n_colors=128))
plt.show()

对离散属性相关性的处理:
利用熵来进行离散属性相关性的计算
熵:用于衡量不确定性,条件熵不具有对称性


s1 = pd.Series(['X1','X1','X2','X2','X2','X2'])
s2 = pd.Series(['Y1','Y1','Y1','Y2','Y2','Y2'])
def getEntropy(s):
if type(s)!= pd.core.series.Series:
s = pd.Series(s)
prt_ary = s.groupby(by=s).count().values/float(len(s))
return (-(np.log2(prt_ary))*prt_ary).sum()
#条件熵需要注意
def getCondEntropy(s1,s2):#s1条件下s2的熵
d = dict()
for i in list(range(len(s1))):
d[s1[i]] = d.get(s1[i],[])+[s2[i]]
#{'X1': ['Y1', 'Y1'], 'X2': ['Y1', 'Y2', 'Y2', 'Y2']}
return sum([getEntropy(d[k]) * len(d[k])/float(len(s1)) for k in d])
def getEntropyGain(s1,s2):
return (getEntropy(s2) - getCondEntropy(s1,s2))
def getEntropyGainRatio(s1,s2):
return getEntropyGain(s1,s2)/getEntropy(s2)
import math
def getCoor(s1,s2):
return getEntropyGain(s1,s2)/math.sqrt(getEntropy(s1)*getEntropy(s2))
print('熵:',getEntropy(s2))
print('条件熵:',getCondEntropy(s1,s2))
print('熵增益:',getEntropyGain(s1,s2))
print('熵增益率:',getEntropyGainRatio(s2,s1))
print('相关性:',getCoor(s1,s2))
'''
结果:
熵: 1.0
条件熵: 0.5408520829727552
熵增益: 0.4591479170272448
熵增益率: 0.5
相关性: 0.4791387674918639
'''
4.2.4因子分析
探索性因子分析:通过协方差矩阵、相关性矩阵等指标分析多元属性变量的本质结构,并可以进行转化、降维等操作,得到影响目标属性的最主要因子。
验证性因子分析:验证一个因子与关注的属性之间是否有一定的关联,有何关联。

from sklearn.decomposition import PCA my_pca = PCA(n_components=7) #拟合时不允许出现离散型数据,drop默认情况下从行进行删除 low_mat = my_pca.fit_transform(df.drop(labels=['salary','department','left'],axis=1)) #通过PCA降维后信息的准确性
print("Ratio:",my_pca.explained_variance_ratio_) sns.heatmap(pd.DataFrame(low_mat).corr(),vmin=-1,vmax=1,cmap = sns.color_palette("RdBu",n_colors=128)) plt.show() #结果:Ratio: [9.98565340e-01 8.69246970e-04 4.73865973e-04 4.96932182e-05 2.43172315e-05 9.29496619e-06 8.24128218e-06]

通过PCA,将原来的特征空间变成了一个正交的特征空间
4.2.5聚类分析、回归分析(暂略)
可参考作者另一篇作文【面试问题汇总 - 大脸猫12581 - 博客园 (cnblogs.com)第7部分与第8部分】







