植被指数第二弹
植被指数第二弹—窄带绿度(Narrowband Greenness )#
窄带绿度指数对叶绿素含量、叶子表面冠层、叶聚丛、冠层结构非常敏感。
它的计算,采用的是红色与近红外区域部分—红边,红边是介于690 nm ~ 740 nm之间的区域;相比之下,窄带绿度指数比宽带绿度指数,对植被信息的反映更加敏感(尤其是茂密植被)。
它的计算离不开光谱导数,emm这个就粗略一提,不展开讲了;因为我不会...😮💨
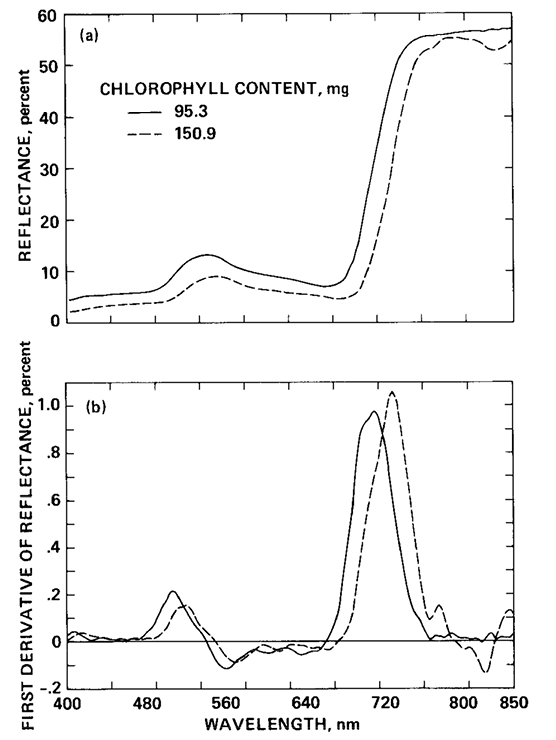
导数分光光度法:利用以吸光度对波长(波数)的导数为纵坐标,以波长(波数)为横坐标所记录的导数光谱进行测定的一种光度分析技术。
光谱数据求导是常用的近红外光谱预处理方法,可以消除基线漂移、提高光谱分辨率。一阶导可以去除常数基线(?),二阶导数可消除一次函数基线(线性函数),三阶导数可以去除二次函数基线。峰形曲线的一阶导数有2个峰,二阶导数为3个峰,三阶导数则有4个峰。但光谱求导会增加噪声水平,降低光谱信噪比。
光谱一阶导数的求导算法:差分法、Savitzky-Golay求导法、小波变换法。
下面看具体的窄带绿度指数:
- 植被指数第二弹—窄带绿度(Narrowband Greenness )
- 1 REP(Red Edge Position Index)
- 2 MTCI(MERIS terrestrial chlorophyll index)
- 3 Vogelmann指数
- 4-1 NDREI(Normalized Difference Red Edge Index)
- 4-2 NDVI705(Red Edge Normalized Difference Vegetation Index)
- 5-1 mSR705(Modified Red Edge Simple Ratio Index)
- 5-2 mNDVI705(Modified Red Edge Normalized Difference Vegetation Index)
1 REP(Red Edge Position Index)#
植被吸收-反射光谱在红-近红外有着丰富的信息,红光附近有一个吸收峰,而到了近红外反射率急剧上升;有人认为红光附近的吸收峰和叶绿素浓度相关,而近红外附近反射率急剧上升与叶片内部散射有关[1]。而当叶绿素浓度增加,吸收特征变宽,红边向长波段方向移动。
要算的,就是在690 nm ~ 740 nm光谱范围内,反射率导数最大(光谱一阶导的高峰)对应的波长。常用于农作物监测、估产、环境胁迫,生态系统干扰探测,光合作用模型,和由气候或其他因素产生的冠层胁迫性。
和对应的其实还有, Red Well Position红井位置[2],对应的是叶绿素的吸收峰(反射谷)。
找到这个斜率最大(拐点)的反射率,主要思路是曲线拟合。而这部分曲线拟合有下面几种方法[2]:
①线性插值
这一方法,1988由法国的G Guyot和F Baret提出[3](原文是法文,根本看不懂😂)。从一篇引用文献来看,他们做了两个假设:①假设波长670nm到780nm的反射曲线是直线。②红边位置在700-740nm之间。计算思路是:首先算出红边位置的反射率,再根据反射率得出红边位置。
②多项式拟合
这个算法的思路是用一个五次多项式,去拟合红波段反射谷到近红外“肩点”反射这部分反射率曲线。
表示Hyperion 661.45-783.48nm的13个波段。
③拉格朗日插值法
1998年,Dawson和Curran基于三点拉格朗日插值法,确定红边位置,并且将它和叶绿素浓度联系起来,并认为该方法可以很好地监测到红边的移动。该方法以光谱反射率一阶导为基础,采用一个二阶多项式曲线去拟合三个一阶导数波段,得出。然后对拉格朗日方程求二阶导,以确定最大斜率(红边)位置。
其中,,,是红边附近波长反射率的一阶导。
④逆高斯拟合
其中,是红边反射率的最高值(肩点),和是反射率的最低值和红井位置(REP,Red Well Postion)。是波长,是高斯分布的方差。红遍位置计算公式为:。
2 MTCI(MERIS terrestrial chlorophyll index)#
MERIS是Envisat卫星(欧空局的对地观测卫星系列之一,于2002年3月1日发射,2012年4月8日,与地球失去联系)搭载的传感器之一。有论文发现在高叶绿素浓度地区,可采用MERIS 8、9、10波段构建MTCI,来更好地指示叶绿素浓度。
高叶绿素浓度地区,REP与叶绿素浓度之间是单调关系,而此时,MTCI与叶绿素浓度呈线性关系。

中等分辨率成像光谱仪(MERIS, Medium Resolution Imaging Spectrometer)陆地叶绿素指数MTCI
3 Vogelmann指数#
1993年,Vogelmann提出[4]。主要针对高光谱数据,如VIRIS(Visible/Infrared Intelligent Spectrometer)。当叶绿素浓度降低,红边位置蓝移,(680-750nm)钟形曲线向三角趋近绿峰。
3-1 VOG1(Vogelmann Red Edge Index 1)#
Vogelmann红边指数1(REIP,Red Edge Inflection Point)
3-2 VOG2(Vogelmann Red Edge Index 2)#
Vogelmann红边指数2
3-3 VOG3(Vogelmann Red Edge Index 3)#
Vogelmann红边指数3
上面的红边位置等指数基于高光谱分辨率,而Sentinel-2中也纳入了红边波段,下面的指数的计算可以很好地通过三个波段反映植被特征。归一化差异红色边缘指数和红边归一化植被指数类似,具体应用中,各位各自甄别挑选。两者和形式类似,但和相比,充分利用高光谱分辨率数据(AVIRIS),更能体现叶绿素含量。
下面是Sentinel-2红边波段信息,本文采用Sentinel-2作为例子,给出公式与代码。
| 波段 | Sentinel-2A 中心波长(nm) | Sentinel-2B 中心波长(nm) | 空间分辨率(m) |
|---|---|---|---|
| 5-Vegetation red edge | 704.1 | 703.8 | 20 |
| 6-Vegetation red edge | 740.5 | 739.1 | 20 |
| 7-Vegetation red edge | 782.8 | 779.7 | 20 |
| 1-Coastal aerosol | 442.7 | 442.2 | 60 |
4-1 NDREI(Normalized Difference Red Edge Index)#
其中,的中心波长在715nm左右。
def getNDRE(image):
ndre = image.normalizedDifference(['B7','B5']).rename("NDRE")
image = image.addBands(ndre)
return(image)
4-2 NDVI705(Red Edge Normalized Difference Vegetation Index)#
def getND750(image):
ND750 = image.normalizedDifference(['B6','B5']).rename("ND750")
image = image.addBands(ND750)
return(image)
和是由Daniel A Sims和John A Gamon在2002年提出来的[5]。
5-1 mSR705(Modified Red Edge Simple Ratio Index)#
叶表反射多往往是因为叶表发生镜面反射,这使得叶片在整个可见光区域的反射率增加。为减少这一影响,选用表征叶表反射率,得到改进红边比值植被指数。
def getMSR(image):
MSR = image.normalizedDifference(['B6','B1']).rename("MSR")
image = image.addBands(MSR)
return(image)
5-2 mNDVI705(Modified Red Edge Normalized Difference Vegetation Index)#
改进红边归一化植被指数,就是在的基础上,修正了叶片镜面发射的影响。Datt在1999年基于同一思想提出一指数,但是是基于,Daniel A Sims和John A Gamon认为更好,因为它比更稳定,更低,且花青素吸收峰在445nm附近。
def getmND750(image):
mND750 = image.expression(
'(R750 - R705) / (R750 + R705 - 2 * R445)', {
'R750': image.select('B6').divide(10000),
'R705': image.select('B5').divide(10000),
'R445': image.select('B1').divide(10000)
}).rename("mND750")
image = image.addBands(mND750)
return(image)
参考材料:
[2]Extraction of red edge optical parameters from Hyperion data for estimation of forest leaf area index | IEEE Journals & Magazine | IEEE Xplore
[3]https://adsabs.harvard.edu/pdf/1988ESASP.287..279G
[4]Red edge spectral measurements from sugar maple leaves: International Journal of Remote Sensing: Vol 14, No 8 (tandfonline.com)
[5]Relationships between leaf pigment content and spectral reflectance across a wide range of species, leaf structures and developmental stages - ScienceDirect





【推荐】编程新体验,更懂你的AI,立即体验豆包MarsCode编程助手
【推荐】凌霞软件回馈社区,博客园 & 1Panel & Halo 联合会员上线
【推荐】抖音旗下AI助手豆包,你的智能百科全书,全免费不限次数
【推荐】博客园社区专享云产品让利特惠,阿里云新客6.5折上折
【推荐】轻量又高性能的 SSH 工具 IShell:AI 加持,快人一步
· CSnakes vs Python.NET:高效嵌入与灵活互通的跨语言方案对比
· DeepSeek “源神”启动!「GitHub 热点速览」
· 我与微信审核的“相爱相杀”看个人小程序副业
· Plotly.NET 一个为 .NET 打造的强大开源交互式图表库
· 上周热点回顾(2.17-2.23)