生信-cufflinks输入输出文件分析
转自:https://wenku.baidu.com/view/8d6a95d20d22590102020740be1e650e52eacf2a.html
输出包含3个文件:转录组的组装.gtf 转录本表达水平.fpkm_tracking 基因表达水平.fpkm_tracking
1.转录组的组装.gtf ——transcripts.gtf
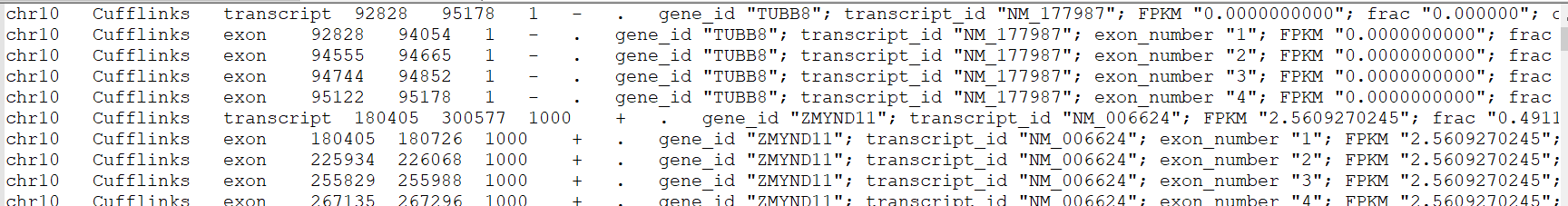
转自:http://yangl.net/2016/06/03/cufflinks/
1.chrX来源染色体名称
2.来源,产生此文件的程序
3.结构,常为transcript或exon
4.开始
5.结束
6.得分
7.Cufflinks猜测isoform来自参考序列的那一条链,一般是'+','-'或'.';
8. Cufflinks不去预测起始或终止密码子框的位置
9及以后是attribute的位置:
geneid;transcript_id;FPKM(Fragments Per Kilobase of exon model per Million mapped fragments)
frac 保留着的一项,忽略即可,以后可能会取消这个;conf_lo isoform丰度的95%置信区间的下边界,即 下边界值 = FPKM * ( 1.0 - conf_lo ) ;
conf_hi isoform丰度的95%置信区间的上边界,即 上边界值 = FPKM * ( 1.0 + conf_hi ) ;
cov 计算整个transcript上read的覆盖度;
full_read_support yes 当使用 RABT assembly 时,该选项报告所有的intr ons和exons是否完全被reads所覆盖


 浙公网安备 33010602011771号
浙公网安备 33010602011771号