HDF5介绍
Python and HDF5 ,Andrew Collette
HDF5,the most recent version of the “Hierarchical Data Format” originally developed at the National Center for Supercomputing Applications (NCSA), has rapidly emerged as the mechanism of choice for storing scientific data in Python.
HDF5是最初由美国国家超级计算应用中心(NCSA)开发的“层次数据格式”的最新版本,它已迅速成为在Python中存储科学数据的首选机制。
问题引入
当我还是一名研究生时,我遇到了一个严重的问题:一个全新的数据集,由在全国公认的等离子研究设备上整整一周辛苦收集的数百万个数据点组成,其中包含的值太小了。大约40个数量级太小。我和我的顾问挤在他的办公室里,在运行我们可视化套件的崭新G5 Power Mac前,试图找出问题所在。数据已从机器上正确获取。看起来来自epriment数字化仪的原始原始文件很好。我在IDL编程中编写了一个(非常大的)脚本在我的Thinkpad笔记本电脑上使用可视化工具将原始数据转换为文件可以使用。这种内部格式本身很简单:只有一个固定宽度的短标题然后是浮点数据的二进制转储。尽管如此,我还是花了一个小时左右编写一个程序来验证和绘制笔记本电脑上的文件。他们很好。然而,当加载到可视化工具中时,所有在IDL中看起来很漂亮的数据都变成了一堆毫无特色的、非结构化的价值观,大约在10-41之间.最后,它来到了我们面前:数字转换器和我的Thinkpad都使用“小端”格式来表示浮点数,与G5 Mac的“大端”格式不同。写入一台机器的原始值无法在另一台机器上读取,反之亦然。我记得我觉得这太愚蠢了(还有其他不太礼貌的变体)。得知这个问题如此普遍,以至于IDL提供了一个特殊的例程来处理它(SWAP_ENDIAN),并没有改善我的心情。当时,我并不太关心数据存储的细节。这件事和其他类似的事情改变了我的想法。作为一名科学家,我最终认识到,我们在组织和存储数据时所做的选择也是一种选择关于沟通。标准的、精心设计的格式不仅使个人生活更轻松(并消除了像“endian”问题这样的愚蠢的时间浪费),而且还使与全球受众共享数据成为可能。
Organizing Data and MetaData
import h5py
f=h5py.File("example.hdf5",'w')
#测试数据1
temperature=np.random.radom(1024)
f["temperature"]=temperature#温度数据
f["start_time"]=start_time#采样时间
f['station'=15#采样地点
Coping with Large Data Volumes
f=h5py.File("example.hdf5",'r')
#温度
temp=f["temperature"]
#像numpy数组一样进行访问
temp[:10]#前10个
temp[:10:2]#设置步长
优点
优点1
HDF5最大的优势之一是它支持子集和部分I/O。
请记住,实际数据存在在磁盘上;当切片应用到HDF5数据集时,会找到适当的数据并加载到内存中。以这种方式进行切片,它利用了HDF5的底层子设置功能,因此速度非常快。
优点2
关于HDF5的另一个优点是,你可以控制存储空间的分配方式。例如,除了一些元数据外,一个全新的数据集会占用零空间,并且默认情况下,字节只在磁盘上使用来保存您实际写入的数据。
当存储量较高时,您甚至可以基于数据集使用压缩方法:
>>> compressed_dataset = f.create_dataset("comp", shape=(1024,), dtype='int32',
compression='gzip')
>>> compressed_dataset[:] = np.arange(1024)
>>> compressed_dataset[:]
array([ 0, 1, 2, ..., 1021, 1022, 1023])
What Exactly is HDF5?
HDF5 is a great mechanism for storing large numerical arrays of homogenous type, for data models that can be organized hierarchically and benefit from tagging of datasets with arbitrary metadata.(HDF5是一种很好的机制,用于存储“同质类型的大型数值数组”,用于可以“分层组织”的数据模型,并受益于使用“任意元数据”标记数据集。)
It’s quite different from SQL-style relational databases. HDF5 has quite a few organizational tricks up its sleeve (see Chapter 8, for example), but if you find yourself needing to enforce relationships between values in various tables, or wanting to perform JOINs on your data, a relational database is probably more appropriate. Likewise, for tiny 1D datasets you need to be able to read on machines without HDF5 installed. Text format like CSV (with all their warts) are a reasonable alternative.(它与SQL风格的关系数据库截然不同。HDF5有很多组织技巧,但如果您发现需要在各种表中的值之间强制执行关系,或者希望对数据执行JOIN,则关系数据库可能更合适。同样,对于微小的1D数据集,您需要能够在未安装HDF5的机器上读取。像CSV这样的文本格式(及其所有缺点)是一种合理的选择。)
HDF5 is just about perfect if you make minimal use of relational features and have a need for very high performance, partial I/O, hierarchical organization, and arbitrary metadata(如果您对关系特性的使用最少,并且需要非常高的性能、部分I/O、分层组织和任意元数据,那么HDF5几乎是完美的。).
So what, specifically, is “HDF5”? I would argue it consists of three things:
-
A file specification and associated data model(文件规范和相关数据模型。).
-
A standard library with API access available from C, C++, Java, Python, and others(可从C、C++、Java、Python等获得API访问的标准库。).
-
A software ecosystem, consisting of both client programs using HDF5 and “analysis platforms” like MATLAB, IDL, and Python(一个软件生态系统,由使用HDF5的客户端程序和MATLAB、IDL和Python等“分析平台”组成。).
HDF5:The File
在前面的简短示例中,您看到了HDF5数据模型的三个主要元素:
-
数据集:将数字数据存储在磁盘上的类似数组的对象;
-
组:存储数据集的分层容器和其他组;
-
属性:可以附加到数据集(和组!)的用户定义的元数据位。
使用这些基本抽象,用户可以构建特定的“应用程序格式”以适合问题领域的方法对数据进行分析。例如,我们的“天气”每个站使用一组“站”代码,每个测量站使用单独的数据集参数,属性用于保存有关数据集含义的附加信息。对于实验室或其他组织来说,很常见的一种“表单内格式”,即指定组、数据集和属性的排列方式
用于存储信息。
- 元素查看
>>> f.keys()
[u'15', u'big', u'comp']
>>> f["/15"].keys()
[u'temperature', u'wind']
HDF5:library
HDF5文件规范和开源库由HDF Group维护,总部位于伊利诺伊州香槟市的非营利组织。以前是伊利诺伊大学厄巴纳-香槟分校,HDF集团的主要产品是HDF5软件库。
HDF5:The Ecosystem
HDF5特别有用的一个方面是您可以读写几乎每个平台的文件。IDL语言多年来一直支持HDF5;MATLAB也有类似的支持,现在甚至使用HDF5作为其默认格式
“.mat”保存文件。绑定也可用于Python、C++、Java、.NET和LabView,在其他中。机构用户包括NASA的地球观测系统“EOS5”格式是HDF5容器之上的应用程序格式前面的示例更简单。即使是竞争NetCDF格式的最新版本,NetCDF4是使用HDF5组、数据集和属性实现的。
HDF5 Basis
逻辑结构
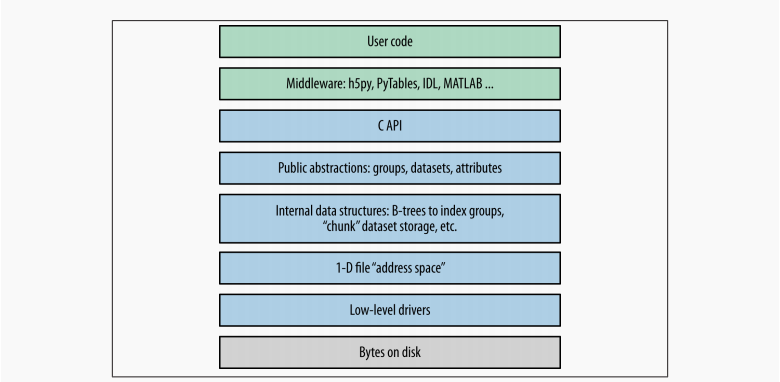
大多数客户端代码,包括Python包h5py和PyTables,都使用本机C语言
API(HDF5本身用C编写)。正如我们在介绍中看到的,HDF5数据模型
由三个主要的公共抽象组成:数据集、组和属性。C API(及其之上的Python代码)旨在操纵这些对象。
HDF5使用各种内部数据结构来表示组、数据集和属性。例如,组使用名为“B树”的结构对其条目进行索引这使得检索和创建组成员非常快速,即使在数百数千个对象存储在一个组中。在性能方面,您通常只关心这些数据结构性能考虑。例如,当使用分块存储时了解数据在磁盘上的实际组织方式非常重要。
接下来的两层与数据如何进入磁盘有关。HDF5型对象都位于1D逻辑地址空间中,就像在常规文件中一样。然而,有一个这个空间和磁盘上字节的实际排列之间的额外层。HDF5驱动程序注意写磁盘的机制,在这个过程中可以做一些惊人的事情东西。
例如,HDF5核心驱动程序允许您使用完全位于内存中的文件速度极快。通过族驱动程序,可以将单个文件拆分为大小规则的片段。mpio驱动程序允许您使用
消息传递接口(MPI)库。全部的对于在较高级别的组、数据集和属性。
python package
- h5py
- PyTables
HDF5 Tools
-
HDFView
HDFView是HDF Group提供的HDF5文件的免费图形浏览器。它有点基础,但是用Java编写的,因此可以在Windows、Linux和Mac上使用。有一个内置的类似电子表格的数据浏览器和基本的绘图功能。 -
ViTables
另一个免费图形浏览器ViTables中打开的相同HDF5文件。它针对处理PyTables文件进行了优化,尽管它可以处理通用的HDF5文件非常好。ViTables的一个主要优点是它预先安装了这样的
Python发行版称为PythonXY,因此您可能已经拥有了它。 -
命令行工具
如果您习惯了命令行,那么安装HDF命令行工具绝对值得。这些通常可通过包管理器获得;如果没有,您可以在www.hdfgroup.org上获取。Windows版本也可用。
$ h5ls demo.hdf5 array Dataset {10} group Group scalar Dataset {SCALAR}
常见模式
>>> f = h5py.File("name.hdf5", "w") # New file overwriting any existing file
>>> f = h5py.File("name.hdf5", "r") # Open read-only (must exist)
>>> f = h5py.File("name.hdf5", "r+") # Open read-write (must exist)
>>> f = h5py.File("name.hdf5", "a") # Open read-write (create if doesn't exist)
文件驱动
文件驱动程序位于文件系统和更高级别的HDF5组之间标签、标签和属性。他们处理映射HDF5“地址的机制空间”转换为磁盘上的字节排列。通常你不必担心哪个驱动程序正在使用,因为默认驱动程序适用于大多数应用程序。
驱动程序的优点在于,一旦文件打开,它们就完全透明。您只需正常使用HDF5库,驱动程序负责存储机制。
core driver
核心驱动程序将文件完全存储在内存中。显然,可以存储的数据量是有限的,但代价是惊人的快速读写。当你想加快内存访问速度,但又想使用HDF5结构时,这是一个很好的选择。要启用,请将驱动程序关键字设置为“core”:
>>> f = h5py.File("name.hdf5", driver="core")
您还可以告诉HDF5创建一个磁盘上的“备份存储”文件,关闭时文件映像将保存到该文件中:
>>> f = h5py.File("name.hdf5", driver="core", backing_store=True)
顺便说一下,backing_store关键字还会告诉HDF5在打开文件时从磁盘加载任何现有图像。因此,如果整个文件都能放在内存中,您只需要读写一次图像;像数据集读写、属性创建等等,根本不需要任何磁盘I/O。
family driver
有时,将一个文件分割成多个图像是很方便的,所有这些图像都具有一定的最大大小。此功能最初是为了支持无法处理2GB以上文件大小的文件系统而实现的。
>>> # Split the file into 1-GB chunks
>>> f = h5py.File("family.hdf5", driver="family", memb_size=1024**3)
memb_size的默认值为231-1,与驱动程序的历史起源保持一致。
mpio driver
此驱动程序是并行HDF5的核心。它允许您同时从多个进程访问同一文件。您可以有数十个甚至数百个并行计算过程,所有这些过程共享磁盘上单个文件的一致视图。
正确使用mpio驱动程序可能很棘手。
用户块(The user Block)
HDF5的一个有趣的特性是,文件前面可能有任意的用户数据。打开文件时,库会在文件开头查找HDF5标头,然后是512字节,然后是1024字节,依此类推,以2的幂表示。文件开头的这种空间称为“用户块”,您可以在其中存储任何需要的数据。
>>> f = h5py.File("userblock.hdf5", "w", userblock_size=512)
>>> f.userblock_size # Would be 0 if no user block present
512
>>> f.close()
>>> with open("userblock.hdf5", "rb+") as f: # Open as regular Python file
... f.write("a"*512)
Working with Datasets
数据集是HDF5的核心功能。您可以将它们视为磁盘上的NumPy数组。HDF5中的每个数据集都有名称、类型和形状,并支持随机访问。与内置的np.save和friends不同,不需要将整个数组作为一个块进行读写;您可以使用标准的NumPy语法进行切片,以读取和写入所需的部分。
>>> f = h5py.File("testfile.hdf5")
>>> arr = np.ones((5,2))
>>> f["my dataset"] = arr
>>> dset = f["my dataset"]
>>> dset
<HDF5 dataset "my dataset": shape (5, 2), type "<f8">
Type and shape
>>> dset.dtype
dtype('float64')
>>> dset.shape
(5, 2)
Reading and Writing
>>> out = dset[...]
>>> out
array([[ 1., 1.],
[ 1., 1.],
[ 1., 1.],
[ 1., 1.],
[ 1., 1.]])
>>> type(out)
<type 'numpy.ndarray'>
切片到数据集对象将返回NumPy数组。记住当您这样做时实际发生的情况:h5py将切片选择转换为数据集的一部分,并让HDF5从磁盘读取数据。换句话说,忽略缓存,切片操作会导致磁盘读取或写入。
Creating Empty Datasets
>>> dset = f.create_dataset("test1", (10, 10))
>>> dset
<HDF5 dataset "test1": shape (10, 10), type "<f4">
>>> dset = f.create_dataset("test2", (10, 10), dtype=np.complex64)
>>> dset
<HDF5 dataset "test2": shape (10, 10), type "<c8">
HDF5足够聪明,可以只在磁盘上分配实际需要的空间来存储您所写的数据。下面是一个示例:假设您希望创建一个1D数据集,该数据集可以保存来自长期运行实验的4GB数据样本:
>>> dset = f.create_dataset("big dataset", (1024**3,), dtype=np.float32)
>>> dset[0:1024] = np.arange(1024)
>>> f.flush()
Automatic Type Conversion and Direct Reads
>>> big_out = np.empty((100, 1000), dtype=np.float64)
>>> dset.read_direct(big_out)
Reading with astype
>>> with dset.astype('float64'):
... out = dset[0,:]
>>> out.dtype
dtype('float64')
Fill values
>>> dset = f.create_dataset('empty', (2,2), dtype=np.int32)
>>> dset[...]
array([[0, 0],
[0, 0]])
>>> dset = f.create_dataset('filled', (2,2), dtype=np.int32, fillvalue=42)
>>> dset[...]
array([[42, 42],
[42, 42]])
>>> dset.fillvalue
42
Creating Resizable Datasets
>>> dset = f.create_dataset('resizable', (2,2), maxshape=(2,2))
>>> dset.shape
(2, 2)
>>> dset.maxshape
(2, 2)
>>> dset.resize((1,1))
>>> dset.shape
(1, 1)
>>> dset.resize((2,2))
>>> dset.shape
(2, 2)
>>> dset.resize((2,3))
ValueError: unable to set extend dataset (Dataset: Unable to initialize object)
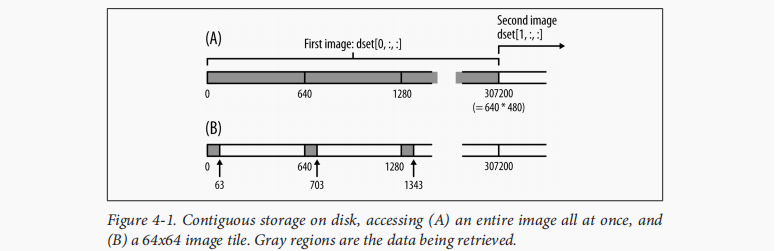
Contiguous Storage
>>> f = h5py.File("imagetest.hdf5")
>>> dset = f.create_dataset("Images", (100, 480, 640), dtype='uint8')
>>> image = dset[0, :, :]
>>> image.shape
(480, 640)
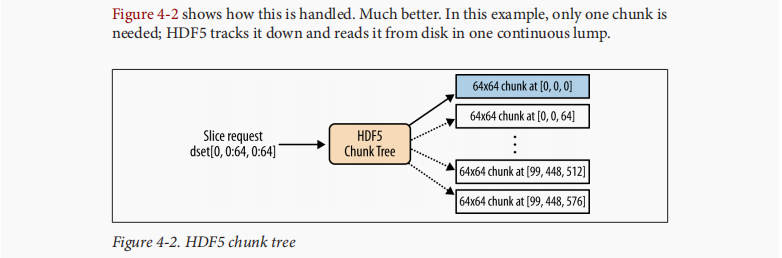
Chunked Storage
>>> dset = f.create_dataset('chunked', (100,480,640), dtype='i1', chunks= (1,64,64))
Auto-Chunking
>>> dset = f.create_dataset("Images2", (100,480,640), 'f', chunks=True)
>>> dset.chunks
(13, 60, 80)
Compression Filters
>>> dset = f.create_dataset("BigDataset", (1000,1000), dtype='f', compres
sion="gzip")
>>> dset.compression
'gzip'
GZIP/DEFLATE Compression
在HDF5中。它随HDF5的每次安装一起提供,具有以下优点:
• 适用于所有HDF5类型
• 内置HDF5,随处可见
• 中低速压缩
• 还可以通过使用SHUFFLE来提高性能(请参阅上的“SHUFILE过滤器”
第52页)
对于GZIP压缩器,compress_opts可以是从0到9的整数
默认值为4。
>>> dset = f.create_dataset("Dataset", (1000,), compression="gzip")
>>> dset = f.create_dataset("Dataset2", (1000,), compression=9)
>>> dset.compression
'gzip'
>>> dset.compression_opts
9
szip
SZIP是NASA广泛使用的专利压缩技术。一般来说,如果你与使用卫星数据的人交换文件,你只需要担心这一点。由于专利许可限制,HDF5的许多安装都禁用了压缩机(而不是解压器)。
>>> dset= myfile.create_dataset("Dataset3", (1000,), compression="szip")
LZF
对于只使用Python的文件,LZF是一个不错的选择。它与h5py一起发货;C源代码可用于BSD许可证下的第三方程序。与GZIP相比,它以较低的压缩比为代价,针对非常、非常快的压缩进行了优化。如果数据集有大量冗余数据点,则是最佳使用情况。此筛选器没有压缩端口。
>>> dset = myfile.create_dataset("Dataset4", (1000,), compression="lzf")
Groups, Links, and Iteration: The “H” in HDF5
The Root Group and Subgroups
>>> f = h5py.File("Groups.hdf5")
>>> subgroup = f.create_group("SubGroup")
>>> subgroup
<HDF5 group "/SubGroup" (0 members)>
>>> subgroup.name
u'/SubGroup'
>>> subsubgroup = subgroup.create_group("AnotherGroup")
>>> subsubgroup.name
u'/SubGroup/AnotherGroup'
>>> out = f.create_group('/some/big/path')
>>> out
<HDF5 group "/some/big/path" (0 members)>
Group basis
>>> f["Dataset1"] = 1.0
>>> f["Dataset2"] = 2.0
>>> f["Dataset3"] = 3.0
>>> subgroup["Dataset4"] = 4.0
Dictionary-Style Access
>>> dset1 = f["Dataset1"]
>>> dset4 = f["SubGroup/Dataset4"] # Right
>>> dset4 = f["SubGroup"]["Dataset4"] # Works, but inefficient
Special Properties
>>> f = h5py.File('propdemo.hdf5','w')
>>> grp = f.create_group('hello')
>>> grp.file == f
True
当您想检查文件是读/写还是只获取文件名时,这非常有用。第二个是.父属性。这将返回包含您的对象的Group对象:
>>> grp.parent
<HDF5 group "/" (1 members)>
links
Hard links
>>> f = h5py.File('linksdemo.hdf5','w')
>>> grpx = f.create_group('x')
>>> grpx.name
u'/x'
>>> f['y'] = grpx
>>> grpy = f['y']
>>> grpy == grpx
True
>>> grpx.name
u'/x'
>>> grpy.name
u'/y'
soft Links
>>> f = h5py.File('test.hdf5','w')
>>> grp = f.create_group('mygroup')
>>> dset = grp.create_dataset('dataset', (100,))
>>> f['hardlink'] = dset
>>> f['hardlink'] == grp['dataset']
True
>>> grp.move('dataset', 'new_dataset_name')
>>> f['hardlink'] == grp['new_dataset_name']
True
>>> grp.move('new_dataset_name', 'dataset')
>>> f['softlink'] = h5py.SoftLink('/mygroup/dataset')
>>> f['softlink'] == grp['dataset']
True
>>> softlink = h5py.SoftLink('/some/path')
>>> softlink
<SoftLink to "/some/path">
>>> softlink.path
'/some/path'
>>> grp.move('dataset', 'new_dataset_name')
>>> dset2 = grp.create_dataset('dataset', (50,))
>>> f['softlink'] == dset
False
>>> f['softlink'] == dset2
True
Iteration and Containership
How groups Are Actually stored?
>>> f = h5py.File('iterationdemo.hdf5','w')
>>> f.create_group('1')
>>> f.create_group('2')
>>> f.create_group('10')
>>> f.create_dataset('data', (100,))
>>> f.keys()
[u'1', u'10', u'2', u'data']
Dictionary-Style Iteration
{ key:value for key,value in f.items()}
Storing Metadata with Attributes
Attribute Basics
>>> f = h5py.File('attrsdemo.hdf5','w')
>>> dset = f.create_dataset('dataset',(100,))
>>> dset.attrs['title'] = "Dataset from third round of experiments"
>>> dset.attrs['sample_rate'] = 100e6 # 100 MHz digitizer setting
>>> dset.attrs['run_id'] = 144
>>> dset.attrs['title']
'Dataset from third round of experiments'
>>> dset.attrs['sample_rate']
100000000.0
>>> dset.attrs['run_id']
144
>>> [x for x in dset.attrs]
[u'title', u'sample_rate', u'run_id']
>>> dset.attrs['ones'] = np.ones((100,))
>>> dset.attrs['ones']
array([ 1., 1., 1., 1., 1., 1., 1., 1., 1., 1., 1., 1., 1.,
1., 1., 1., 1., 1., 1., 1., 1., 1., 1., 1., 1., 1.,
1., 1., 1., 1., 1., 1., 1., 1., 1., 1., 1., 1., 1.,
1., 1., 1., 1., 1., 1., 1., 1., 1., 1., 1., 1., 1.,
1., 1., 1., 1., 1., 1., 1., 1., 1., 1., 1., 1., 1.,
1., 1., 1., 1., 1., 1., 1., 1., 1., 1., 1., 1., 1.,
1., 1., 1., 1., 1., 1., 1., 1., 1., 1., 1., 1., 1.,
1., 1., 1., 1., 1., 1., 1., 1., 1.])
不过,这是有限度的。在HDF5中,使用默认设置(“紧凑”存储,而不是“密集”存储),属性的大小限制为64k。例如,如果我们试图存储一个(100100)数组,它会抱怨:
>>> dset.attrs['ones'] = np.ones((100, 100))
ValueError: unable to create attribute (Attribute: Unable to initialize object)
解决此限制的一种方法是将数据存储在数据集中,并链接到数据集
带有对象引用(参见第8章):
>>> ones_dset = f.create_dataset('ones_data', data=np.ones((100,100)))
>>> dset.attrs['ones'] = ones_dset.ref
>>> dset.attrs['ones']
<HDF5 object reference>
>>> ones_dset = f[dset.attrs['ones']]
>>> ones_dset[...]
array([[ 1., 1., 1., ..., 1., 1., 1.],
[ 1., 1., 1., ..., 1., 1., 1.],
[ 1., 1., 1., ..., 1., 1., 1.],
...,
[ 1., 1., 1., ..., 1., 1., 1.],
[ 1., 1., 1., ..., 1., 1., 1.],
[ 1., 1., 1., ..., 1., 1., 1.]])
Strings and File Compatibility
>>> dset.attrs['title_fixed'] = np.string_("Another title")
这通常不是问题,但一些基于FORTRAN的旧程序无法处理可变长度字符串。如果这对您的应用程序来说是个问题,请使用np.string_,或者等效地使用NumPy类型S的数组。
Python Objects
>>> dset.attrs['object'] = {}
TypeError: Object dtype dtype('object') has no native HDF5 equivalent
这是故意的。正如错误消息所示,HDF5没有“原生”的内置类型来表示Python对象,而像HDF5这样的面向可移植性格式的序列化对象通常被认为是坏消息。以“blob”形式存储数据会破坏HDF5的出色类型系统和互操作性。
然而,我不能告诉你如何编写应用程序。如果您真的想存储Python对象,最好的方法是将它们“pickle”(序列化)为字符串:
>>> import pickle
>>> pickled_object = pickle.dumps({'key': 42}, protocol=0)
>>> pickled_object
"(dp0\nS'key'\np1\nI42\ns."
>>> dset.attrs['object'] = pickled_object
>>> obj = pickle.loads(dset.attrs['object'])
>>> obj
{'key': 42}
Explicit Typing
>>> f = h5py.File('attrs_create.hdf5','w')
>>> dset = f.create_dataset('dataset', (100,))
>>> dset.attrs.create('two_byte_int', 190, dtype='i2')
>>> dset.attrs['two_byte_int']
190
>>> f.flush()
$ h5ls -vlr attrs_create.hdf5
Opened "attrs_create.hdf5" with sec2 driver.
/ Group
Location: 1:96
Links: 1
/dataset Dataset {100/100}
Attribute: two_byte_int scalar
Type: native short
Data: 190
Location: 1:800
Links: 1
Storage: 400 logical bytes, 0 allocated bytes
Type: native float
>>> dset.attrs['strings'] = ["Hello", "Another string"]
>>> dset.attrs['strings']
array(['Hello', 'Another string'],
dtype='|S14')
>>> dt = h5py.special_dtype(vlen=str)
>>> dset.attrs.create('more_strings', ["Hello", "Another string"], dtype=dt)
>>> dset.attrs['more_strings']
array([Hello, Another string], dtype=object)
这似乎是一个小区别,但当与第三方代码交谈时,这可能是工作程序和错误消息之间的区别。



 浙公网安备 33010602011771号
浙公网安备 33010602011771号