通过fastaread读取DNA序列并进行检测matlab仿真
1.算法描述
fastaread
fastaread函数是matlab生物信息学工具箱内置的一个函数,给我们的使用上带来了巨大的方便。对于基因DNA序列,转录RNA序列和表达蛋白序列的读取非常方便。
使用语法为:
p53nt = fastaread('p53nt.txt') %
p53nt.txt 为fasta 格式存储序列的文件 返回的p53nt为一个结构变量,包括Header 和 Sequence两个成员名称。
DNA序列
DNA序列或基因序列是使用一串字母表示的真实的或者假设的携带基因信息的DNA分子的一级结构。部分DNA序列或基因序列使用一串字母表示的真实的或者假设的携带基因信息的DNA分子的一级结构。
可能的字母只有A,C,G和T,分别代表组成DNA的四种核苷酸——腺嘌呤,胞嘧啶,鸟嘌呤,胸腺嘧啶。每个字母代表一种碱基,两个碱基形成一个碱基对,碱基对的配对规律是固定的,即是:A-T,C-G。典型的他们无间隔的排列在一起,例如序列AAAGTCTGAC。任意长度大于4的一串核苷酸被称作一个序列。关于它的生物功能,则依赖于上下文的序列,一个序列可能被正读,反读;包含编码或者无编码。DNA序列也可能包含"junk DNA"。
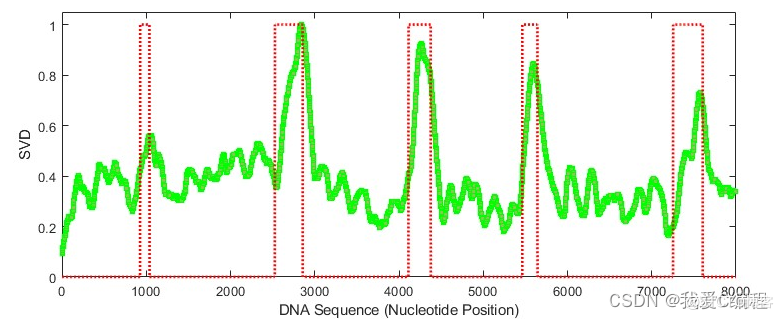
2.仿真效果预览
matlab2022a仿真结果如下:
3.MATLAB核心程序
[xA xT xC xG] = dna_binary(sample_dna);
xA = [zeros(1,floor(frmsz/2)) xA zeros(1,ceil(frmsz/2))]; % Zero padding from both sides
xT = [zeros(1,floor(frmsz/2)) xT zeros(1,ceil(frmsz/2))];
xC = [zeros(1,floor(frmsz/2)) xC zeros(1,ceil(frmsz/2))];
xG = [zeros(1,floor(frmsz/2)) xG zeros(1,ceil(frmsz/2))];
R = 0.992;
theta = (2*pi)/3;
% Filter Coefficients %
% ------------------- %
b = [1 0 -1];
a = [1 -2*R*cos(theta) R^2];
% Filtering the signal (DNA Sequence) %
% ----------------------------------- %
uA = filter(b,a,xA);
uT = filter(b,a,xT);
uC = filter(b,a,xC);
uG = filter(b,a,xG);
% Detection of Period-3 Behavior using Singular Value Decomposition %
% ----------------------------------------------------------------- %
for n = 1:length(sample_dna)
sa = reshape(uA(n:n+frmsz-1),3,frmsz/3);
XA(n) = max(svd(sa));
st = reshape(uT(n:n+frmsz-1),3,frmsz/3);
XT(n) = max(svd(st));
sc = reshape(uC(n:n+frmsz-1),3,frmsz/3);
XC(n) = max(svd(sc));
sg = reshape(uG(n:n+frmsz-1),3,frmsz/3);
XG(n) = max(svd(sg));
end
X = XA + XT + XC + XG;


 浙公网安备 33010602011771号
浙公网安备 33010602011771号