R_Studio(癌症)数据连续属性离散化处理
对“癌症.csv”中的肾细胞癌组织内微血管数进行连续属性的等宽离散化处理(分为3类),并用宽值找替原来的值
癌症.csv
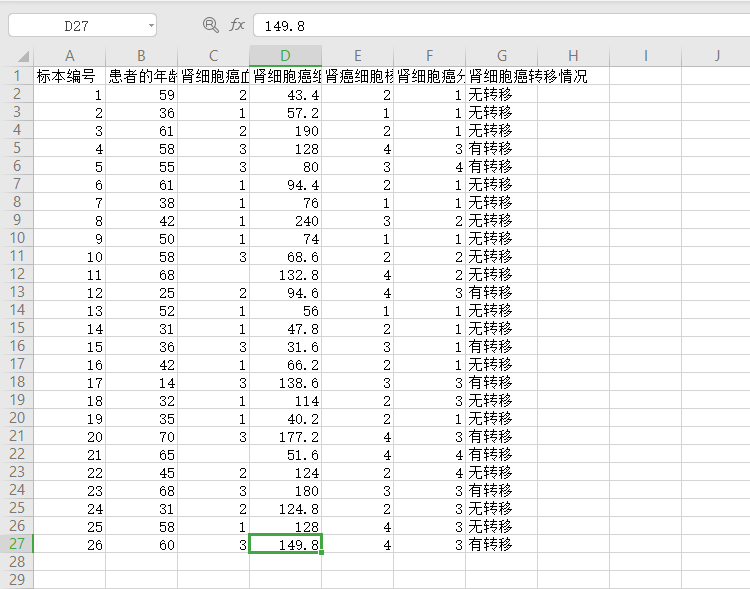

setwd('D:\\data') list.files() dat=read.csv(file="癌症.csv",header=TRUE) #等宽离散化 v1=ceiling(dat[,1]) #等频离散化 names(data)='f'#变量重命名 attach(dat) seq(0,length(f),length(f)/2)#等频划分为6组 v=sort(f)#按大小排序作为离散化依据 v2=rep(0,26)#定义新变量 for(i in 1:26) v2[i]=ifelse(f[i]<=v[13],1, ifelse(f[i]<=v[26],2)) detach(dat) #聚类离散化 result=kmeans(dat[,4],2) v3=result$cluster #图示结果 plot(dat[,4],v1,xlab='肾细胞癌组织内微血管数',ylab='等宽离散化') plot(dat[,4],v2,xlab='肾细胞癌组织内微血管数',ylab='等频离散化') plot(dat[,4],v3,xlab='肾细胞癌组织内微血管数',ylab='聚类离散化')
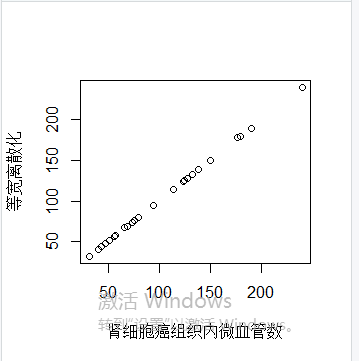
等宽离散化:将连续数据按照等宽区间标准离散化数据
setwd('D:\\data') list.files() dat=read.csv(file="癌症.csv",header=TRUE) #等宽离散化 v1=ceiling(dat[,4]) #图示结果 plot(dat[,4],v1,xlab='肾细胞癌组织内微血管数',ylab="等宽离散化")
等频离散化:将相同数量的数据放进一个区间
setwd('D:\\data') list.files() dat=read.csv(file="癌症.csv",header=TRUE) #等频离散化 names(data)='f'#变量重命名 attach(dat) seq(0,length(f),length(f)/2)#等频划分为6组 v=sort(f)#按大小排序作为离散化依据 v2=rep(0,26)#定义新变量 for(i in 1:26) v2[i]=ifelse(f[i]<=v[13],1, ifelse(f[i]<=v[26],2)) #图示结果 plot(dat[,4],v2,xlab='肾细胞癌组织内微血管数',ylab="等频离散化")

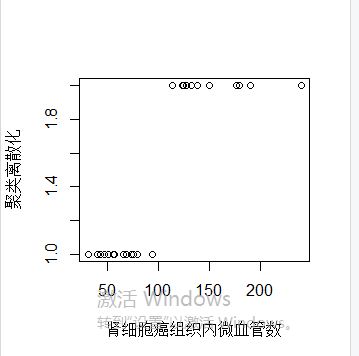
聚类离散化:一维聚类离散包括两个过程:通过聚类算法(K-Means算法)将连续属性值进行聚类,处理聚类之后的到的k个簇,得到每个簇对应的分类值(类似这个簇的标记)
setwd('D:\\data') list.files() dat=read.csv(file="癌症.csv",header=TRUE) #聚类离散化 result=kmeans(dat[,4],2) v3=result$cluster #图示结果 plot(dat[,4],v3,xlab='肾细胞癌组织内微血管数',ylab='聚类离散化')
(如需转载学习,请标明出处)



