【技术翻译】支持向量机简明教程及其在python和R下的调参
原文: Simple Tutorial on SVM and Parameter Tuning in Python and R

介绍
数据在机器学习中是重要的一种任务,支持向量机(SVM)在模式分类和非线性回归问题中有着广泛的应用. SVM最开始是由N. Vapnik and Alexey Ya. Chervonenkis
在1963年提出。从那时候开始,各种支持向量机被成功用于解决各种现实问题,比如文本聚类,图像分类,生物信息学(蛋白质分类,癌症分类),手写字符识别等等.
内容
1. 什么是支持向量机(support vector machine)?
2. 支持向量机如何工作?
3. 支持向量机的推导
4. 支持向量机的优点和缺点
5. Python和R下的支持向量机实现
什么是支持向量机?
支持向量机是一种可以用来解决分类问题和回归问题的监督式机器学习算法. 它使用一种叫做核方法(kernel trick)的技术来对数据进行转换,
基于这些转换,它在各种可能的解中找到最优的边界(optimal margin).
简而言之, 为了根据数据标签将数据分开来,SVM对数据进行一些很复杂的转换. 这篇文章,我们只会讨论SVM的分类算法.
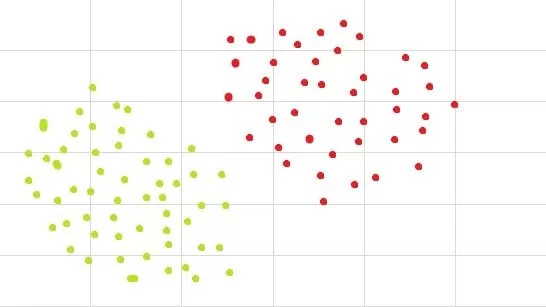
支持向量机如何工作?
要了解SVM如何工作最主要的是了解最大化训练数据间隔的最优分隔超*面(optimal separating hyperplane)是什么. 我们一个一个术语来接*这个目标.
什么是分类超*面?
对于上面那幅图,我们是能够分开那些数据的. 比如我们可以在数据中间画一条线, 线下面全是绿色数据点,线上面全是红色数据点.
那么问题来了,它明明是条线,你为什么叫它超*面呢?
在上面的图中, 我们仅仅考虑了最简单的情形,数据分布于2-维*面. 然而SVM在一般的n-维空间也能工作. 而在更高维空间, 超*面是*面的一般情况.
比如:
1 维数据,一个点代表着超*面
2 维数据,一条线代表着超*面
3 维数据,一个*面代表着超*面
对于更高维数据,就叫做超*面
前面我们提到,SVM的目标是找到最优分隔超*面. 那么什么样的超*面才是最优的呢?事实是存在分隔超*面并不一定代表它就是最优的.
下面我们通过几张图来理解这个问题.
1. 很多超*面
下图中有很多超*面,那个才是分隔超*面呢?很容易看出线B 是能够很好区分两个类的超*面.
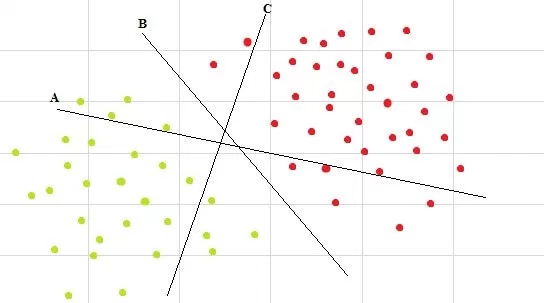
2. 很多分隔超*面
也可能出现有多个分隔超*面的情况. 我们如何找到最优分隔超*面呢, 直观上来说,如果我们选择了一个距离一类点很*的超*面,那么它的泛化能力肯定不是很好. 所以要找到距离各个类中的点都尽可能元的超*面.
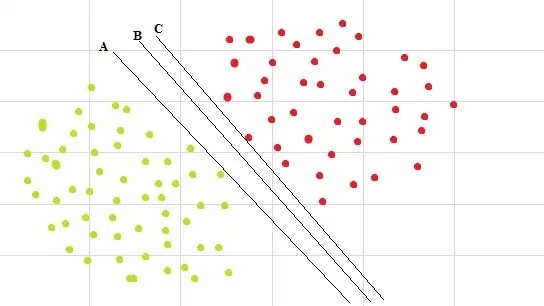
在上图中,最优超*面就是超*面B.
因此, 最大化每一类中距离超*面最*的点和超*面之间的距离,就能得到最优分隔超*面. 这个距离被称作: 边界(margin).
SVM的目标就是找到最优超*面, 因为它不仅仅可以对现有的数据分类,也能够对未知的数据进行预测. 最优超*面也就是有着最大边界(margin)的超*面.
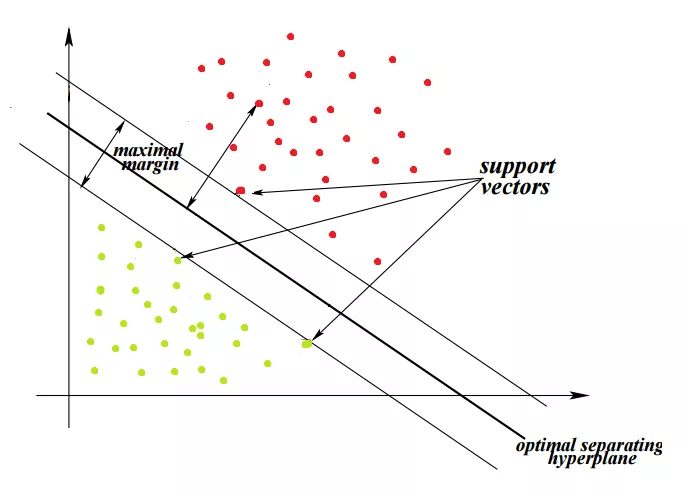
数学推导
现在我们已经大概了解了算法背后的基本概念,接下来我们看看SVM的数学推导
我假设你已经对基本的向量,向量代数,数学,正交投影有了基本的了解. 这些概念都可以在这篇文章中有介绍: 机器学习中的线性代数
超*面方程
你肯定知道直线方程可以是: $y = mx + c$, m是直线的斜率, c是直线的y轴截距.
更一般的超*面的方程如下:
$$W^Tx = 0$$
式中, $x$和$w$是向量, $W^Tx$,代表向量的点积, $W$通常被称为权向量.
对于上面的直线$y - mx -c = 0$. 在这种情况下:
$$w = \begin{pmatrix} -c\\-m\\1 \end{pmatrix}$$
$$x = \begin{pmatrix}1\\x\\y \end{pmatrix}$$
$W^Tx = 0$
仅仅是同一样东西的两种不同表达. 那么我们为什么使用$W^Tx = 0$. 仅仅是因为在面对高维数据的时候,这种表达更好处理. $W$代表垂直*面的向量, 这种特性在我们计算点到超*面距离的时候很有用.
理解限制条件
在我们分类问题中的训练数据是$\{(x_1,y_1), (x_2, y_2),...,(x_n,y_n)\}\in R^n * \{-1, 1\}.$ 这意味着训练数据是一些$x_i$, n 维向量, $y_i$是$x_i$的标签,$y_i$ = 1 意味着特征向量$x_i$属于类别1,反之属于类别 -1.
在一个分类问题中,我们试着找出一个函数 $y = f(x):R^n\rightarrow \{-1,1\}$,这个函数是从训练数据学习得到. 然后我们用这个函数来预测未知数据的类别.
f(x)有无数种可能性,我们必须给他加一个限制条件,缩小f(x)的范围. 在SVM的例子中, f(x) 必须满足$W^Tx = 0$
也可以被表示为 $\vec{w}\cdot\vec{x} + b = 0; \vec{w} \in R^n and b \in R$
这将输入空间分为两个部分,一部分是-1类别,一部分是1类别
接下来的文章,我们会考虑2维向量,假设$H_0$是数据的分隔超*面,满足以下条件:
$\vec{w}\cdot\vec{x} + b = 0 $
在有$H_0$情况下,我们可以选择两个其他的超*面$H_1$和$H_2$,他们也可以分隔数据且满足以下方程:
$\vec{w}\cdot\vec{x} + b = -\ sigma $ $\vec{w}\cdot\vec{x} + b = \sigma $
这使得$H_0$到$H_1$和$H_2$的距离相等
变量$\sigma$的不确定的,因此我们可以令\sigma = 1来简化问题.
$\vec{w}\cdot\vec{x} + b =-1 $ $\vec{w}\cdot\vec{x} + b = 1 $
接下来我们要确定这二者之间没有其他点. 因此我们会选择满足以下限制条件的超*面, 对于每个向量$x_i$:
要么:$对于属于-1 类的x_i 来说 \vec{w} \cdot \vec{x} \leq -1$
要么:$对于属于 1类的x_i 来说 \vec{w} \cdot \vec{x} \geq 1$
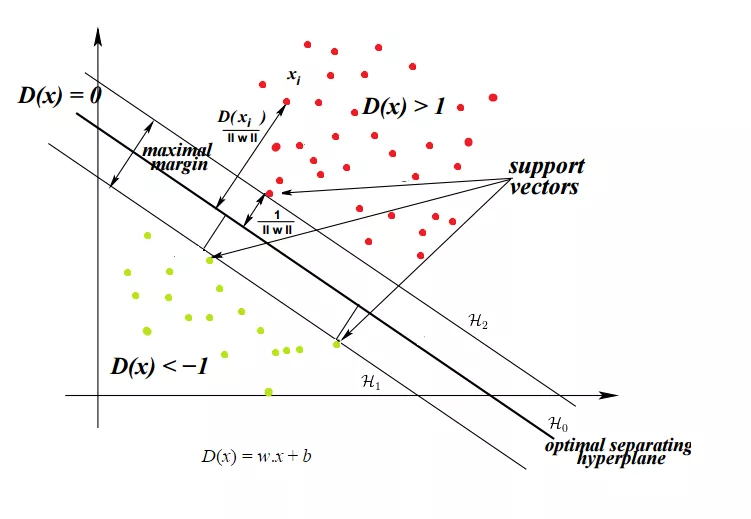
上述限制条件可以整合到一个式子中:$y_i(\vec{w}\cdot\vec{x_i}) \geq 1 i \in [1,n]$
为了简洁我们略去计算margin的推导: margin用m来表示:
$m = \frac{2}{||\vec{w}||}$
式子中的唯一变量就是$w$,因此最大化margin我们可以转化为最小化$||\vec{w}||$, 优化目标可以转化为以下:
$$Min \frac{||\vec{w}||}{2}$$
$$S.t. y_i(\vec{w} \cdot \vec{x_i}+b) \geq 1, 对于\forall i = 1,...,n$$
上面的式子在我们的数据是完全线性可分的时候是有效的,但是有些时候我们的数据并不是完全线性可分的,或者说数据由于受到噪声的干扰,即使线性可分,但是噪声会使得找到的超*面并不是最优的.
对于这个问题我们引入松弛变量(slack variable), 允许一些点落到分类间隔之内,但是对于这些点我们进行一些惩罚:
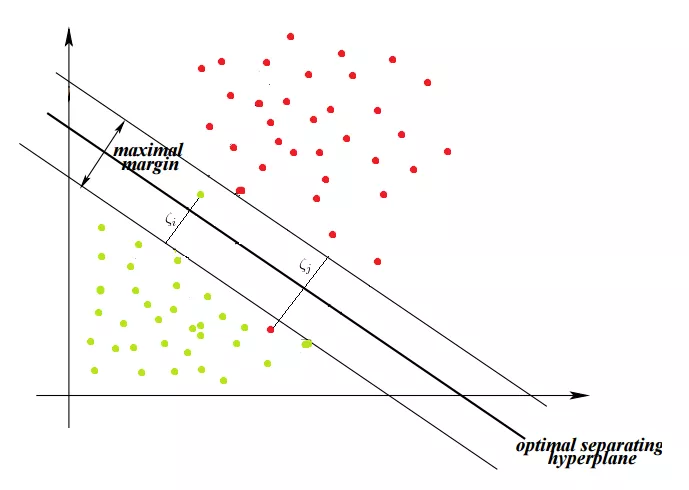
在这种情况下,算法尽量使得松弛变量为0. 算法最小化的不是错分类的综述,而是最小化总的错分距离.
限制条件现在变为:$y_i(\vec{w}\cdot\vec{x_i} + b) \geq 1- \varsigma_i, \forall 1 \geq i \leq n_i, \varsigma_i \geq 0$
优化目标变为:$\frac{||\vec{w}||}{2} + C\sum_i \varsigma_i, \forall 1 \geq i \leq n, \varsigma_i \geq 0$
参数$C$是正则化参数控制松弛变量大小和分类间隔大小的*衡.
小$C$会使得越过边界的点更容易被忽视,从而使得边界增大.
大$C$会使得越过边界的点更难被忽视,从而使得边界减小.
对于$C = \inf$, 所有的限制条件都是强制的.
对于2-维*面来说,最好的分类方式就是一条直线,对于3-维空间来说,最好的分类方式就会一个*面。但是并不是每次都能使用直线或者*面达到完美的分类,有时候我们需要用非线性区域来将类别分开.SVM使用核函数来解决这种非线性分类问题,核函数能够将数据映射到一个不同的空间,在这个空间中,我们可以使用线性超*面将数据分开. 这种方法被称为核方法.
假设 $\phi$是将$x_i$映射到$\phi(x)$的核函数,限制条件变为:
$$y_i(\vec{w}\cdot\vec{x_I} +b) \geq 1- \varsigma_i, \forall 1 \leq i \geq n, \varsigma_i \geq 0$$
优化目标变为:$\\frac{|| \vec{w}||}{2} + C\sum_i \varsigma_i $
这里我们不深入如何去解这个优化问题,最常见解决这类问题的方法是凸优化(convex optimization)
SVM的优点和缺点
针对特定的数据集,每个分类算法都有自己的优点和缺点. SVM的优点如下:
- 优化目标的凸优化本质保证了找到最优解的可行性. 而且这个最优解是全局最优解而不是局部最优解
- SVM既适用于线性可分数据也适用于非线性可分数据(使用核方法就可以), 只要找到对应的惩罚参数C就可以.
- 对于高维数据和低维数据,SVM都有效, 即使在高维数据下SVM也能很高效工作,因为SVM的特性只由支持向量数目确定,而不是整个数据的维度, 非支持向量之外的数据点对于SVM无关紧要,甚至可以去除.
SVM缺点如下:
- 他们不适合大数据集训练,因为训练时间很长,而且对计算性能要求较高
- 对于有重叠类别的噪声数据,SVM不是很有效
Python和R下的SVM
python下最常用的实现机器学习算法的库是scikit-learn, SVM在scikit-learn下的分类函数是svm.SVC()
sklearn.svm.SVC(c = 1.0, kernel = 'rbf',degree=3, gamma='auto')
参数如下:
C:正则化参数
kernel: 算法中使用的核函数, 可以是 'linear','poly','rbf','sigmoid','precomputed', 或者是用户函数. 默认值是'rbf'
degree: 'poly' 多项式核函数中的维度,默认是3,其他核函数忽略这个参数
gamma: 'rbf','poly','sigmoid' 中的系数, 如果gamma是'auto' 那么会默认使用1/n 个特征
这里很多参数我在这篇文章中都没有提到过,进一步了解你可以查看这里.
我们可以通过改变参数 C, $\gamma$ 和核函数来优化SVM, scikit-learn中参数参数优化函数是gridSearch().
sklearn.model_selection.GridSearchCV(estimator, param_grid)
参数如下:
estimator:是我们我估计的对象,比如这里就是svm.SVC()
param_grid:包含要优化的参数名字和要优化的值的字典或者列表
在上述代码中,我们要考虑优化的参数是C和gamma, 这些参数的最优值在我们给定的参数中,这里我们仅仅给出了一些值,我们也可以给出一系列取值范围,但是这样程序执行的时间会变长.
我自己写的一个简单的svm iris数据集分类简单实现:
# -*- coding: utf-8 -*-
import numpy as np
import matplotlib.pyplot as plt
from sklearn import svm, datasets
from sklearn.cross_validation import train_test_split
from sklearn.model_selection import GridSearchCV
#载入数据
iris = datasets.load_iris()
#正则化参数
C = 1
#可以选择不同的特征组合
x = iris.data[:,:2]
y = iris.target
#测试数据集和训练数据集划分
x_train,x_test, y_train, y_test = train_test_split(x,y,random_state = 1)
#linear核函数
linear_model = svm.SVC(kernel='linear',C = C).fit(x_train,y_train)
#rbf核函数
rbf_model = svm.SVC(kernel='rbf',C = C).fit(x_train,y_train)
linear_predict = linear_model.predict(x_test)
rbf_predict = rbf_model.predict(x_test)
count = 0
for i in range(len(linear_predict)):
if linear_predict[i]==y_test[i]:
count += 1
linear_accuracy = float(count)/len(linear_predict)
print(linear_accuracy)
count = 0
for i in range(len(rbf_predict)):
if rbf_predict[i]==y_test[i]:
count += 1
rbf_accuracy = float(count)/len(rbf_predict)
print(rbf_accuracy)
#模型选择
parameters = {"kernel":('linear','rbf','poly'),'C':[0.5,1,2,3,10]}
searchmodel = GridSearchCV(svm.SVC(),param_grid =parameters)
searchmodel.fit(x,y)
print(searchmodel.best_score_)
print(searchmodel.best_estimator_.C)
print(searchmodel.best_estimator_.kernel)




