用随机森林分类
分类方法有很多种,什么多分类逻辑回归,KNN,决策树,SVM,随机森林等,
比较好用的且比较好理解的还是随机森林,现在比较常见的有python和R的实现。原理就不解释了,废话不多说,show me the code
import csv
import numpy as np
from sklearn.ensemble import RandomForestRegressor
from sklearn import preprocessing
from sklearn.utils import shuffle
from sklearn.metrics import mean_squared_error, explained_variance_score
def load_dataset(filename):
file_reader = csv.reader(open(filename,'rt'), delimiter=',')
X, y = [], []
for row in file_reader:
X.append(row[0:4])
y.append(row[-1])
# Extract feature names
feature_names = np.array(X[0])
return np.array(X[1:]).astype(np.float32),np.array(y[1:]).astype(np.float32), feature_names
if __name__=='__main__':
X, y, feature_names = load_dataset("D:\\yudata.csv")
X, y = shuffle(X, y, random_state=7)
# X, y = random.shuffle(X, y)
num_training = int(0.9 * len(X))
X_train, y_train = X[:num_training],y[:num_training]
X_test, y_test = X[num_training:],y[num_training:]
rf_regressor = RandomForestRegressor(n_estimators=1000, max_depth=10, min_samples_split=2)
rf_regressor.fit(X_train, y_train)
y_pred = rf_regressor.predict(X_test)
mse = mean_squared_error(y_test, y_pred)
evs = explained_variance_score(y_test,y_pred)
print(y_test)
print(y_pred)
print(rf_regressor.feature_importances_)
训练数据的话,使用csv,例如
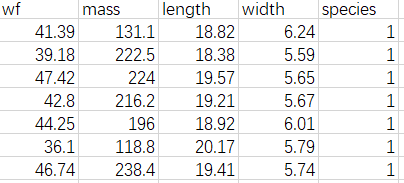
有几个地方要注意,
1: open(filename,'rt') ,不要写成‘rb’,可以写成‘r’
2:shuffle, 在random包里有,numpy也有,sklearn.utils里也有,本文使用的是最后者,from sklearn.utils import shuffle
具体原理可以参考 https://blog.csdn.net/huanhuan_Coder/article/details/82787923 写得比较好,前两者得shuffle函数都是参数是单列表,
这个sklearn的包可以是多列表,如代码所示,有意思的是,对于多个参数,他们打乱的顺序是一样的,超赞。
3:转换值的时候遇到了astype(np.float32) ValueError: could not convert string to float,后来调试发现csv数据里面有一个空值,所以转不了数字,报错了。
4:min_samples_split =1 也报错了,改成2就行了。
5:模型里的rf_regressor.feature_importances_ ,里面都是参数的权重,哪些重要,不信,可以把csv的列顺序替换一下,看看结果是不是变化了。
6:以上代码参考了https://blog.csdn.net/weixin_42039090/article/details/80640890
R语言版本的实现:具体参考 https://blog.csdn.net/nieson2012/article/details/51279332
显然R语言的版本更加精简,也通俗易懂。
library(”randomForest”) data(iris) set.seed(100) ind=sample(2,nrow(iris),replace=TRUE,prob=c(0.8,0.2)) iris.rf=randomForest(Species~.,iris[ind==1,],ntree=50,nPerm=10,mtry=3,proximity=TRUE,importance=TRUE) print(iris.rf) iris.pred=predict( iris.rf,iris[ind==2,] ) table(observed=iris[ind==2,"Species"],predicted=iris.pred )
最后还是补充一点:参考 https://blog.csdn.net/qq_37423198/article/details/76922207
RF的主要优点有:
1) 训练可以高度并行化,对于大数据时代的大样本训练速度有优势。
2) 由于可以随机选择决策树节点划分特征,这样在样本特征维度很高的时候,仍然能高效的训练模型。
3) 在训练后,可以给出各个特征对于输出的重要性
4) 由于采用了随机采样,训练出的模型的方差小,泛化能力强。
5) 相对于Boosting系列的Adaboost和GBDT, RF实现比较简单。
6) 对部分特征缺失不敏感。
7) 对数据集的适应能力强:既能处理离散型数据,也能处理连续型数据,数据集无需规范化
8) 在创建随机森林的时候,对generlization error使用的是无偏估计
9) 训练过程中,能够检测到feature间的互相影响
RF的主要缺点有:
1) 在某些噪音比较大的样本集上,RF模型容易陷入过拟合。
2) 取值划分比较多的特征容易对RF的决策产生更大的影响,从而影响拟合的模型的效果。
最后,再来一段权重的直方图吧
import matplotlib.pyplot as plt
import seaborn as sns
color = sns.color_palette()
sns.set_style('darkgrid')
features_list = feature_names
feature_importance = rf_regressor.feature_importances_
sorted_idx = np.argsort(feature_importance)
plt.figure(figsize=(5, 7))
plt.barh(range(len(sorted_idx)), feature_importance[sorted_idx], align='center')
plt.yticks(range(len(sorted_idx)), features_list[sorted_idx])
plt.xlabel('Importance')
plt.title('Feature importances')
plt.draw()
plt.show()
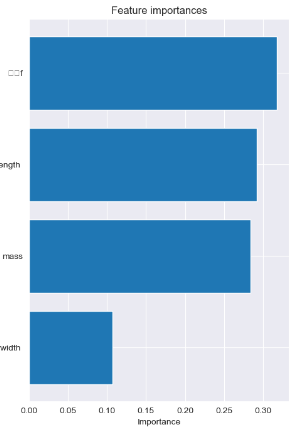
再再补充一点。。
这篇文章写得不错,https://www.cnblogs.com/pinard/p/6156009.html
对bagging和GBDT的区别那段,
GBDT的子采样是无放回采样,而Bagging的子采样是放回采样。
对于一个样本,它在某一次含m个样本的训练集的随机采样中,每次被采集到的概率是1m1m。不被采集到的概率为1−1m1−1m。如果m次采样都没有被采集中的概率是(1−1m)m(1−1m)m。当m→∞m→∞时,(1−1m)m→1e≃0.368(1−1m)m→1e≃0.368。也就是说,在bagging的每轮随机采样中,训练集中大约有36.8%的数据没有被采样集采集中。
对于这部分大约36.8%的没有被采样到的数据,我们常常称之为袋外数据(Out Of Bag, 简称OOB)。这些数据没有参与训练集模型的拟合,因此可以用来检测模型的泛化能力。
bagging对于弱学习器没有限制,这和Adaboost一样。但是最常用的一般也是决策树和神经网络。
bagging的集合策略也比较简单,对于分类问题,通常使用简单投票法,得到最多票数的类别或者类别之一为最终的模型输出。对于回归问题,通常使用简单平均法,对T个弱学习器得到的回归结果进行算术平均得到最终的模型输出。
由于Bagging算法每次都进行采样来训练模型,因此泛化能力很强,对于降低模型的方差很有作用。当然对于训练集的拟合程度就会差一些,也就是模型的偏倚会大一些。



